Letzte Aktualisierung: 30.09.2024
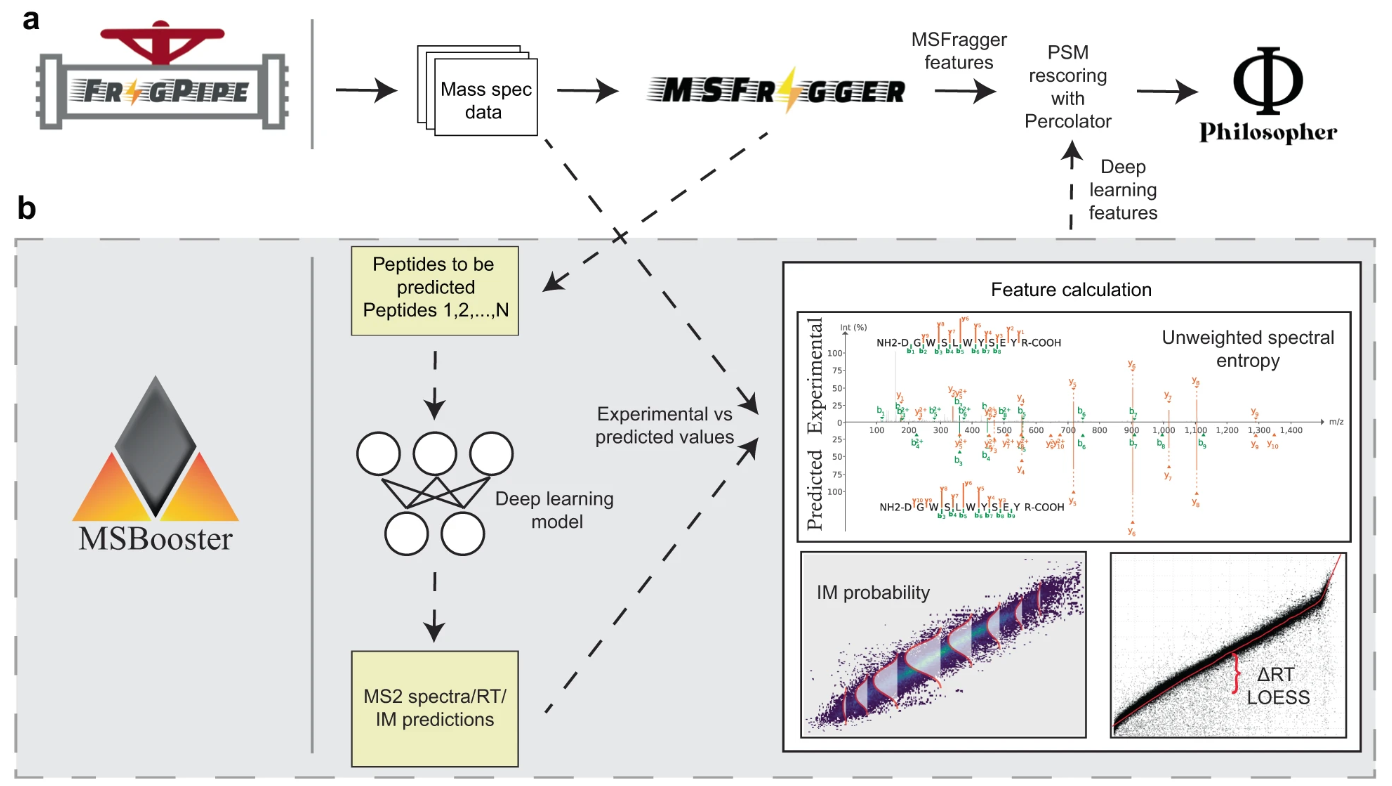
MSBooster ist ein Tool zum Einbeziehen von Spektralbibliotheksvorhersagen in die Peptid-Spektrum-Match-Bewertung (PSM) in Bottom-up-Tandem-Flüssigkeitschromatographie-Massenspektrometrie-Proteomikdaten. Es ist grob in 4 Schritte unterteilt:
Peptidextraktion aus PSMs in Suchergebnissen und Formatierung für die Eingabedateien von Prädiktoren für maschinelles/Deep Learning (ML/DL).
Aufrufen der Vorhersagemodelle und Speichern der Ausgabe
Feature-Berechnung
Hinzufügen neuer Funktionen zur Suchergebnisdatei
MSBooster ist mit vielen Arten von Datenbanksuchen kompatibel, einschließlich HLA-Immunpeptidomik, DDA und DIA sowie Einzelzell-Proteomik. Es ist in FragPipe integriert und in viele seiner Workflows integriert. MSBooster wurde unter Berücksichtigung anderer FragPipe-Tools entwickelt, beispielsweise FragPipe-PDV.
MSBooster ist für die Verarbeitung mehrerer Eingabedateiformate und -modelle ausgestattet:
| Ausgabe des Massenspektrometers |
|---|
| .mzML |
| .mgf |
| PSM-Datei |
|---|
| .Stift |
| .pepXML (in Bearbeitung) |
| Vorhersagemodell |
|---|
| DIA-NN |
| Koina-Modelle |
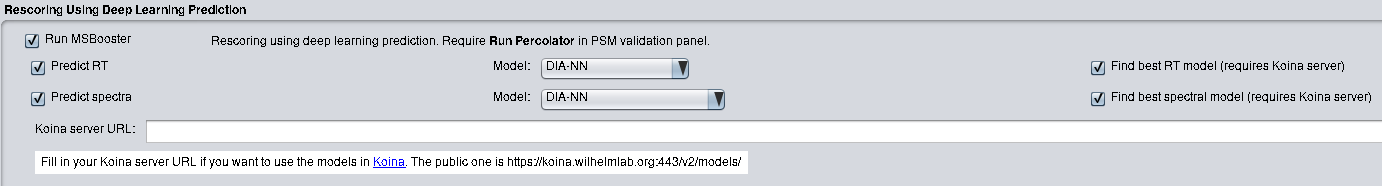
MSBooster kann auf Windows- und Linux-Systemen ausgeführt werden. Wenn Sie FragPipe verwenden, sind außer der Installation von FragPipe keine weiteren Installationsschritte erforderlich. MSBooster befindet sich auf der Registerkarte „Validierung“. Aktivieren Sie Retentionszeitfunktionen mit „Predict RT“ und MS/MS-Spektralfunktionen mit „Predict spectra“. Informationen zum Ausführen einer Analyse finden Sie in der FragPipe-Dokumentation.
Wenn Sie den eigenständigen MSBooster zur Ausführung in der Befehlszeile verwenden, laden Sie bitte die neueste JAR-Datei von Releases herunter. MSBooster erfordert außerdem DIA-NN für die MS/MS- und RT-Vorhersage. Bitte installieren Sie DIA-NN und notieren Sie sich den Pfad zur ausführbaren DIA-NN-Datei (z. B. DiaNN.exe für Windows, diann-1.8.1.8 für Linux).
Sie können MSBooster mit einem Befehl ähnlich dem folgenden ausführen:
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
Die Mindestparameter, die übergeben werden müssen, sind:
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
Sie können diese Parameter zwar einzeln übergeben, es ist jedoch einfacher, einen in jede Zeile der paramsList-Datei zu platzieren. Eine Vorlage finden Sie unter msbooster_params.txt.
Die folgenden Parameter dienen der allgemeinen Verwendung. Koina-spezifische Parameter finden Sie in der Koina-Dokumentation
paramsList (String) : Speicherort der Textdatei, die Parameter für diesen Lauf enthält
fragger (String) : Dateipfad der Datei fragger.params aus der MSFragger-Ausführung. MSBooster liest mehrere Parameter ein und passt darauf basierend interne Parameter an, z. B. Fragmentmassenfehlertoleranz und Massenoffsets
outputDirectory (String) : Wo die neuen Dateien ausgegeben werden sollen
editedPin (String) : MSBooster benennt die neue Datei basierend auf den bereitgestellten. Beispielsweise wird A.pin ein Gegenstück namens A_edited.pin haben. Um den Standardwert „bearbeitet“ zu ändern, geben Sie hier eine neue Zeichenfolge ein
renamePin (int) : ob eine neue Pin-Datei generiert oder die alte neu geschrieben werden soll. Der Standardwert ist hier 1, der nicht überschrieben wird. Wenn Sie diesen Wert auf 0 setzen, wird die alte PIN-Datei überschrieben
deletePreds (boolean) : Legt fest, ob die Dateien, in denen Modellvorhersagen gespeichert sind, nach Abschluss eines erfolgreichen Laufs gelöscht werden sollen. Standardmäßig auf „false“ eingestellt. Setzen Sie den Wert auf „true“, wenn Sie diese löschen möchten
loadingPercent (int) : Wie oft der Fortschritt von Aufgaben mithilfe eines Fortschrittsberichters gemeldet werden soll. Standardmäßig auf 10 eingestellt, was bedeutet, dass alle 10 % eine Aktualisierung gedruckt wird.
numThreads (int) : Anzahl der zu verwendenden Threads. Standardmäßig auf 0 eingestellt, wodurch alle verfügbaren Threads minus 1 verwendet werden
splitPredInputFile (int) : wird nur verwendet, wenn DIA-NN-Vorhersagen aufgrund eines Speichermangelfehlers (137) fehlschlagen. Standardmäßig auf 1 eingestellt, Sie können diesen Wert jedoch erhöhen, um anzugeben, in wie viele kleinere Dateien die DIA-NN-Eingabedatei aufgeteilt werden soll. Jede Datei wird dann nacheinander vorhergesagt, was die Speicherbelastung verringert
plotExtension (String) : welches Dateiformat Plots standardmäßig haben sollten. PNG, und PDF ist ebenfalls zulässig
features (String) : Liste der zu berechnenden Features. Groß- und Kleinschreibung beachten, komm-getrennt ohne Leerzeichen dazwischen. Der Standardwert ist „predRTrealUnits,unweightedSpectralEntropy,deltaRTLOESS“.
spectraPredFile (String) : Wenn Sie alte Spektralvorhersagen (z. B. von DIA-NN oder Koina) wiederverwenden, können Sie hier den Speicherort der Datei angeben
RTPredFile (String) : wie spectraPredFile, jedoch für RT-Vorhersagen
IMPredFile (String) : wie spectraPredFile, jedoch für IM-Vorhersagen
spectraModel (String) : welches Spektralvorhersagemodell verwendet werden soll
rtModel (String) : wie spectraModel, aber für RT
imModel (String) : wie spectraModel, aber für IM
useSpectra (boolean) : ob spektralvorhersagebasierte Funktionen verwendet werden sollen. Standardmäßig auf „true“ gesetzt
useRT (boolean) : ob RT-vorhersagebasierte Funktionen verwendet werden sollen. Standardmäßig auf „true“ gesetzt
useIM (boolean) : Gibt an, ob IM-Vorhersage-basierte Funktionen verwendet werden sollen. Standardmäßig auf „false“ gesetzt
ppmTolerance (float) : Fragmentfehler-PPM-Toleranz (Standard 20 ppm)
matchWithDaltons (boolean) : ob vorhergesagte und beobachtete Fragmente in Daltons abgeglichen werden sollen (Standardwert falsch)
DaTolerance (float) : Wie viele Daltons um den vorhergesagten Peak herum soll nach einem experimentellen Peak gesucht werden (Standard 0,05)
useTopFragments (boolean) : ob die spektrale Vorhersage nach den N Peaks mit der höchsten Intensität gefiltert werden soll (Standard: true)
topFragments (int) : bis zu wie viele vorhergesagte Fragmente für die Feature-Berechnung verwendet werden sollen (Standard 20). Wird nur angewendet, wenn useTopFragments wahr ist
removeRankPeaks (boolean) : Standardmäßig auf „true“ gesetzt, wodurch Fragmente aus den experimentellen Spektren herausgefiltert werden, sobald sie übereinstimmen. Bei „falsch“ können experimentelle Fragmente von mehreren PSMs aus demselben Scan abgeglichen werden
useBasePeak (boolean) : ob eine Untergrenze auf MS2-Vorhersagen angewendet werden soll, um nur Fragmente mit höherer Intensität zu verwenden (Standard: true)
percentBasePeak (float) : Prozentsatz, bei dem ein Fragment mit einer Intensität von einigen Prozent der Basispeakintensität in die Ähnlichkeitsberechnung einbezogen wird. Wird nur angewendet, wenn useBasePeak wahr ist (Standard 1).
loessEscoreCutoff (float) : Erwartungswert-Grenzwert, der für den ersten Durchgang beim Sammeln von PSMs für die RT/IM-Kalibrierung verwendet wird. Der Standardwert ist 10^-3,5 oder ungefähr 0,000316
rtLoessRegressionSize (int) : maximale Anzahl von PSMs, die für die RT LOESS-Kalibrierung verwendet werden (Standard 5000)
imLoessRegressionSize (int) : wie rtLoessRegressionSize, aber für IM (Standard 1000)
minLoessRegressionSize (int) : Mindestanzahl von PSMs, die für den Versuch der LOESS RT/IM-Kalibrierung erforderlich sind (Standard 100). Wenn weniger als diese Anzahl an PSMs verfügbar sind, wird stattdessen die lineare Regression verwendet
minLinearRegressionSize (int) : Mindestanzahl von PSMs, die für den Versuch der linearen Regressions-RT/IM-Kalibrierung erforderlich sind (Standard 10). Wenn weniger als diese Anzahl an PSMs verfügbar sind, wird keine Kalibrierung versucht
loessBandwidth (String) : Liste der Bandbreiten, die für die RT/IM-LOESS-Kalibrierung getestet werden sollen (Standard 0,01, 0,05, 0,1, 0,2). Dies muss durch Kommas getrennt werden und darf keine Leerzeichen dazwischen enthalten
regressionSplits (int) : Anzahl der Kreuzvalidierungen, die für die RT/IM-LOESS-Kalibrierung verwendet werden (Standard 5)
massesForLoessCalibration (String) : Massen für Massenverschiebungen, die an ihre eigenen Kalibrierungskurven angepasst werden sollen. Die Liste ist durch Kommas getrennt und darf keine Leerzeichen dazwischen enthalten. Die Massen sollten mit der gleichen Stellenanzahl wie in der PIN-Datei geschrieben werden
loessScatterOpacity (float) : Opazität von Streudiagrammen in LOESS-Kalibrierungszahlen, von 0 bis 1 (Standard 0,35)
.pin-Datei mit neuen Funktionen. Standardmäßig werden neue Pin-Dateien mit der Endung „_edited.pin“ erstellt. Die verwendeten Standardfunktionen sind „unweighted_spectral_entropy“, „delta_RT_loess“ und „pred_RT_real_units“. Wenn Ionenmobilitätsfunktionen aktiviert sind, werden auch „delta_IM_loess“ und „ion_mobility“ einbezogen
spectraRT.tsv und spectraRT_full.tsv: Eingabedateien für das DIA-NN-Vorhersagemodell
spectraRT.predicted.bin: eine Binärdatei mit Vorhersagen von DIA-NN, die von MSBooster zur Feature-Berechnung verwendet werden soll. Bei Verwendung von FragPipe-PDV werden diese Dateien verwendet, um Spiegeldiagramme experimenteller und vorhergesagter Spektren zu erstellen
MSBooster erstellt mehrere Diagramme, mit denen Sie weiter untersuchen können, wie Ihre Daten im Vergleich zu Modellvorhersagen abschneiden.
Ordner „MSBooster_plots“:
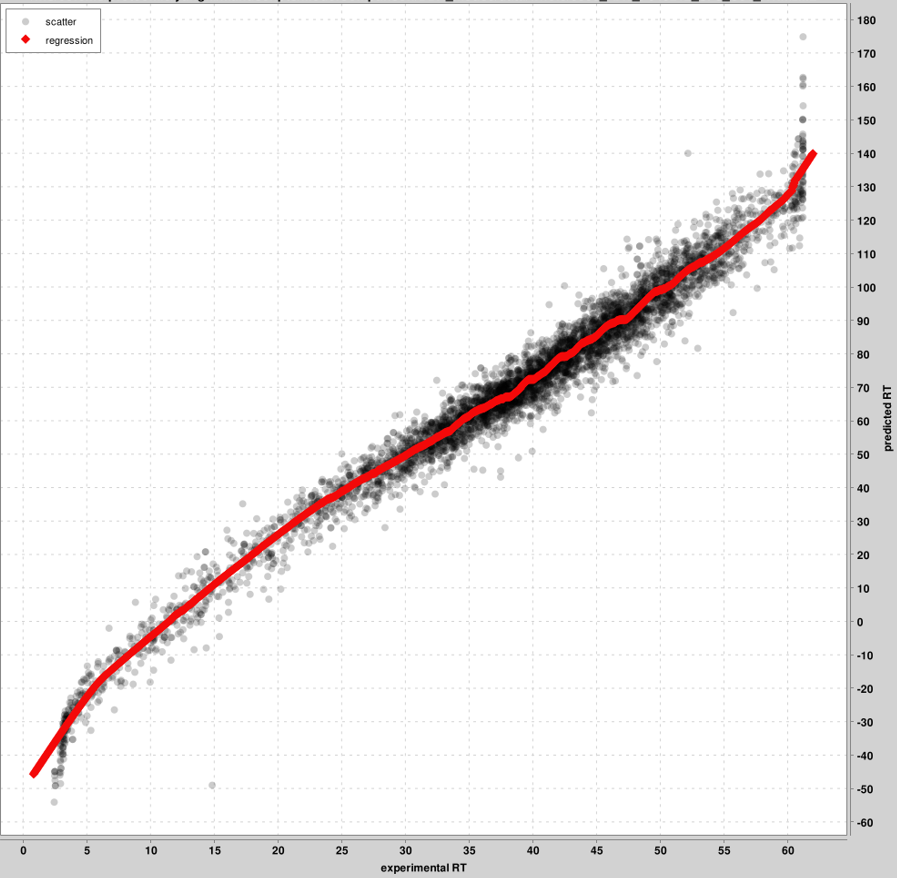
RT_Kalibrierungskurven: Bis zu den besten 5000 PSMs werden für die Kalibrierung zwischen der experimentellen und der vorhergesagten RT-Skala verwendet. Diese Top-PSMs werden in der Grafik dargestellt, nicht alle PSMs. Pro Pin-Datei wird ein Diagramm erstellt
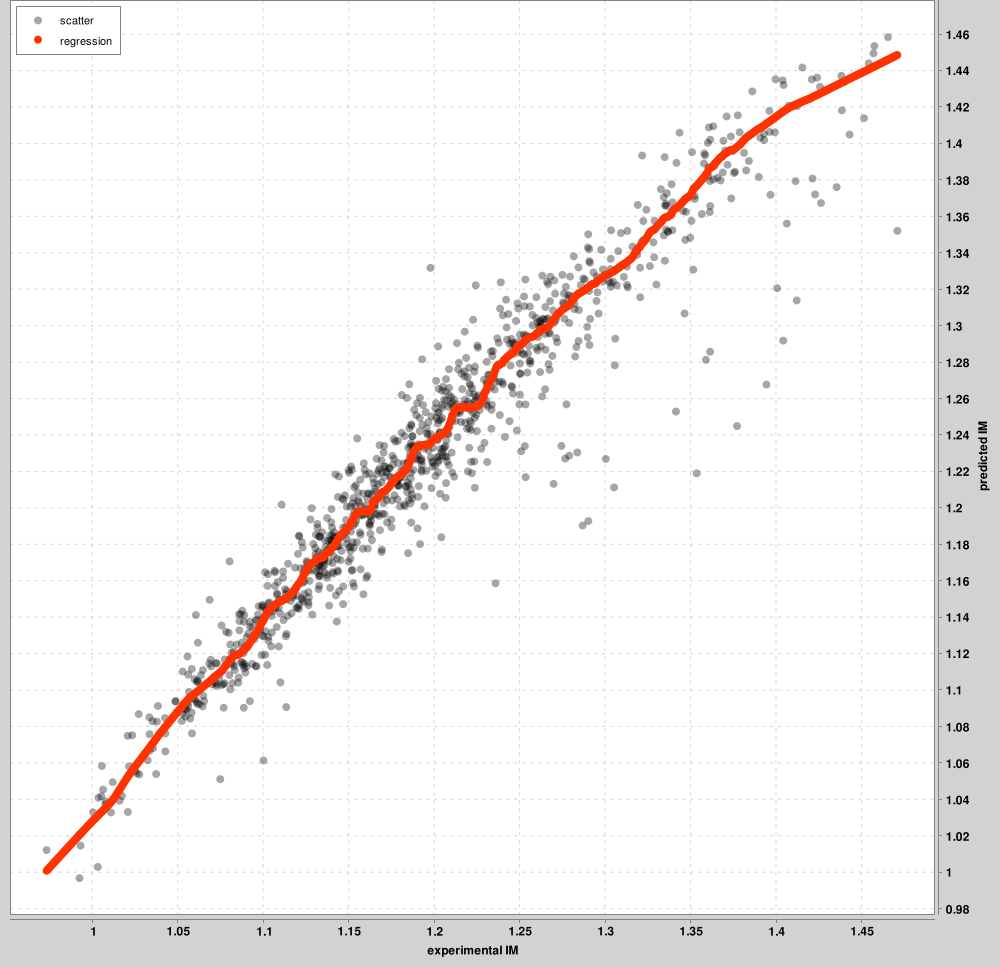
IM_Kalibrierungskurven: Bis zu den besten 1000 PSMs werden für die Kalibrierung zwischen den experimentellen und vorhergesagten IM-Skalen verwendet. Diese Top-PSMs werden in der Grafik dargestellt, nicht alle PSMs. Für jeden Ladezustand wird eine eigene Kurve gelernt. Die folgende Abbildung ist ein Beispiel für Ladung-2-Vorläufer
score_histograms: überlagerte Histogramme aller Ziel- und Täuschungs-PSMs für jede Pin-Datei. Einige Merkmale werden hier im Logarithmus dargestellt, um die bimodale Verteilung von echten und falschen Positiven besser zu veranschaulichen. In den Pin-Dateien wird jedoch der ursprüngliche Wert verwendet, nicht die im Logarithmus skalierte Version. Hier sind Histogramme für die ungewichteten spektralen Entropie- und Delta-RT-Werte dargestellt, es werden jedoch für alle Merkmale ähnliche Histogramme erstellt
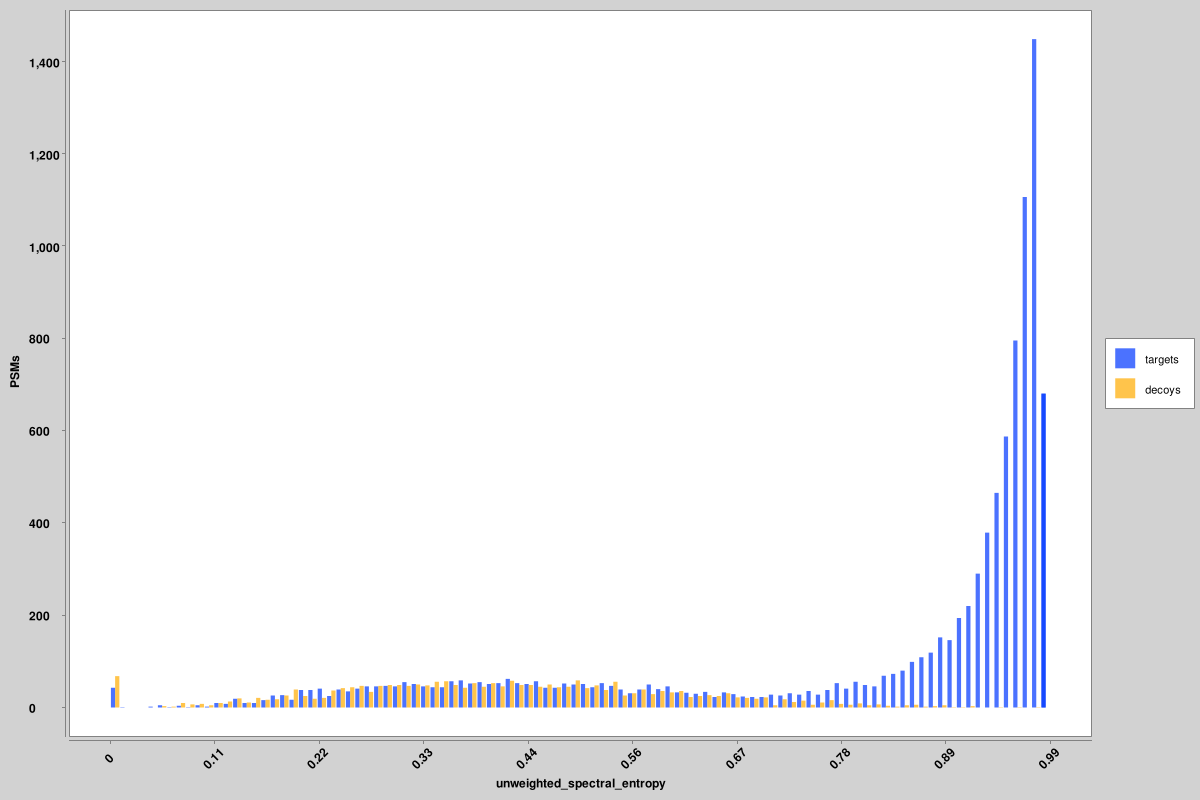
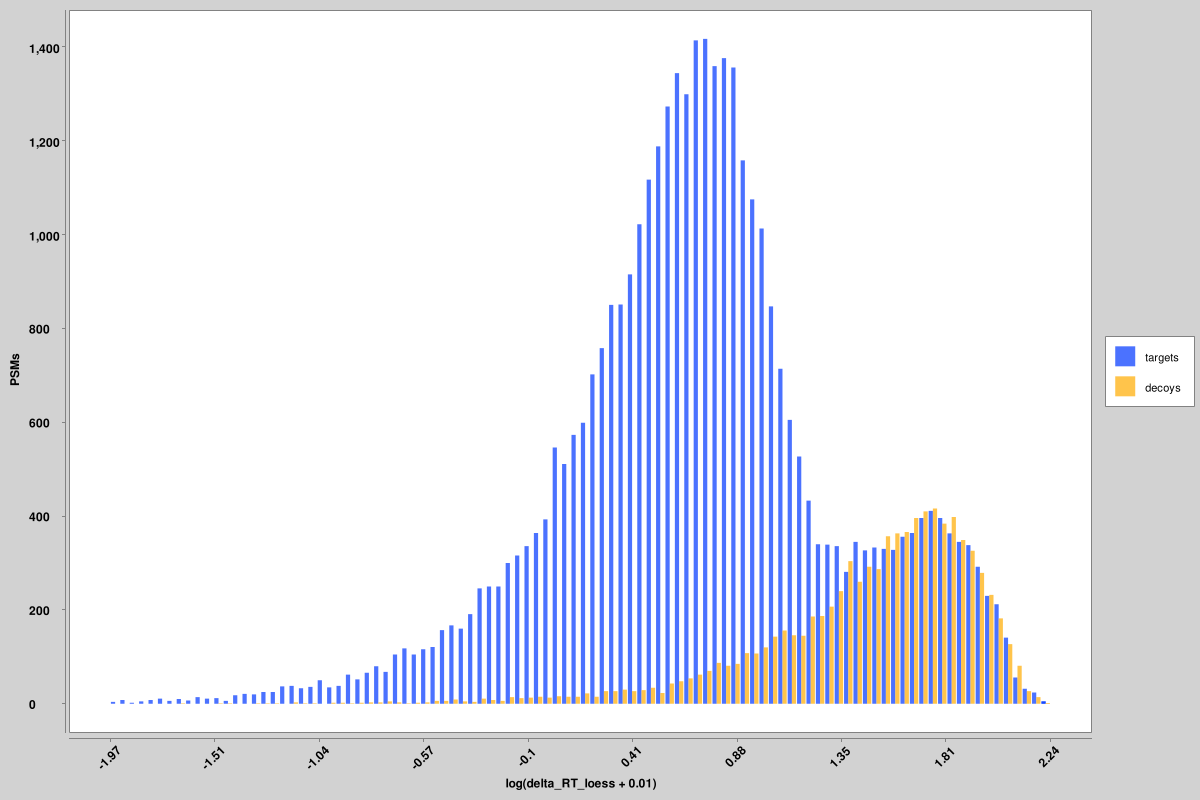
Verwenden Sie Peptidvorhersagemodelle von Koina für die Generierung von MSBooster-Funktionen: https://fragpipe.nesvilab.org/docs/tutorial_koina.html
Einlesen von Vorhersagen aus beliebigen Modellen über MGF-Dateien
Dokumentation aller zulässigen Funktionen und deren Qualitätskontrolle mit grafischer Ausgabe
Bitte geben Sie Folgendes an, wenn Sie MSBooster verwenden: https://www.nature.com/articles/s41467-023-40129-9