Dies ist das offizielle Repository für „One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompts“
Es handelt sich um ein wissensgestütztes universelles Segmentierungsmodell, das auf einer beispiellosen Datensammlung (72 öffentliche medizinische 3D-Segmentierungsdatensätze) basiert und 497 Klassen aus 3 verschiedenen Modalitäten (MR, CT, PET) und 8 Regionen des menschlichen Körpers segmentieren kann, gesteuert durch Text (anatomisch). Terminologie).
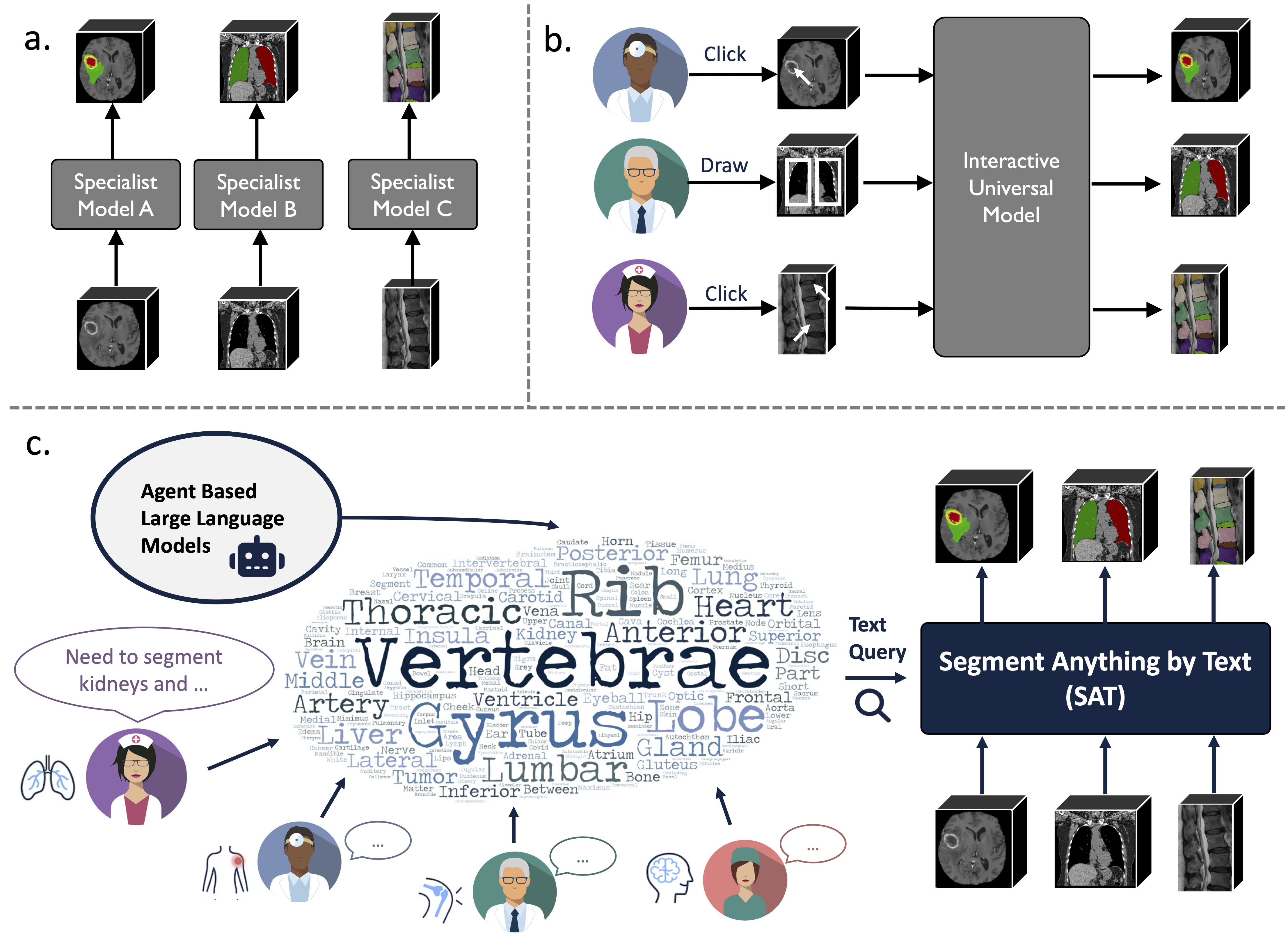
Es kann leistungsfähiger und effizienter sein als das Training und der Einsatz einer Reihe von Spezialmodellen. Weitere Informationen finden Sie auf unserer Website oder in unserer Zeitung.
2024.08 ? Basierend auf SAT- und großen Sprachmodellen erstellen wir einen umfassenden, groß angelegten und regionalgesteuerten 3D-Thorax-CT-Interpretationsdatensatz. Es enthält eine Segmentierung auf Organebene für 196 Kategorien und Berichte mit mehreren Granularitäten, in denen jeder Satz auf der entsprechenden Segmentierung basiert. Überprüfen Sie es auf Huggingface.
2024.06 ? Wir haben den Code zum Erstellen von SAT-DS veröffentlicht, einer Sammlung von 72 öffentlichen Segmentierungsdatensätzen, die über 22.000 3D-Bilder, 302.000 Segmentierungsmasken und 497 Klassen aus 3 verschiedenen Modalitäten (MRT, CT, PET) und 8 Regionen des menschlichen Körpers enthält Wir bauen SAT. Wir bieten auch Shortcut-Download-Links für 42/72-Datensätze an, die von uns für Sie vorverarbeitet und gepackt werden und nach dem Herunterladen und Extrahieren sofort einsatzbereit sind. Weitere Informationen finden Sie in diesem Repo.
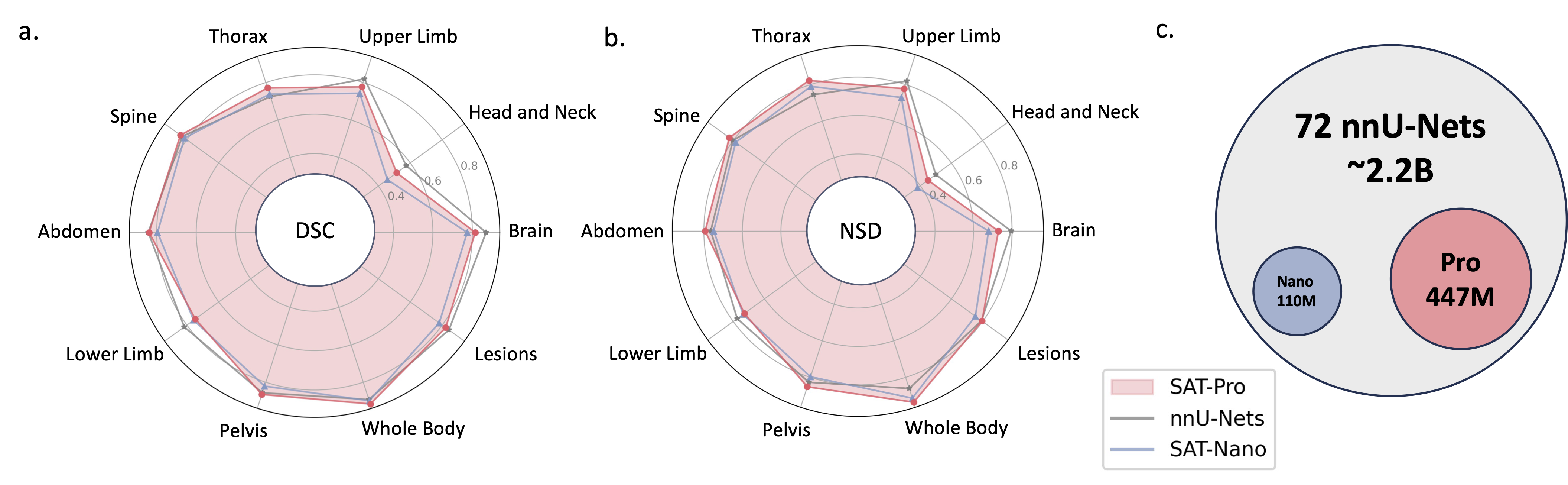
2024.05 ? Wir trainieren eine neue Version von SAT mit größerer Modellgröße ( SAT-Pro ) und mehr Datensätzen ( 72 ), die jetzt 497 Klassen unterstützt! Wir erneuern auch SAT-Nano und veröffentlichen einige Varianten von SAT-Nano, die auf verschiedenen visuellen Backbones (U-Mamba und SwinUNETR) und Text-Encodern (MedCPT und BERT-Base) basieren. Weitere Einzelheiten zu diesem Update finden Sie in unserem neuen Dokument.
Die Implementierung von U-Net basiert auf einer angepassten Version dynamischer Netzwerkarchitekturen, um es zu installieren:
cd model
pip install -e dynamic-network-architectures-main
Einige weitere wichtige Anforderungen:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
Sie müssen auch mamba_ssm installieren, wenn Sie die U-Mamba-Variante von SAT-Nano möchten
S1. Erstellen Sie die Umgebung gemäß requirements.txt .
S2. Laden Sie den Checkpoint von SAT und Text Encoder von Huggingface herunter.
S3. Bereiten Sie die Daten in einer JSONL-Datei vor. Sehen Sie sich die Demo in data/inference_demo/demo.jsonl an.
Für jede zu segmentierende Probe werden image (Pfad zum Bild), labe (Name des Segmentierungsziels), dataset (zu welchem Datensatz die Probe gehört) und modality (CT, MRT oder Haustier) benötigt. Die von SAT unterstützten Modalitäten und Klassen finden Sie in Tabelle 12 des Dokuments.
orientation_code (Ausrichtung) ist standardmäßig RAS , was für die meisten Bilder in der axialen Ebene geeignet ist. Für Bilder in der Sagittalebene (z. B. Wirbelsäulenuntersuchung) stellen Sie dies auf ASR ein. Das Eingabebild sollte die Form H,W,D haben. Unser Datenverarbeitungscode normalisiert das Eingabebild in Bezug auf Ausrichtung, Intensität, Abstand usw. Zwei erfolgreich verarbeitete Bilder finden Sie in demoprocessed_data . Stellen Sie sicher, dass die Normalisierung korrekt durchgeführt wird, um die Leistung von SAT zu gewährleisten.
S4. Inferenz mit SAT-Pro starten?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d ist die Stapelgröße der Eingabebild-Patches und muss basierend auf dem GPU-Speicher angepasst werden (siehe Tabelle unten); Es wird empfohlen, --max_queries größer als die Klassen im Inferenzdatensatz festzulegen, es sei denn, Ihr GPU-Speicher ist sehr begrenzt;
| Modell | Batchsize_3d | GPU-Speicher |
|---|---|---|
| SAT-Pro | 1 | ~ 34 GB |
| SAT-Pro | 2 | ~ 62 GB |
| SAT-Nano | 1 | ~ 24 GB |
| SAT-Nano | 2 | ~ 36 GB |
S5. Überprüfen Sie --rcd_dir auf Ausgaben. Die Ergebnisse werden nach Datensätzen organisiert. Für jeden Fall werden das Eingabebild, das aggregierte Segmentierungsergebnis und ein Ordner mit Segmentierungen jeder Klasse gefunden. Alle Ausgaben werden als Nifiti-Dateien gespeichert. Diese können Sie mit dem ITK-SNAP visualisieren.
Wenn Sie SAT-Nano verwenden möchten, das auf 72 Datensätzen trainiert wurde, ändern Sie einfach --vision_backbone in „UNET“ und ändern Sie den --checkpoint und --text_encoder_checkpoint entsprechend.
Für andere SAT-Nano-Varianten (trainiert anhand von 49 Datensätzen):
UNET-Ours: set --vision_backbone 'UNET' und --text_encoder 'ours' ;
UNET-CPT: set --vision_backbone 'UNET' und --text_encoder 'medcpt' ;
UNET-BB: set --vision_backbone 'UNET' und --text_encoder 'basebert' ;
UMamba-CPT: set --vision_backbone 'UMamba' und --text_encoder 'medcpt' ;
SwinUNETR-CPT: set --vision_backbone 'SwinUNETR' und --text_encoder 'medcpt' ;
Einige Vorbereitungen vor Beginn des Trainings:
sh/ um den Trainingsprozess zu starten. Nehmen Sie zum Beispiel SAT-Pro: sbatch sh/train_sat_pro.sh
Dies erfordert auch die Erstellung von Testdaten im Anschluss an dieses Repo. Sie können auf das Slurm-Skript sh/evaluate_sat_pro.sh zurückgreifen, um den Bewertungsprozess zu starten:
sbatch sh/evaluate_sat_pro.sh
Wenn Sie diesen Code für Ihre Forschung oder Ihr Projekt verwenden, geben Sie bitte Folgendes an:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}