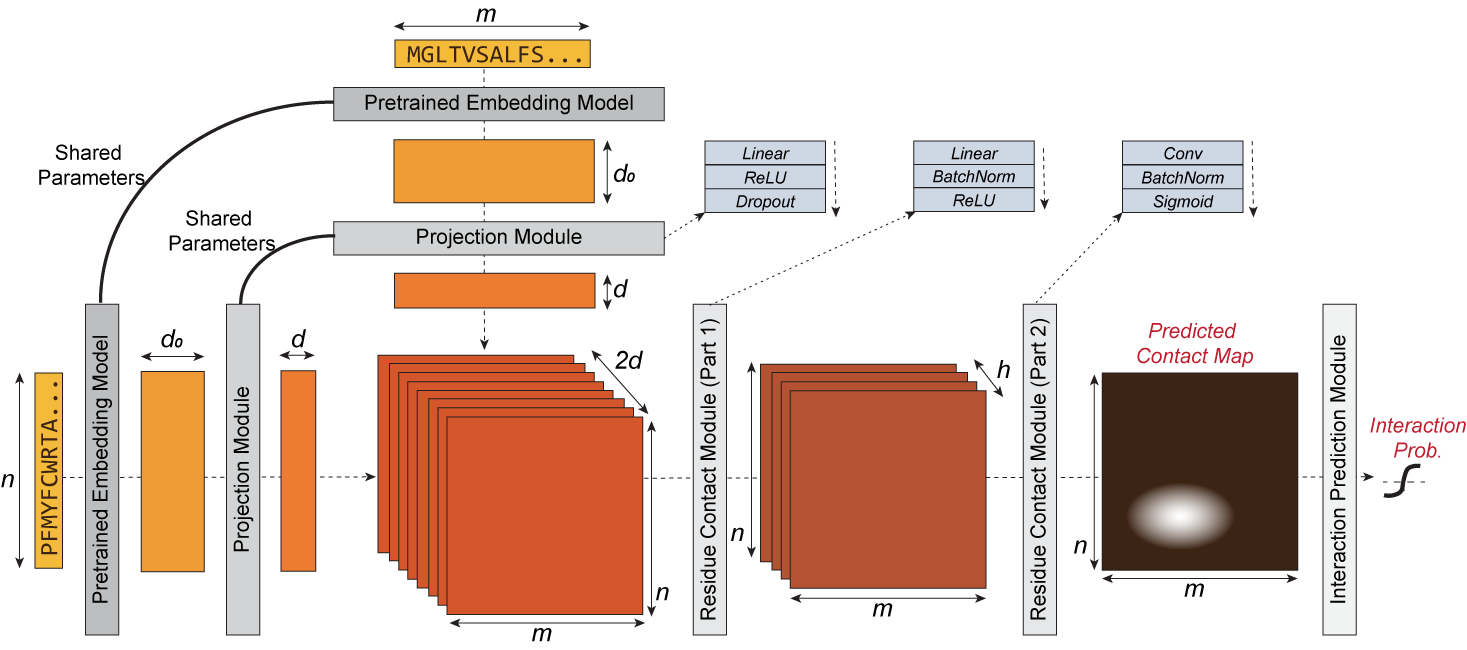
D-SCRIPT ist eine Deep-Learning-Methode zur Vorhersage einer physikalischen Interaktion zwischen zwei Proteinen anhand ihrer Sequenzen. Es lässt sich gut auf neue Arten verallgemeinern und ist robust gegenüber Einschränkungen in der Trainingsdatengröße. Sein Design spiegelt die Intuition wider, dass für die physikalische Interaktion zweier Proteine eine Teilmenge der Aminosäuren jedes Proteins mit dem anderen in Kontakt stehen muss. Die Zwischenstufen von D-SCRIPT setzen diese Intuition direkt um, wobei die vorletzte Stufe in D-SCRIPT eine grobe Schätzung der Interprotein-Kontaktkarte des Proteindimers ist. Dieses strukturell motivierte Design verbessert die Interpretierbarkeit der Ergebnisse und verbessert die Generalisierbarkeit über Arten hinweg, da die Struktur evolutionär stärker konserviert ist als die Sequenz.
D-SCRIPT wird in der Arbeit „Sequence-based Prediction of Protein-Protein Interactions: A Structure-Aware Interpretable Deep Learning Model“ von Sam Sledzieski, Rohit Singh, Lenore Cowen und Bonnie Berger beschrieben.
Homepage
Dokumentation