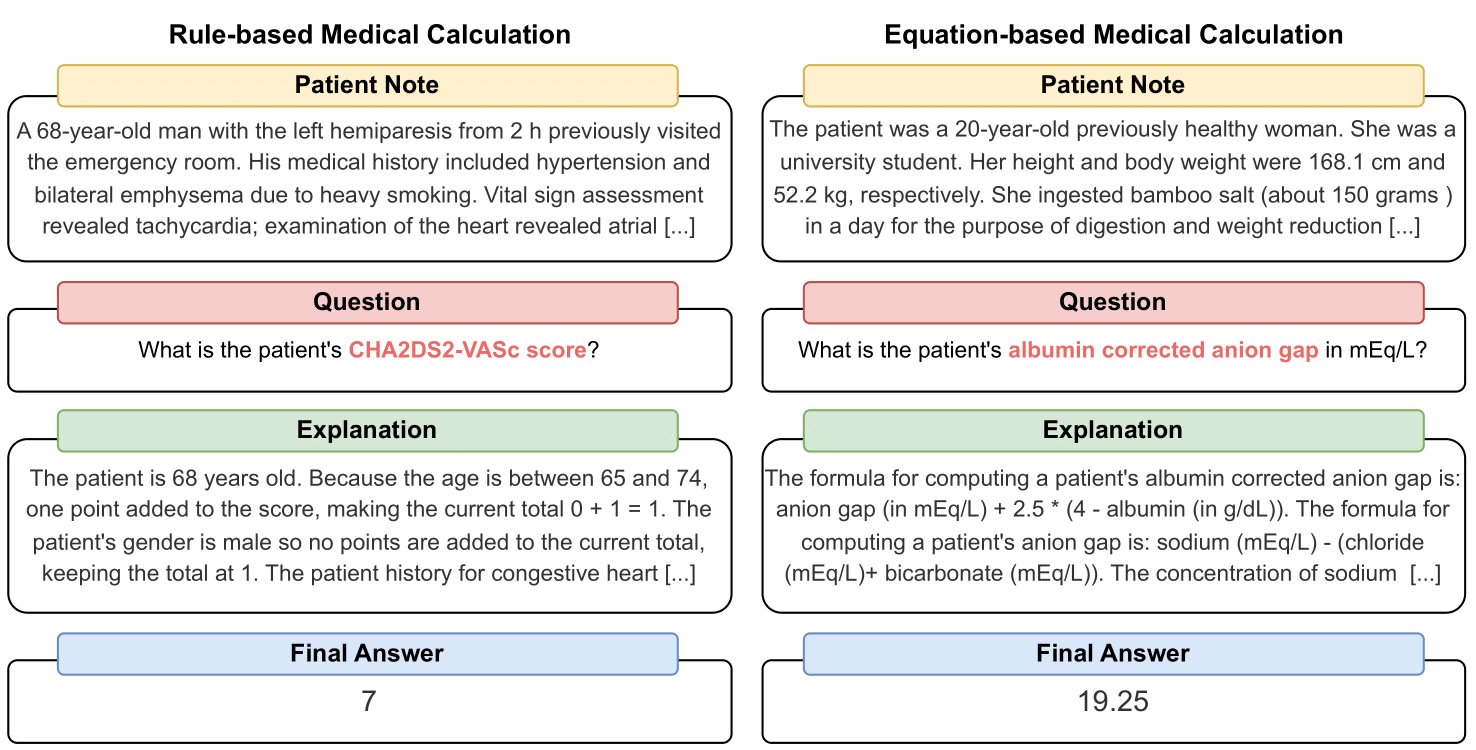
MedCalc-Bench ist der erste medizinische Berechnungsdatensatz, der zum Benchmarking der Fähigkeit von LLMs verwendet wird, als klinische Rechner zu dienen. Jede Instanz im Datensatz besteht aus einer Patientennotiz, einer Frage zur Berechnung eines bestimmten klinischen Werts, einem endgültigen Antwortwert und einer Schritt-für-Schritt-Lösung, die erklärt, wie die endgültige Antwort erhalten wurde. Unser Datensatz umfasst 55 verschiedene Berechnungsaufgaben, bei denen es sich entweder um regelbasierte Berechnungen oder um gleichungsbasierte Berechnungen handelt. Dieser Datensatz enthält einen Trainingsdatensatz mit 10.053 Instanzen und einen Testdatensatz mit 1.047 Instanzen.
Insgesamt hoffen wir, dass unser Datensatz und Benchmark als Aufruf zur Verbesserung der rechnerischen Denkfähigkeiten von LLMs im medizinischen Umfeld dienen.
Unser Vorabdruck ist verfügbar unter: https://arxiv.org/abs/2406.12036.
Um die CSV-Datei für den MedCalc-Bench-Bewertungsdatensatz herunterzuladen, laden Sie bitte die Datei test_data.csv im dataset dieses Repositorys herunter. Sie können den Testsatz-Split auch von HuggingFace unter https://huggingface.co/datasets/ncbi/MedCalc-Bench herunterladen.
Zusätzlich zu den 1.047 Evaluierungsinstanzen stellen wir auch einen Trainingsdatensatz von 10.053 Instanzen zur Verfügung, der zur Feinabstimmung von Open-Source-LLMs verwendet werden kann (siehe Abschnitt C des Anhangs). Die Trainingsdaten finden Sie in der Datei dataset/train_data.csv.zip und können entpackt werden, um train_data.csv zu erhalten. Dieser Trainingsdatensatz ist auch im Train-Split des HuggingFace-Links zu finden.
Jede Instanz im Datensatz enthält die folgenden Informationen:
Um alle für dieses Projekt benötigten Pakete zu installieren, führen Sie bitte den folgenden Befehl aus: conda env create -f environment.yml . Dieser Befehl erstellt die Conda-Umgebung medcalc-bench . Zum Ausführen von OpenAI-Modellen müssen Sie Ihren OpenAI-Schlüssel in dieser Conda-Umgebung bereitstellen. Sie können dies tun, indem Sie den folgenden Befehl in der medcalc-bench -Umgebung ausführen: export OPENAI_API_KEY = YOUR_API_KEY , wobei YOUR_API_KEY Ihr OpenAI-API-Schlüssel ist. Sie müssen in dieser Umgebung auch Ihr HuggingFace-Token bereitstellen, indem Sie den folgenden Befehl ausführen: export HUGGINGFACE_TOKEN=your_hugging_face_token , wobei your_hugging_face_token Ihr Huggingface-Token ist.
Um die Tabelle 2 aus der Arbeit zu reproduzieren, cd zunächst in den evaluation . Führen Sie dann bitte den folgenden Befehl aus: python run.py --model <model_name> and --prompt <prompt_style> .
Die Optionen für --model sind unten:
Die Optionen für --prompt sind unten:
Daraus erhalten Sie eine JSONL-Datei, die den Status jeder Frage ausgibt: Beim Ausführen von run.py werden die Ergebnisse in einer Datei namens <model>_<prompt>.jsonl gespeichert. Diese Datei befindet sich im outputs .
Jeder Instanz im JSONL sind die folgenden Metadaten zugeordnet:
{
"Row Number": Row index of the item,
"Calculator Name": Name of calculation task,
"Calculator ID": ID of the calculator,
"Category": type of calculation (risk, severity, diagnosis for rule-based calculators and lab, risk, physical, date, dosage for equation-based calculators),
"Note ID": ID of the note taken directly from MedCalc-Bench,
"Patient Note": Paragraph which is the patient note taken directly from MedCalc-Bench,
"Question": Question asking for a specific medical value to be computed,
"LLM Answer": Final Answer Value from LLM,
"LLM Explanation": Step-by-Step explanation by LLM,
"Ground Truth Answer": Ground truth answer value,
"Ground Truth Explanation": Step-by-step ground truth explanation,
"Result": "Correct" or "Incorrect"
}
Darüber hinaus stellen wir die mittlere Genauigkeit und den Prozentsatz der Standardabweichung für jede Unterkategorie in einem JSON mit dem Titel results_<model>_<prompt_style>.json bereit. Die kumulative Genauigkeit und Standardabweichung aller 1.047 Instanzen finden Sie unter „Gesamt“-Schlüssel des JSON. Diese Datei befindet sich im results .
Zusätzlich zu den Ergebnissen für Tabelle 2 im Hauptpapier haben wir LLMs auch aufgefordert, Code zu schreiben, um Arithmetik durchzuführen, anstatt dass das LLM dies selbst tun muss. Die Ergebnisse hierfür finden Sie in Anhang D. Aufgrund der begrenzten Rechenleistung haben wir die Ergebnisse nur für GPT-3.5 und GPT-4 ausgeführt. Um die Eingabeaufforderungen zu untersuchen und mit dieser Einstellung auszuführen, überprüfen Sie bitte die Datei generate_code_prompt.py im evaluation .
Um diesen Code auszuführen, wechseln Sie einfach cd in den Ordner evaluations und führen Sie Folgendes aus: python generate_code_prompt.py --gpt <gpt_model> . Die Optionen für <gpt_model> sind entweder 4 für die Ausführung von GPT-4 oder 35 für die Ausführung von GPT-3.5-turbo-16k. Die Ergebnisse werden dann in einer JSONL-Datei mit dem Namen code_exec_{model_name}.jsonl im outputs gespeichert. Beachten Sie, dass in diesem Fall model_name gpt_4 lautet, wenn Sie sich für die Ausführung mit GPT-4 entschieden haben. Andernfalls lautet model_name gpt_35_16k , wenn Sie die Ausführung mit GPT-3.5-turbo ausgewählt haben.
Die Metadaten für jede Instanz in der JSONL-Datei für die Code-Interprepter-Ergebnisse sind dieselben Instanzinformationen wie im obigen Abschnitt. Der einzige Unterschied besteht darin, dass wir den LLM-Chat-Verlauf zwischen dem Benutzer und dem Assistenten speichern und einen „LLM-Chat-Verlauf“-Schlüssel anstelle des „LLM-Erklärungs“-Schlüssels haben. Darüber hinaus werden die Unterkategorie und die Gesamtgenauigkeit in einer JSON-Datei mit dem Namen results_<model_name>_code_augmented.json gespeichert. Dieses JSON befindet sich im results .
Diese Forschung wurde vom NIH Intramural Research Program der National Library of Medicine unterstützt. Darüber hinaus wurden die Beiträge von Soren Dunn mithilfe der Delta Advanced Computing- und Datenressource erstellt, die von der National Science Foundation (Auszeichnung OAC Tel.:2005572) und dem Bundesstaat Illinois unterstützt wird. Delta ist eine gemeinsame Initiative der University of Illinois Urbana-Champaign (UIUC) und ihres National Center for Supercomputing Applications (NCSA).
Für die Kuratierung der Patientennotizen in MedCalc-Bench verwenden wir nur öffentlich verfügbare Patientennotizen aus veröffentlichten Fallberichtsartikeln in PubMed Central und von Ärzten erstellte anonyme Patientenvignetten. Daher werden in dieser Studie keine identifizierbaren persönlichen Gesundheitsinformationen offengelegt. Während MedCalc-Bench darauf ausgelegt ist, die medizinischen Berechnungsmöglichkeiten von LLMs zu bewerten, ist zu beachten, dass der Datensatz nicht für den direkten diagnostischen Gebrauch oder die medizinische Entscheidungsfindung ohne Überprüfung und Aufsicht durch einen klinischen Fachmann gedacht ist. Einzelpersonen sollten ihr Gesundheitsverhalten nicht allein aufgrund unserer Studie ändern.
Wie in Abschnitt 1 beschrieben, werden medizinische Taschenrechner häufig im klinischen Umfeld verwendet. Angesichts des schnell wachsenden Interesses an der Verwendung von LLMs für domänenspezifische Anwendungen könnten Ärzte im Gesundheitswesen Chatbots wie ChatGPT direkt dazu auffordern, medizinische Berechnungsaufgaben durchzuführen. Allerdings sind die Fähigkeiten von LLMs bei diesen Aufgaben derzeit nicht bekannt. Da im Gesundheitswesen viel auf dem Spiel steht und falsche medizinische Berechnungen schwerwiegende Folgen haben können, darunter Fehldiagnosen, unangemessene Behandlungspläne und potenzielle Schäden für Patienten, ist es von entscheidender Bedeutung, die Leistung von LLMs bei medizinischen Berechnungen gründlich zu bewerten. Überraschenderweise zeigen die Auswertungsergebnisse unseres MedCalc-Bench-Datensatzes, dass alle untersuchten LLMs bei den medizinischen Berechnungsaufgaben Schwierigkeiten haben. Das leistungsstärkste Modell GPT-4 erreicht mit One-Shot-Lernen und Gedankenkettenaufforderung nur eine Genauigkeit von 50 %. Unsere Studie zeigt daher, dass aktuelle LLMs noch nicht für medizinische Berechnungen geeignet sind. Es ist zu beachten, dass hohe Ergebnisse bei MedCalc-Bench zwar keine Garantie für herausragende Leistungen bei medizinischen Berechnungsaufgaben sind, ein Nichtbestehen in diesem Datensatz jedoch darauf hinweist, dass die Modelle für solche Zwecke überhaupt nicht in Betracht gezogen werden dürfen. Mit anderen Worten: Wir glauben, dass das Bestehen des MedCalc-Bench eine notwendige (aber nicht hinreichende) Bedingung für die Verwendung eines Modells für medizinische Berechnungen sein sollte
Bei Änderungen an diesem Datensatz (z. B. Hinzufügen neuer Notizen oder Taschenrechner) aktualisieren wir die README-Anweisungen test_set.csv und train_set.csv. Ältere Versionen dieser Datensätze werden wir weiterhin in einem archive/ Ordner aufbewahren. Wir werden auch die Zug- und Testsets für HuggingFace aktualisieren.
Dieses Tool zeigt die Ergebnisse der Forschung, die in der Computational Biology Branch, NCBI/NLM, durchgeführt wurde. Die auf dieser Website bereitgestellten Informationen sind nicht für den direkten diagnostischen Gebrauch oder die medizinische Entscheidungsfindung ohne Prüfung und Aufsicht durch einen klinischen Fachmann bestimmt. Einzelpersonen sollten ihr Gesundheitsverhalten nicht allein aufgrund der auf dieser Website bereitgestellten Informationen ändern. NIH überprüft nicht unabhängig die Gültigkeit oder Nützlichkeit der von diesem Tool erzeugten Informationen. Wenn Sie Fragen zu den auf dieser Website bereitgestellten Informationen haben, wenden Sie sich bitte an einen Arzt. Weitere Informationen zur Haftungsausschlussrichtlinie von NCBI finden Sie hier.
Je nach Rechner besteht unser Datensatz aus Notizen, die entweder aus in Python implementierten vorlagenbasierten Funktionen entworfen, von Ärzten handgeschrieben wurden oder aus unserem Datensatz Open-Patients stammen.
Open-Patients ist ein aggregierter Datensatz mit 180.000 Patientennotizen, die aus drei verschiedenen Quellen stammen. Wir haben die Berechtigung, den Datensatz aus allen drei Quellen zu verwenden. Die erste Quelle sind die USMLE-Fragen von MedQA, die unter der MIT-Lizenz veröffentlicht werden. Die zweite Quelle unseres Datensatzes sind Trec Clinical Decision Support und Trec Clinical Trial, die zur Weiterverbreitung zur Verfügung stehen, da es sich bei beiden um staatliche Datensätze handelt, die der Öffentlichkeit zugänglich gemacht werden. Schließlich wird PMC-Patients unter der CC-BY-SA 4.0-Lizenz veröffentlicht und wir haben daher die Erlaubnis, PMC-Patients in Open-Patients und MedCalc-Bench zu integrieren, aber der Datensatz muss unter derselben Lizenz veröffentlicht werden. Daher werden unsere Notizquelle Open-Patients und der daraus kuratierte Datensatz MedCalc-Bench beide unter der CC-BY-SA 4.0-Lizenz veröffentlicht.
Basierend auf der Begründung der Lizenzregeln entsprechen sowohl Open-Patients als auch MedCalc-Bench der CC-BY-SA 4.0-Lizenz, jedoch tragen die Autoren dieses Dokuments die volle Verantwortung im Falle einer Rechtsverletzung.
@misc { khandekar2024medcalcbench ,
title = { MedCalc-Bench: Evaluating Large Language Models for Medical Calculations } ,
author = { Nikhil Khandekar and Qiao Jin and Guangzhi Xiong and Soren Dunn and Serina S Applebaum and Zain Anwar and Maame Sarfo-Gyamfi and Conrad W Safranek and Abid A Anwar and Andrew Zhang and Aidan Gilson and Maxwell B Singer and Amisha Dave and Andrew Taylor and Aidong Zhang and Qingyu Chen and Zhiyong Lu } ,
year = { 2024 } ,
eprint = { 2406.12036 } ,
archivePrefix = { arXiv } ,
primaryClass = { id='cs.CL' full_name='Computation and Language' is_active=True alt_name='cmp-lg' in_archive='cs' is_general=False description='Covers natural language processing. Roughly includes material in ACM Subject Class I.2.7. Note that work on artificial languages (programming languages, logics, formal systems) that does not explicitly address natural-language issues broadly construed (natural-language processing, computational linguistics, speech, text retrieval, etc.) is not appropriate for this area.' }
}