Suite molekularer und rechnerischer Arbeitsabläufe für Spatial Total RNA-Sequencing (STRS)
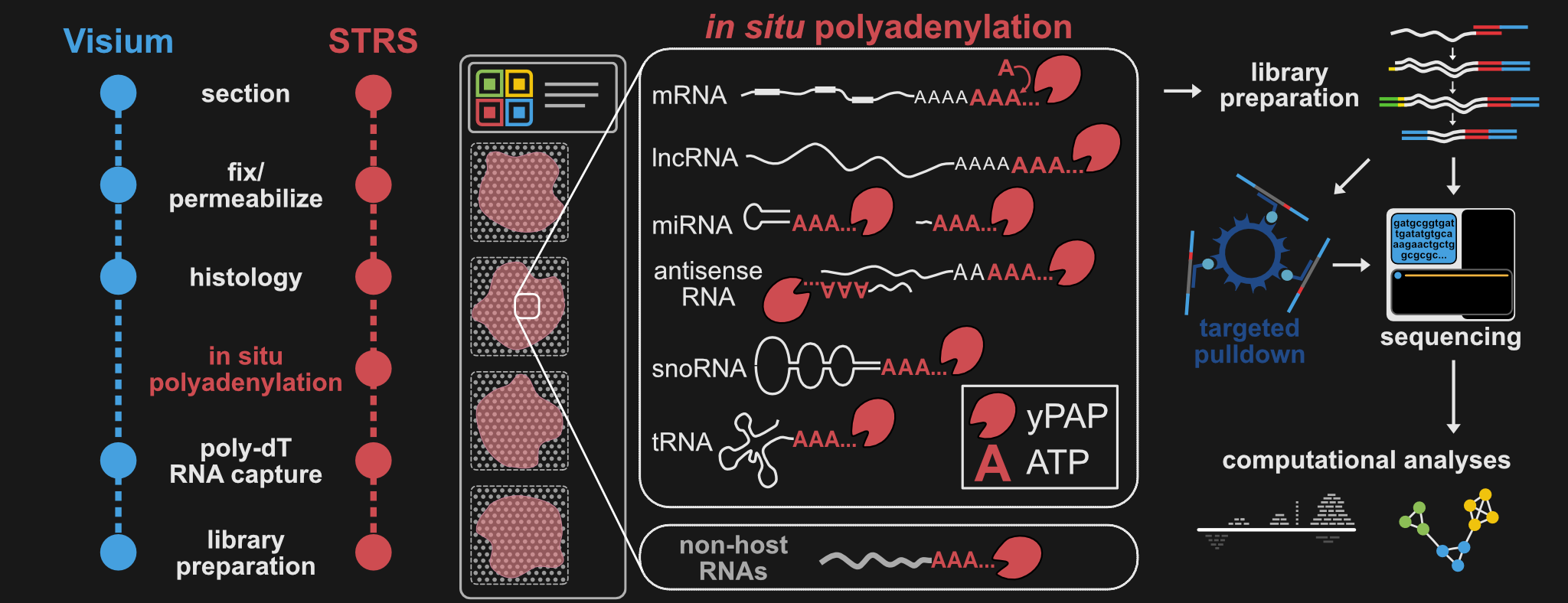
Dieses Repository verfügt über alle Protokolle, Pipelines und Skripte, die Sie zum Ausführen und Analysieren von STRS für fast jede Probe benötigen!
txg_snake an, eine in Arbeit befindliche Pipeline für 10x-Bibliotheken, insbesondere STRS-Datensätze!McKellar et al, Nature Biotechnology , 2022
bioRxiv-Preprint-Link
manuscripts : .pdf-Dateien des Vorabdrucks und des nachfolgenden Manuskripts (hoffentlich!)
protocols : Microsoft Word- und PDF-Dokumente der hier verwendeten Protokolle
pipelines : In unserer Studie verwendete Snakemake-Workflows. Dazu gehören Alignment-Pipelines für STRS-Daten (kallisto, STARsolo und miRge3.0), kleine RNAseq-Daten (STAR & miRge3.0), SmartSeqTotal (kallisto) und VASAdrop (kallisto).
scripts : Der gesamte andere Code, den wir verwendet haben! Enthält hauptsächlich R-Skripte und Hilfsfunktionen, die in unseren räumlichen Analysen verwendet werden.
references : Informationen und Skripte zu Referenzgenomen und Anmerkungen, die in dieser Studie verwendet werden
resources : verschiedene Metadaten, Genlisten und andere Informationen, die wir zur Analyse unserer Daten verwendet haben. Weitere Informationen finden Sie in den README-Dateien in jedem Unterverzeichnis
STRS-Datensätze: GSE200481
SkM/Herz kleine RNAseq-Daten: GSE200480
Einzelkern-Gesamt-RNAseq-Daten (C2C12-Zelllinie): GSE209780
Kleine RNA-Atlas-Daten (Isakova et al, PNAS , 2020): GSE119661
SmartSeqTotal-Daten (Isakova et al, PNAS , 2021): GSE151334
VASAdrop-Daten (Salmen et al, Nature Biotechnology , 2022): GSE176588
Iwijn De Vlaminck ([email protected])
Benjamin D. Cosgrove ([email protected])
David W. McKellar ([email protected])