Dieses Projekt besteht aus verschiedenen Methoden zur Stichprobenerhebung in der Bevölkerung und zur Bewertung verschiedener Methoden. Wir berücksichtigen viele Situationen, die zu einer Verzerrung der Schätzung des Infektionsniveaus auf der Grundlage der Stichprobe führen können, darunter Non-Responder, die Falsch-Positiv-/Negativ-Rate und die Fähigkeit des Übertragungsprofils für Patienten während ihrer Infektionsperiode. Basierend auf dem EpiABM-Modell kann dieses Paket auch die beste Probenahmemethode ausgeben, indem es Simulationen der Krankheitsübertragung durchführt, um den Vorhersagefehler jeder Probenahmemethode zu sehen.
EpiOS ist noch nicht auf PyPI verfügbar, aber das Modul kann lokal per Pip installiert werden. Das Verzeichnis sollte zunächst auf Ihren lokalen Rechner heruntergeladen werden und kann dann mit dem folgenden Befehl installiert werden:
pip install -e .Wir empfehlen Ihnen außerdem, das EpiABM-Modell zu installieren, um die Daten der Infektionssimulation zu generieren. Sie können pyEpiabm zunächst an einen beliebigen Speicherort auf Ihrem Computer herunterladen und dann mit dem folgenden Befehl installieren:
pip install -e path/to/pyEpiabm Auf Dokumentationen kann über das oben stehende docs Badge zugegriffen werden.
Hier ist ein UML-Klassendiagramm für unser Projekt: 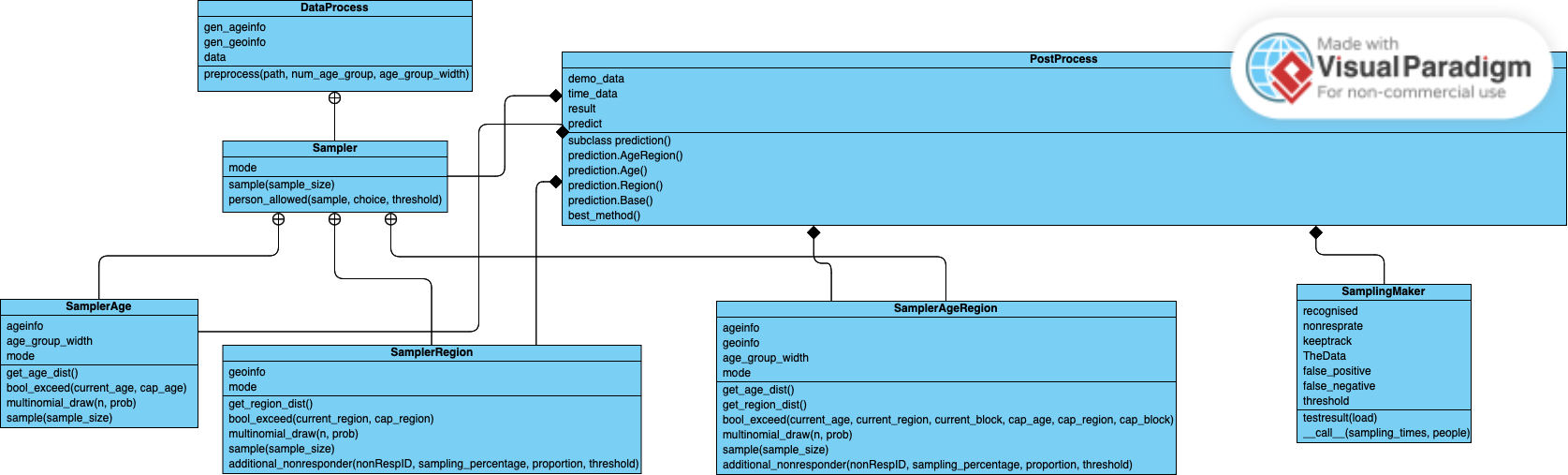
Die Datei params.py enthält alle in diesem Modell erforderlichen Parameter. Darüber hinaus handelt es sich bei den Dateien im input um Beispiele temporärer Dateien, die während der Datenvorverarbeitung generiert wurden. Es wird von den Sampler-Klassen verwendet. Der Parameter data_store_path in jeder Sampler-Klasse ist der Pfad zum Speichern dieser Dateien.
PostProcess , um Diagramme zu erstellen Zunächst müssen Sie ein neues PostProcess Objekt definieren und die von pyEpiabm generierten demografischen Daten demodata und Infektionsdaten timedata eingeben. Zweitens können Sie PostProcess.predict verwenden, um eine Vorhersage basierend auf verschiedenen Stichprobenmethoden durchzuführen. Sie können die Stichprobenmethode, die Sie verwenden möchten, direkt als Methode aufrufen. Geben Sie dann die Zeitpunkte für die Stichprobe und den Stichprobenumfang an. Hier verwenden wir AgeRegion als Stichprobenmethode, [0, 1, 2, 3, 4, 5] als Zeitpunkte für die Stichprobe und 3 als Stichprobengröße. Abschließend können Sie festlegen, ob Sie Nicht-Antwortende berücksichtigen möchten und ob Sie Ihre Ergebnisse mit den wahren Daten vergleichen möchten, indem Sie die Parameter non_responder und comparison angeben.
Als Codebeispiel sehen Sie Folgendes:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)Jetzt wird Ihre Figur unter dem angegebenen Pfad gespeichert!
PostProcess , um die beste Sampling-Methode auszuwählen Zunächst müssen Sie ein neues PostProcess Objekt definieren und die von pyEpiabm generierten demografischen Daten demodata und Infektionsdaten timedata eingeben. Zweitens können Sie PostProcess.best_method verwenden, um die Leistung verschiedener Stichprobenmethoden zu vergleichen. Sie können Methoden angeben, die Sie vergleichen möchten. Geben Sie dann die Stichprobenintervalle und die Stichprobengröße an. Drittens können Sie festlegen, ob Sie Nicht-Antwortende berücksichtigen möchten und ob Sie Ihre Ergebnisse mit den wahren Daten vergleichen möchten, indem Sie die Parameter non_responder und comparison angeben. Da Stichprobenmethoden stochastisch sind, können Sie außerdem die Anzahl der ausgeführten Iterationen angeben, um die durchschnittliche Leistung zu erzielen. Darüber hinaus kann parallel_computation zur Beschleunigung aktiviert werden. Schließlich können Sie hyperparameter_autotune aktivieren, um automatisch die beste Kombination von Hyperparametern zu finden.
Als Codebeispiel sehen Sie Folgendes:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed