AMRICA (AMR Inspector for Cross-Language Alignments) ist ein einfaches Werkzeug zum Ausrichten und visuellen Darstellen von AMRs (Banarescu, 2013), sowohl für zweisprachige Kontexte als auch für die einsprachige Übereinstimmung zwischen Annotatoren. Es basiert auf dem Smatch-System (Cai, 2012) und erweitert es zur Identifizierung von AMR-Interannotator-Übereinstimmungen.
Es ist auch möglich, mit AMRICA manuelle Alignments zu visualisieren, die Sie selbst bearbeitet oder zusammengestellt haben (siehe Common Flags).
Laden Sie die Python-Quelle von Github herunter.
Wir gehen davon aus, dass Sie pip haben. Um die Abhängigkeiten zu installieren (vorausgesetzt, Sie verfügen bereits über die unten genannten Graphviz-Abhängigkeiten), führen Sie einfach Folgendes aus:
pip install argparse_config networkx==1.8 pygraphviz pynlpl
pygraphviz benötigt Graphviz, um zu funktionieren. Unter Linux müssen Sie möglicherweise graphviz libgraphviz-dev pkg-config installieren. Darüber hinaus benötigen Sie zur Vorbereitung zweisprachiger Ausrichtungsdaten GIZA++ und möglicherweise JAMR.
./disagree.py -i sample.amr -o sample_out_dir/
Dieser Befehl liest die AMRs in sample.amr (getrennt durch Leerzeilen) und fügt ihre Graphviz-Visualisierungen in PNG-Dateien ein, die sich in sample_out_dir/ befinden.
Um Visualisierungen von Smatch-Ausrichtungen zu generieren, benötigen wir eine AMR-Eingabedatei mit jedem ::tok oder ::snt -Feld, das tokenisierte Sätze, ::id -Felder mit einer Satz-ID und ::annotator oder ::anno Felder mit einer Annotator-ID enthält. Die Anmerkungen zu einem bestimmten Satz werden der Reihe nach aufgelistet und die erste Anmerkung gilt als Goldstandard für Visualisierungszwecke.
Wenn Sie nur die einzelne Annotation pro Satz ohne Vereinbarung zwischen den Annotatoren visualisieren möchten, können Sie eine AMR-Datei mit nur einem einzelnen Annotator verwenden. In diesem Fall sind Annotator- und Satz-ID-Felder optional. Das resultierende Diagramm ist vollständig schwarz.
Für zweisprachige Alignments beginnen wir mit zwei AMR-Dateien, eine mit den Zielanmerkungen und eine mit den Quellanmerkungen in derselben Reihenfolge, mit den Feldern ::tok und ::id für jede Anmerkung. Wenn wir JAMR-Ausrichtungen für beide Seiten wünschen, fügen wir diese in ein ::alignments -Feld ein.
Die Satzausrichtungen sollten in Form von zwei GIZA++-Ausrichtungs-.NBEST-Dateien vorliegen, einer Quelle-Ziel- und einer Ziel-Quelle. Um diese zu generieren, verwenden Sie das Flag --nbestalignments in Ihrer GIZA++-Konfigurationsdatei und stellen Sie es auf Ihre bevorzugte nbest-Anzahl ein.
Flags können entweder in der Befehlszeile oder in einer Konfigurationsdatei gesetzt werden. Der Speicherort einer Konfigurationsdatei kann mit -c CONF_FILE in der Befehlszeile festgelegt werden.
Zusätzlich zu --conf_file gibt es mehrere andere Flags, die sowohl für einsprachigen als auch für zweisprachigen Text gelten. --outdir DIR ist das einzige erforderliche Verzeichnis und gibt das Verzeichnis an, in das wir die Bilddateien schreiben.
Die optionalen gemeinsamen Flags sind:
--verbose um Sätze auszudrucken, während wir sie ausrichten.--no-verbose um eine ausführliche Standardeinstellung zu überschreiben.--json FILE.json um die Ausrichtungsdiagramme in eine .json-Datei zu schreiben.--num_restarts N um die Anzahl der zufälligen Neustarts anzugeben, die Smatch ausführen soll.--align_out FILE.csv um die Ausrichtungen in eine Datei zu schreiben.--align_in FILE.csv um die Ausrichtungen von der Festplatte zu lesen, anstatt Smatch auszuführen.--layout um den Layoutparameter in graphviz zu ändern.Die Ausrichtungs-.csv-Dateien haben ein Format, bei dem jeder Diagrammübereinstimmungssatz durch eine Leerzeile getrennt ist und jede Zeile innerhalb eines Satzes entweder einen Kommentar oder eine Zeile enthält, die eine Ausrichtung angibt. Zum Beispiel:
3 它 - 1 it
2 多长 - -1
-1 - 2 take
Die durch Tabulatoren getrennten Felder sind der Testknotenindex (wie von Smatch verarbeitet), die Testknotenbezeichnung, der Goldknotenindex und die Goldknotenbezeichnung.
Die einsprachige Ausrichtung erfordert ein zusätzliches Flag, --infile FILE.amr , wobei FILE.amr auf den Speicherort der AMR-Datei festgelegt ist.
Im Folgenden finden Sie eine Beispielkonfigurationsdatei:
[default]
infile: data/events_amr.txt
outdir: data/events_png/
json: data/events.json
verbose
Bei der zweisprachigen Ausrichtung sind mehr Flags erforderlich.
--src_amr FILE für die AMR-Quellanmerkungsdatei.--tgt_amr FILE für die AMR-Zielanmerkungsdatei.--align_tgt2src FILE.A3.NBEST für die GIZA++ .NBEST-Datei, die Ziel an Quelle ausrichtet (mit Ziel als vcb1), generiert mit --nbestalignments N--align_src2tgt FILE.A3.NBEST für die GIZA++ .NBEST-Datei, die Quelle und Ziel ausrichtet (mit Quelle als vcb1), generiert mit --nbestalignments N Wenn nun --nbestalignments N auf >1 gesetzt wurde, sollten wir es mit --num_aligned_in_file angeben. Wenn wir nur die Spitze zählen wollen --num_align_read .
--nbestalignments ist ein schwierig zu verwendendes Flag, da es nur bei einem letzten Ausrichtungslauf generiert wird. Ich selbst konnte es nur mit den Standardeinstellungen von GIZA++ zum Laufen bringen.
Da AMRICA eine Variante von Smatch ist, sollte man zunächst Smatch verstehen. Smatch versucht, eine Übereinstimmung zwischen den variablen Knoten zweier AMR-Darstellungen desselben Satzes zu identifizieren, um die Übereinstimmung zwischen Annotatoren zu messen. Das Matching sollte so gewählt werden, dass der Smatch-Score maximiert wird, der für jede Kante, die in beiden Diagrammen erscheint, einen Punkt zuweist und in drei Kategorien fällt. Jede Kategorie wird in der folgenden Anmerkung „Es hat nicht lange gedauert“ veranschaulicht.
(t / take-10
:ARG0 (i / it)
:ARG1 (l2 / long
:polarity -))
(instance, t, take-10)(ARG0, t, i)(polarity, l2, -)Da das Problem, die Übereinstimmung zu finden, die den Smatch-Score maximiert, NP-vollständig ist, verwendet Smatch einen Hill-Climbing-Algorithmus, um die beste Lösung anzunähern. Beim Seeding wird jeder Knoten nach Möglichkeit mit einem Knoten abgeglichen, der seine Bezeichnung teilt, und die übrigen Knoten im kleineren Diagramm (im Folgenden „Ziel“) werden nach dem Zufallsprinzip zugeordnet. Smatch führt dann einen Schritt aus, indem es die Aktion findet, die die Punktzahl am meisten erhöht, indem es entweder die Übereinstimmungen zweier Zielknoten vertauscht oder eine Übereinstimmung von seinem Quellknoten auf einen nicht übereinstimmenden Quellknoten verschiebt. Dieser Schritt wird wiederholt, bis kein Schritt den Smatch-Score sofort erhöhen kann.
Um lokale Optima zu vermeiden, wird Smatch im Allgemeinen fünfmal neu gestartet.
Für technische Details zum Innenleben von AMRICA ist es möglicherweise nützlicher, unser NAACL-Demopapier zu lesen.
AMRICA beginnt damit, alle konstanten Knoten durch variable Knoten zu ersetzen, die Instanzen der Konstantenbezeichnung sind. Dies ist notwendig, damit wir sowohl die konstanten Knoten als auch die Variablen ausrichten können. Die einzigen Punkte, die zum AMRICA-Score hinzugefügt werden, stammen also aus der Zuordnung von variablen Kanten und Instanzbeschriftungen.
Während Smatch versucht, jeden Knoten im kleineren Diagramm einem Knoten im größeren Diagramm zuzuordnen, entfernt AMRICA Übereinstimmungen, die den modifizierten Smatch-Score oder AMRICA-Score nicht erhöhen.
AMRICA generiert dann Bilddateien aus Graphviz-Diagrammen der Ausrichtungen. Wenn ein Knoten oder eine Kante nur in den Golddaten vorkommt, ist er rot. Wenn dieser Knoten oder diese Kante nur in den Testdaten vorkommt, ist er blau. Wenn der Knoten oder die Kante in unserer endgültigen Ausrichtung eine Übereinstimmung aufweist, ist er schwarz.
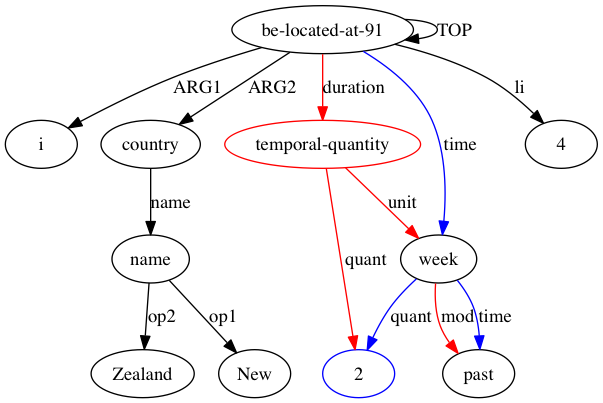
In AMRICA fügen wir, anstatt einen Punkt für jedes perfekt passende Instanzlabel hinzuzufügen, einen Punkt hinzu, der auf einem Wahrscheinlichkeitswert für die Ausrichtung dieser Labels basiert. Der Likelihood-Score ℓ(aLt,Ls[i]|Lt,Wt,Ls,Ws) mit Zielbeschriftungssatz Lt, Quellbeschriftungssatz Ls, Zielsatz Wt, Quellsatz Ws und Ausrichtung aLt,Ls[i]-Zuordnung Lt[ i] auf ein Label Ls[aLt,Ls[i]], wird aus einer Wahrscheinlichkeit berechnet, die durch die folgenden Regeln definiert ist:
Im Allgemeinen scheint das zweisprachige AMRICA mehr zufällige Neustarts zu erfordern als das einsprachige AMRICA, um eine gute Leistung zu erzielen. Dieser Neustartzähler kann mit dem Flag --num_restarts geändert werden.
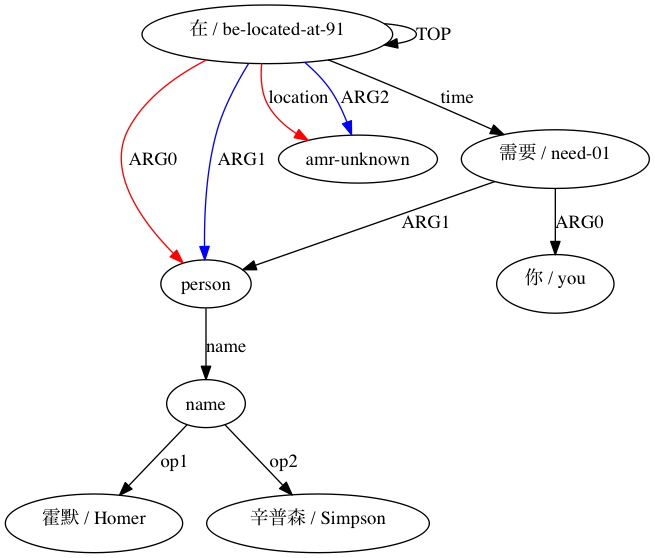
Wir können beobachten, inwieweit die Verwendung von Smatch-ähnlichen Näherungen (hier mit 20 zufälligen Initialisierungen) die Genauigkeit gegenüber der Auswahl wahrscheinlicher Übereinstimmungen aus Rohausrichtungsdaten (intelligente Initialisierung) verbessert. Für eine Paarung, die von (Xue 2014) als strukturell kompatibel erklärt wurde.
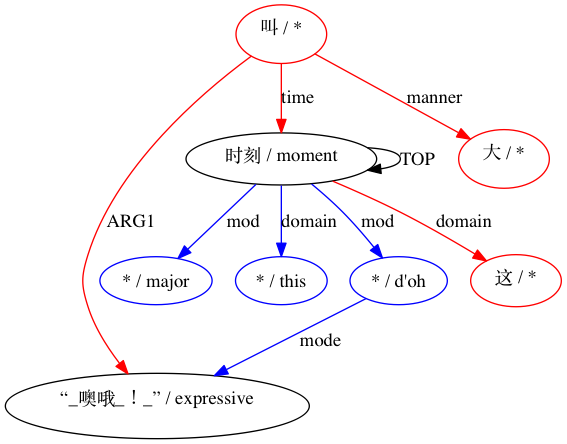
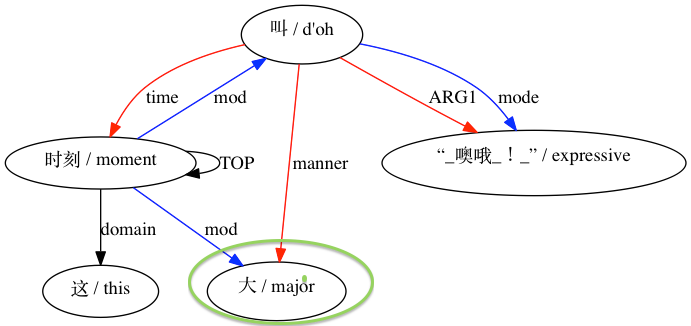
Für eine Paarung, die als inkompatibel gilt:
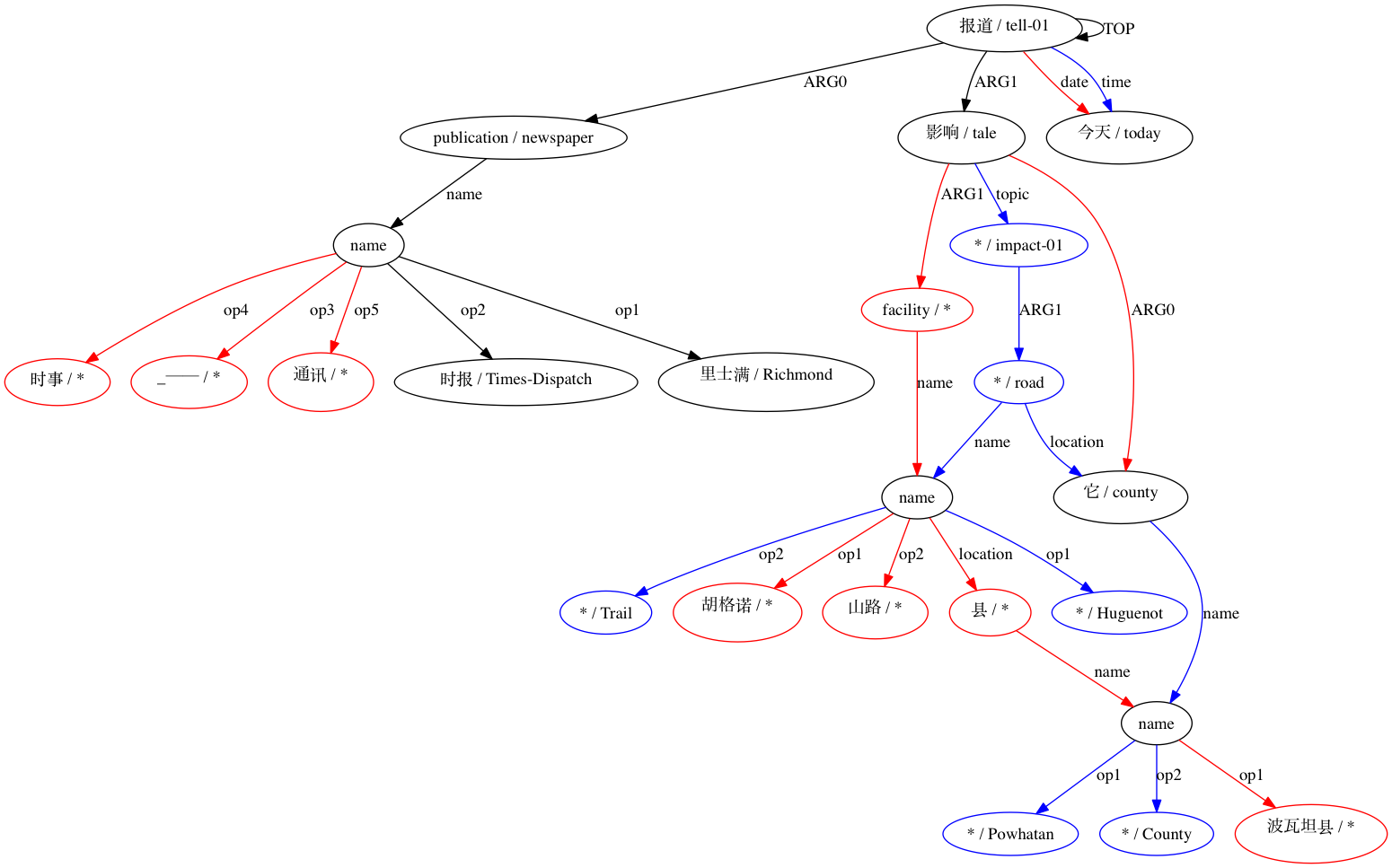
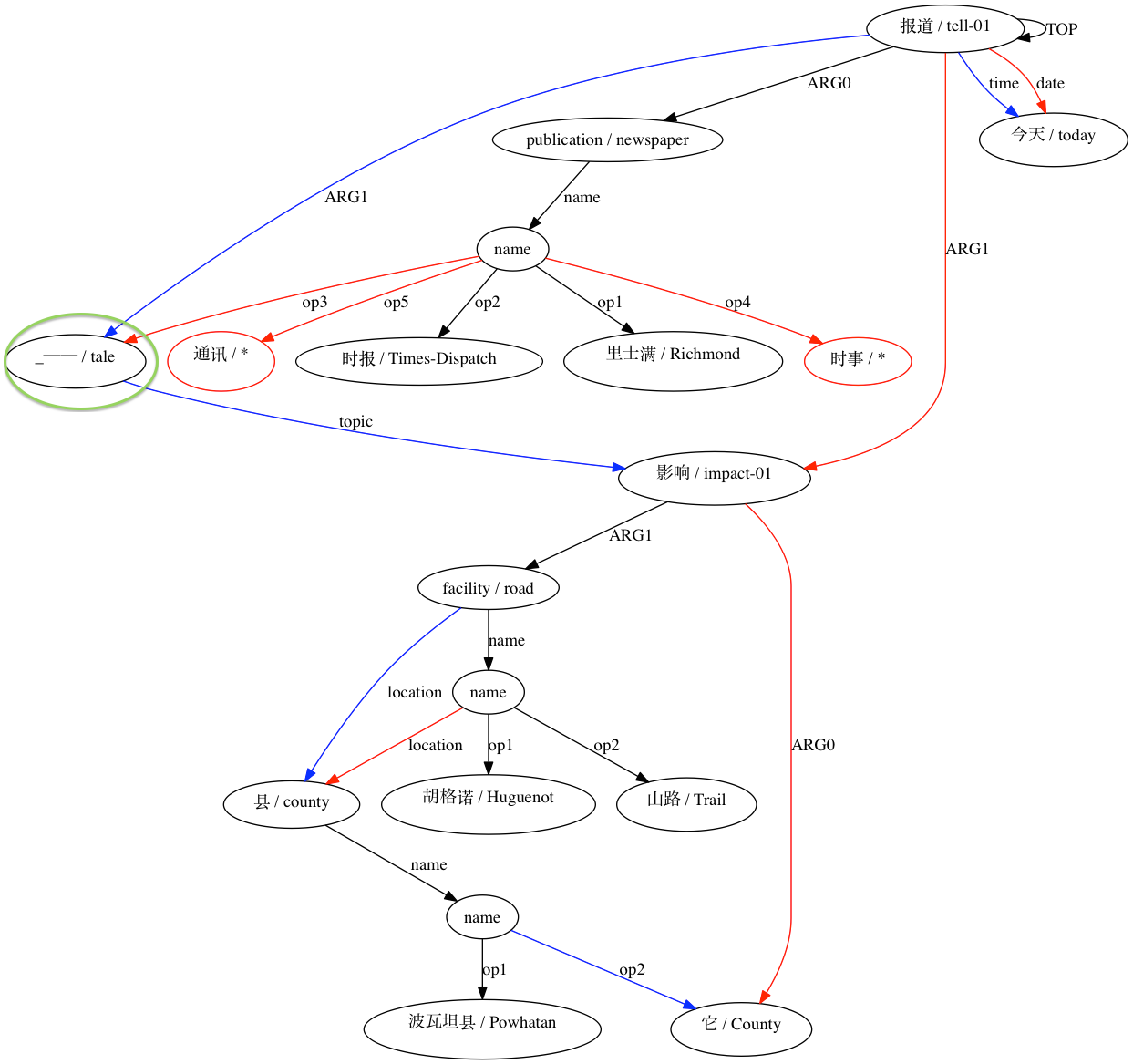
Diese Software wurde teilweise mit Unterstützung der National Science Foundation (USA) unter den Auszeichnungen 1349902 und 0530118 entwickelt. Die University of Edinburgh ist eine in Schottland registrierte gemeinnützige Einrichtung mit der Registrierungsnummer SC005336.