
Dieses Repository ist die offizielle Implementierung von CELL-E 2: Translating Proteins to Pictures and Back with a Bidirection Text-to-Image Transformer.
Erstellen Sie eine virtuelle Umgebung und installieren Sie die erforderlichen Pakete über:
pip install -r requirements.txt
Als nächstes installieren Sie torch = 2.0.0 mit der entsprechenden CUDA-Version
Modelle sind auf Hugging Face verfügbar.
Wir haben auch zwei Bereiche zur Verfügung, in denen Sie Vorhersagen auf Ihren eigenen Daten treffen können!
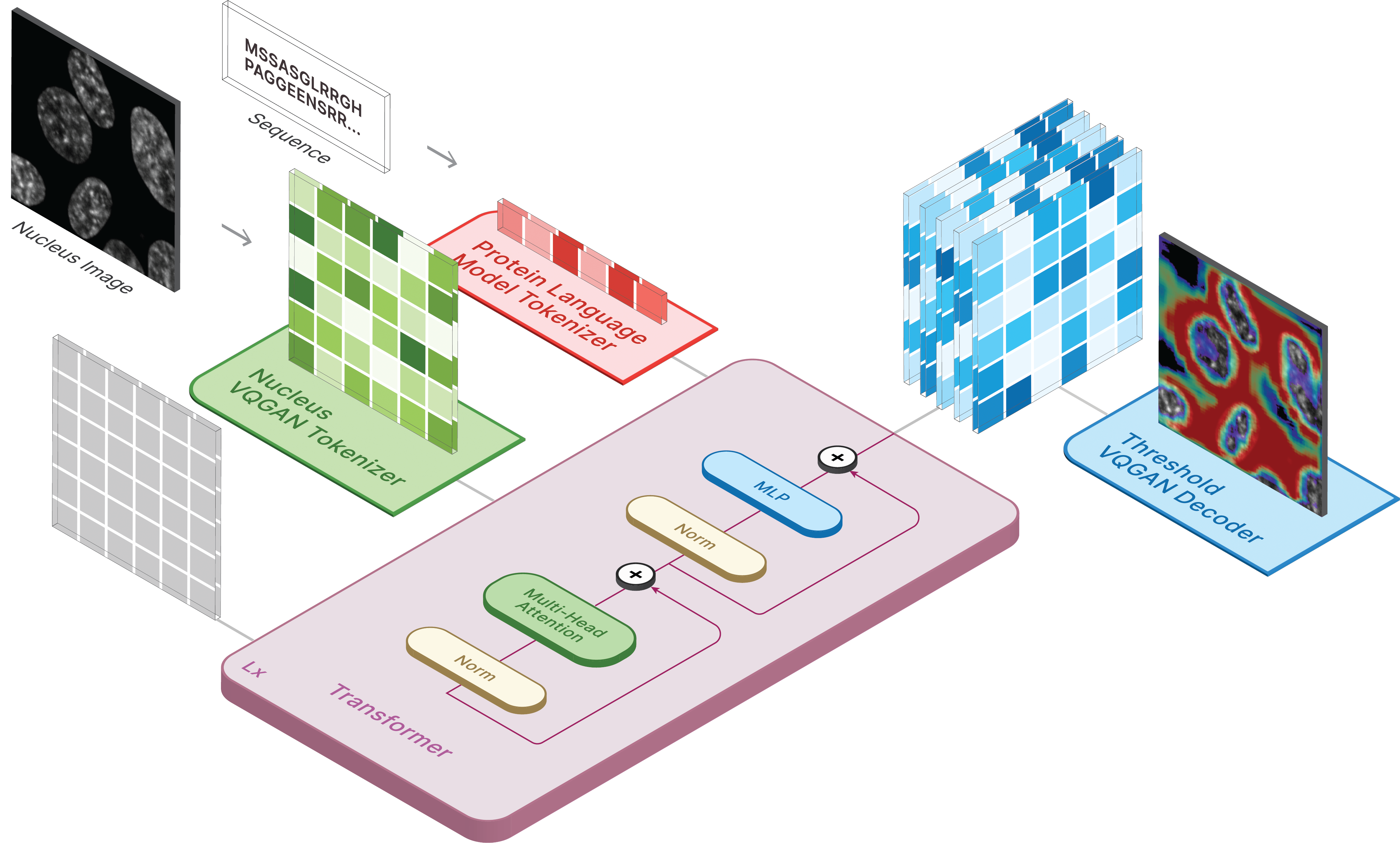
Um Bilder zu generieren, legen Sie das gespeicherte Modell als ckpt_path fest. Diese Methode kann instabil sein. Sehen Sie sich daher Demo.ipynb an, um eine andere Möglichkeit zum Laden zu sehen.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
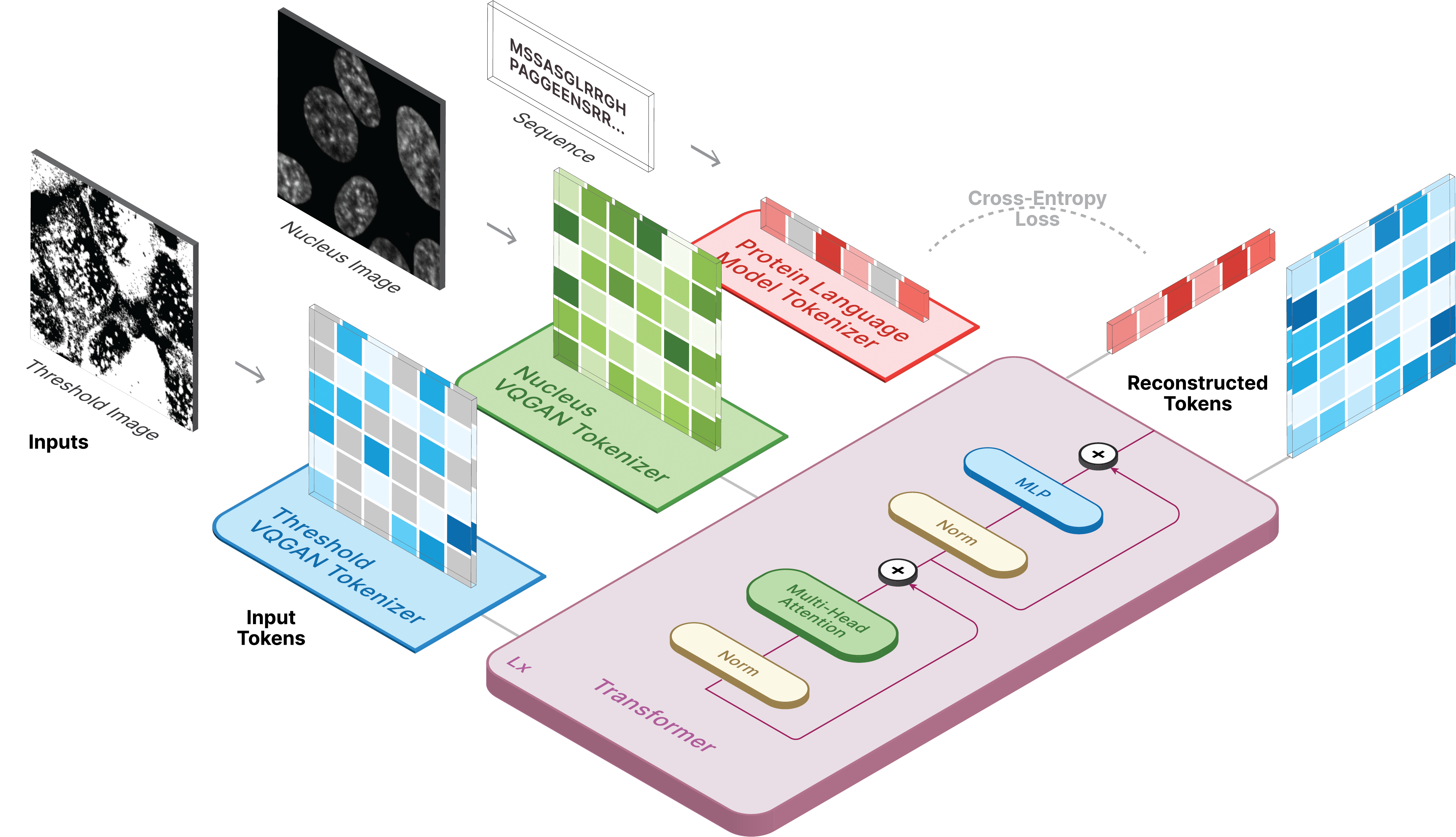
progress = True )Das Training für CELL-E erfolgt in 3 Phasen:
Wenn Sie das Protein-Schwellenwertbild verwenden, legen Sie für den Datensatz threshold: True fest.
Wir verwenden eine leicht modifizierte Version des Taming-Transformers-Codes.
Führen Sie zum Trainieren das folgende Skript aus:
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
Weitere Flags wie --gpus finden Sie im Original-Repo.
Wir stellen Skripte zum Herunterladen von Human Protein Atlas- und OpenCell-Bildern im Skriptordner bereit. Für den Datenlader wird ein data_csv benötigt. Sie müssen eine CSV-Datei generieren, die die Spalten nucleus_image_path , protein_image_path , metadata_path , split (train oder val) und sequence (optional) enthält. Es wird davon ausgegangen, dass diese Datei im selben allgemeinen data wie die Bilder und Metadatendateien vorhanden ist.
Metadaten sind ein JSON, das jede Proteinsequenz begleiten sollte. Wenn eine Sequenz nicht im data_csv erscheint, muss sie in metadata.json mit einem Schlüssel namens protein_sequence erscheinen.
Das Hinzufügen weiterer Informationen kann für die Abfrage einzelner Proteine hilfreich sein. Sie können über retrieve_metadata abgerufen werden, wodurch eine self.metadata Variable innerhalb des Datensatzobjekts erstellt wird.
Führen Sie zum Trainieren das folgende Skript aus:
python celle_main.py --base configs/celle.yaml -t True
Geben Sie --gpus im gleichen Format wie VQGAN an.
CELL-E enthält die folgenden Optionen:
ckpt_path : Vorheriges CELL-E 2-Training fortsetzen. Gespeichertes Modell mit state_dictvqgan_model_path : Gespeichertes Proteinbildmodell (mit state_dict) für Proteinbild-Encodervqgan_config_path : Gespeichertes Protein-Image-Modell im Yaml-Formatcondition_model_path : Gespeichertes Zustandsmodell (Kernmodell) (mit state_dict) für Proteinbild-Encodercondition_config_path : Gespeichertes Bedingungsmodell (Nukleus) im Yaml-Formatnum_images : 1, wenn nur Proteinbild-Encoder verwendet wird, 2, wenn Bedingungsbild-Encoder einbezogen wirdimage_key : nucleus , target oder thresholddim : Dimension der Einbettung des Sprachmodellsnum_text_tokens : Gesamtzahl der Token im Sprachmodell (33 für ESM-2)text_seq_len : Gesamtzahl der berücksichtigten Aminosäurendepth : Tiefe des Transformatormodells, tiefer ist normalerweise besser auf Kosten des VRAMheads : Anzahl der Köpfe, die bei der mehrköpfigen Aufmerksamkeit verwendet werdendim_head : Größe der Aufmerksamkeitsköpfeattn_dropout : Aufmerksamkeits-Abbrecherquote im Trainingff_dropout : Feed-Forward-Abbrecherquote im Trainingloss_img_weight : Auf die Bildrekonstruktion angewendete Gewichtung. Textgewicht = 1loss_text_weight : Auf die Bedingungsbildrekonstruktion angewendete Gewichtung.stable : Normiert Gewichte (für den Fall, dass explodierende Farbverläufe auftreten)learning_rate : Lernrate für den Adam-Optimierermonitor : Parameter zum Speichern von Modellen Bitte zitieren Sie uns, wenn Sie sich entscheiden, unseren Code für einen Teil Ihrer Forschung zu verwenden.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}