
Wir sind stolz, Asclepius vorzustellen, ein fortschrittlicheres klinisches Modell für große Sprachen. Da dieses Modell anhand synthetischer klinischer Notizen trainiert wurde, ist es über Huggingface öffentlich zugänglich. Wenn Sie erwägen, CAMEL zu verwenden, empfehlen wir Ihnen dringend, stattdessen auf Asclepius umzusteigen. Weitere Informationen finden Sie unter diesem Link.
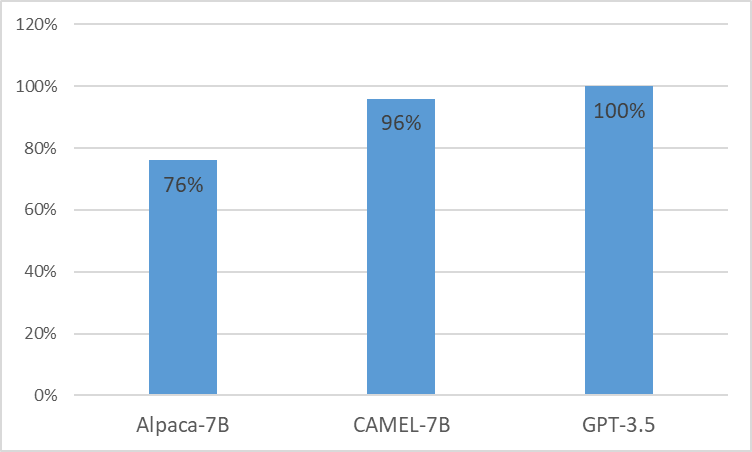
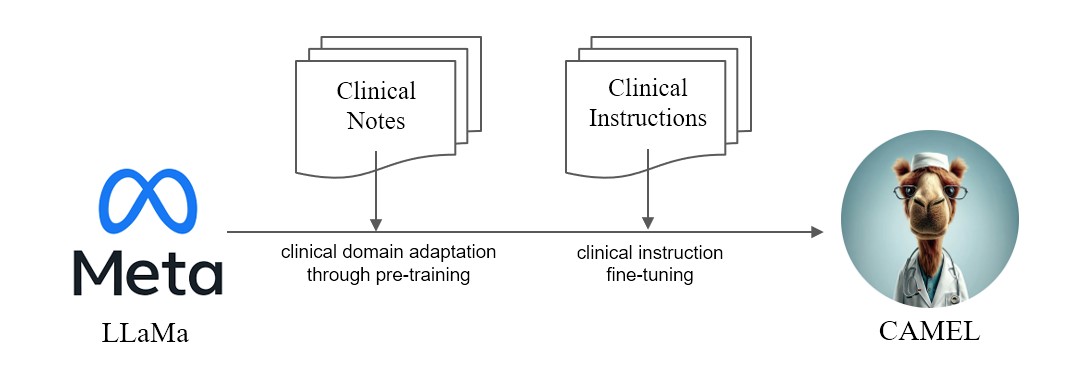
Wir präsentieren CAMEL , ein klinisch angepasstes Modell von LLaMA. Als LLaMA für seine Gründung wird CAMEL außerdem vorab auf die klinischen Hinweise zu MIMIC-III und MIMIC-IV geschult und anhand klinischer Anweisungen verfeinert (Abbildung 2). Unsere vorläufige Bewertung mit GPT-4-Bewertung zeigt, dass CAMEL über 96 % der Qualität von OpenAIs GPT-3.5 erreicht (Abbildung 1). In Übereinstimmung mit den Datennutzungsrichtlinien unserer Quelldaten werden sowohl unser Instruktionsdatensatz als auch unser Modell auf PhysioNet mit beglaubigtem Zugriff veröffentlicht. Um die Replikation zu erleichtern, werden wir außerdem den gesamten Code veröffentlichen, sodass einzelne Gesundheitseinrichtungen unser Modell anhand ihrer eigenen klinischen Notizen reproduzieren können. Weitere Einzelheiten finden Sie in unserem Blogbeitrag .
Aufgrund des Lizenzproblems von MIMIC- und i2b2-Datensätzen können wir den Befehlsdatensatz und die Prüfpunkte nicht veröffentlichen. Wir würden unser Modell und unsere Daten innerhalb weniger Wochen über physionet veröffentlichen.
conda create -n camel python=3.9 -y
conda activate camel
conda install pytorch torchvision torchaudio pytorch-cuda=11.8 -c pytorch -c nvidia -y
pip install pandarallel pandas jupyter numpy datasets sentencepiece openai fire
pip install git+https://github.com/huggingface/transformers.git@871598be552c38537bc047a409b4a6840ba1c1e4
$ python pretraining_preprocess/mimiciii_preproc.py --mimiciii_note_path {MIMICIII_NOTE_PATH} --output_path {OUTPUT_PATH}$ python pretraining_preprocess/mimiciv_preproc.py --discharge_note_path {DISCHAGE_NOTE_PATH} --radiology_note_path {RADIOLOGY_NOTE_PATH} --output_path {OUTPUT_PATH}$ python pretraining_preprocess/tokenize_data.py --data_path {DATA_PATH} --save_path {SAVE_PATH} $ torchrun --nproc_per_node=8 --master_port={YOUR_PORT}
src/train.py
--model_name_or_path "decapoda-research/llama-7b-hf"
--data_path {DATA_FILE}
--bf16 True
--output_dir ./checkpoints
--num_train_epochs 1
--per_device_train_batch_size 2
--per_device_eval_batch_size 2
--gradient_accumulation_steps 8
--evaluation_strategy "no"
--save_strategy "steps"
--save_steps 1000
--learning_rate 2e-5
--weight_decay 0.
--warmup_ratio 0.03
--lr_scheduler_type "cosine"
--logging_steps 1
--fsdp "full_shard auto_wrap"
--fsdp_transformer_layer_cls_to_wrap 'LlamaDecoderLayer'
--tf32 True
--model_max_length 2048
--gradient_checkpointing True
HINWEIS: Um Anweisungen zu generieren, sollten Sie die zertifizierte Azure Openai API verwenden.
Befehlsgenerierung
OPENAI_API_KEYOPENAI_API_BASEOPENAI_DEPLOYMENT_NAME$ python instructino/preprocess_note.py$ python instruction/de_id_gen.py --input {PREPROCESSED_NOTES} --output {OUTPUT_FILE_1} --mode inst$ python instruction/de_id_postprocess.py --input {OUTPUT_FILE_1} --output {OUTPUT_FILE_2}$ python instruction/de_id_gen.py --input {OUTPUT__FILE_2} --output {inst_output/OUTPUT_FILE_deid} --mode ans$ python instruction/instructtion_gen.py --input {PREPROCESSED_NOTES} --output {inst_output/OUTPUT_FILE} --source {mimiciii, mimiciv, i2b2}$ python instruction/merge_data.py --data_path {inst_output} --output {OUTPUT_FILE_FINAL}Führen Sie die Feinabstimmung der Anweisung aus
nproc_per_node und gradient accumulate step an Ihre Hardware an (globale Batchgröße = 128). $ torchrun --nproc_per_node=8 --master_port={YOUR_PORT}
src/instruction_ft.py
--model_name_or_path "decapoda-research/llama-7b-hf"
--data_path {OUTPUT_FILE_FINAL}
--bf16 True
--output_dir ./checkpoints
--num_train_epochs 3
--per_device_train_batch_size 2
--per_device_eval_batch_size 2
--gradient_accumulation_steps 8
--evaluation_strategy "no"
--save_strategy "epoch"
--learning_rate 2e-5
--weight_decay 0.
--warmup_ratio 0.03
--lr_scheduler_type "cosine"
--logging_steps 1
--fsdp "full_shard auto_wrap"
--fsdp_transformer_layer_cls_to_wrap 'LlamaDecoderLayer'
--tf32 True
--model_max_length 2048
--gradient_checkpointing True
--ddp_timeout 18000
Führen Sie das Modell auf MTSamples aus
CUDA_VISIBLE_DEVICES=0 python src/evaluate.py
--model_name {MODEL_PATH}
--data_path eval/mtsamples_instructions.json
--output_path {OUTPUT_PATH}
mtsamples_results.json im Ordner eval bereit.Führen Sie GPT-4 zur Evaluierung aus
python eval/gpt4_evaluate.py --input {INPUT_PATH} --output {OUTPUT_PATH}
@misc{CAMEL,
title = {CAMEL : Clinically Adapted Model Enhanced from LLaMA},
author = {Sunjun Kweon and Junu Kim and Seongsu Bae and Eunbyeol Cho and Sujeong Im and Jiyoun Kim and Gyubok Lee and JongHak Moon and JeongWoo Oh and Edward Choi},
month = {May},
year = {2023}
publisher = {GitHub},
journal = {GitHub repository},
howpublished = {url{https://github.com/starmpcc/CAMEL}},
}