Dieses Repository enthält den Quellcode für unseren Artikel YNet, der zur Veröffentlichung bei MICCAI'18 angenommen wurde.
Y-Net identifizierte korrekt klassifizierte Gewebe, die für die Diagnose nicht wichtig waren. Beispielsweise wurde Stroma als wichtiges Gewebe identifiziert, Blut jedoch nicht. Stroma ist eine wichtige Gewebemarkierung für die Diagnose von Brustkrebs [1] und das Entfernen von Informationen über Stroma verringerte die Genauigkeit der diagnostischen Klassifizierung um etwa 4 %. Weitere Einzelheiten finden Sie im Dokument.
[1] Beck, Andrew H., et al. „Eine systematische Analyse der Brustkrebsmorphologie deckt Stromamerkmale auf, die mit dem Überleben verbunden sind.“ Wissenschaftliche translationale Medizin 3.108 (2011): 108ra113-108ra113.
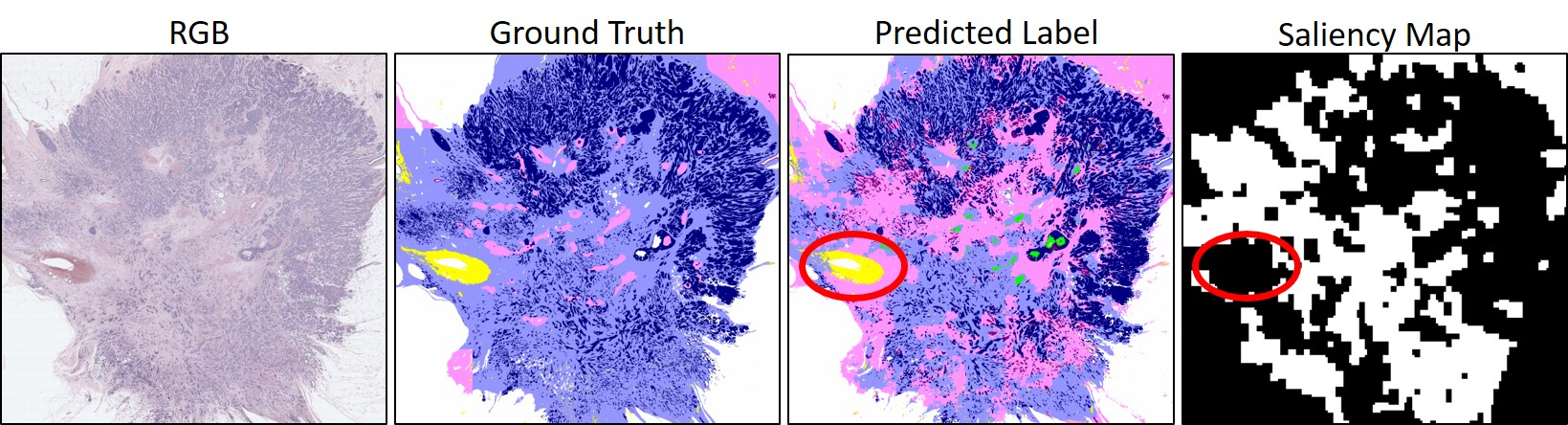
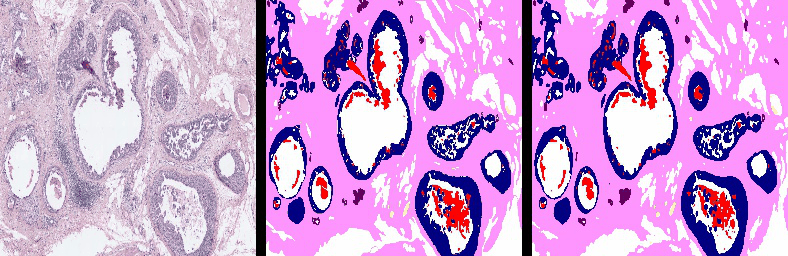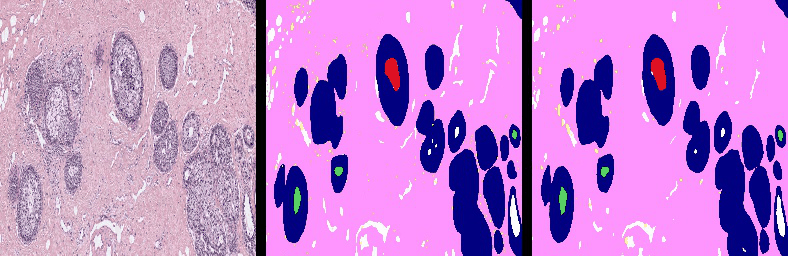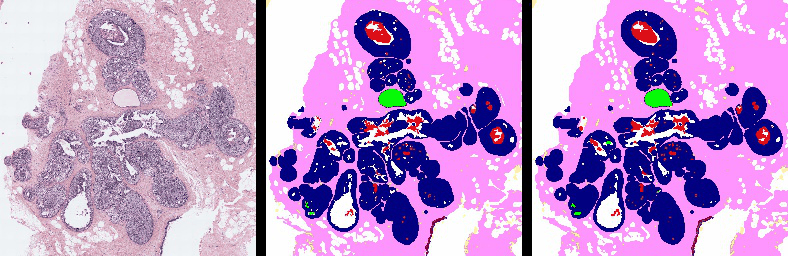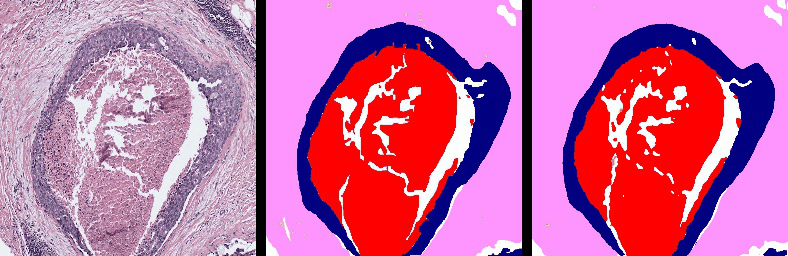
Einige Segmentierungsergebnisse (Links: RGB WSI, Mitte: Ground Truth, Rechts: Vorhersagen von Y-Net)
YNet wird in zwei Phasen trainiert:
Um diesen Code auszuführen, benötigen Sie folgende Bibliotheken:
Wir empfehlen die Verwendung von Anaconda. Wir haben unseren Code auf Ubuntu 16.04 getestet.
Wenn Y-Net für Ihre Forschung nützlich ist, zitieren Sie bitte unseren Artikel.
@inproceedings{mehta2018ynet,
title={{Y-Net: Joint Segmentation and Classification for Diagnosis of Breast Biopsy Images}},
author={Sachin Mehta and Ezgi Mercan and Jamen Bartlett and Donald Weaver and Joann Elmore and Linda Shapiro},
booktitle={International Conference on Medical image computing and computer-assisted intervention},
year={2018},
organization={Springer}
}
@article{mehta2018espnet,
title={ESPNet: Efficient Spatial Pyramid of Dilated Convolutions for Semantic Segmentation},
author={Sachin Mehta, Mohammad Rastegari, Anat Caspi, Linda Shapiro, and Hannaneh Hajishirzi},
journal={European Conference in Computer Vision (ECCV)},
year={2018}
}
Dieser Code wird unter den gleichen Lizenzbedingungen wie ESPNet veröffentlicht.