SHAP (SHapley Additive exPlanations) ist ein spieltheoretischer Ansatz zur Erklärung der Ausgabe jedes Modells für maschinelles Lernen. Es verbindet optimale Kreditzuweisung mit lokalen Erklärungen unter Verwendung der klassischen Shapley-Werte aus der Spieltheorie und ihrer zugehörigen Erweiterungen (Einzelheiten und Zitate finden Sie in den Artikeln).
SHAP kann entweder von PyPI oder Conda-Forge installiert werden:
pip install shap oder conda install -c conda-forge shap
Während SHAP die Ausgabe jedes Modells für maschinelles Lernen erklären kann, haben wir einen exakten Hochgeschwindigkeitsalgorithmus für Baumensemble-Methoden entwickelt (siehe unser Nature MI-Papier). Schnelle C++-Implementierungen werden für XGBoost- , LightGBM- , CatBoost- , Scikit-Learn- und Pyspark- Tree-Modelle unterstützt:
import xgboost
import shap
# train an XGBoost model
X , y = shap . datasets . california ()
model = xgboost . XGBRegressor (). fit ( X , y )
# explain the model's predictions using SHAP
# (same syntax works for LightGBM, CatBoost, scikit-learn, transformers, Spark, etc.)
explainer = shap . Explainer ( model )
shap_values = explainer ( X )
# visualize the first prediction's explanation
shap . plots . waterfall ( shap_values [ 0 ])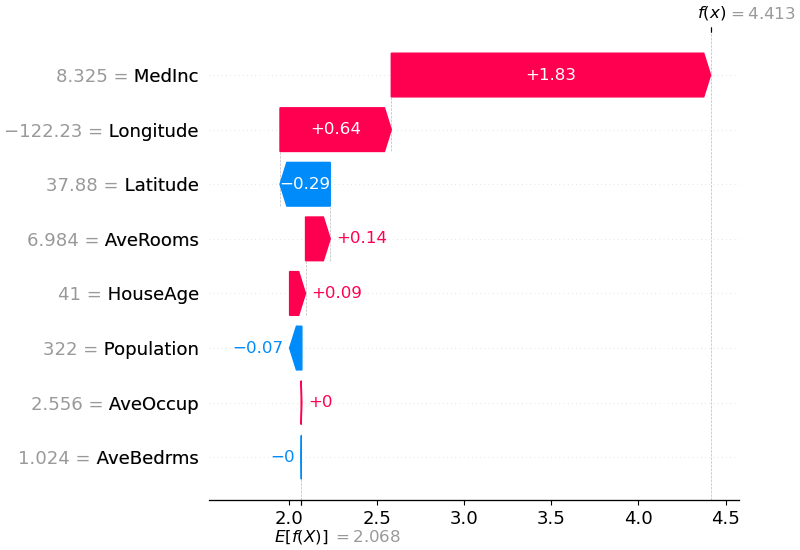
Die obige Erklärung zeigt Funktionen, die jeweils dazu beitragen, die Modellausgabe vom Basiswert (der durchschnittlichen Modellausgabe über den von uns übergebenen Trainingsdatensatz) zur Modellausgabe zu verschieben. Merkmale, die die Vorhersage nach oben treiben, werden in Rot angezeigt, diejenigen, die die Vorhersage nach unten treiben, werden in Blau angezeigt. Eine andere Möglichkeit, dieselbe Erklärung zu visualisieren, ist die Verwendung eines Kraftdiagramms (diese werden in unserem Nature BME-Artikel vorgestellt):
# visualize the first prediction's explanation with a force plot
shap . plots . force ( shap_values [ 0 ])
Wenn wir viele Kraftdiagrammerklärungen wie die oben gezeigte nehmen, sie um 90 Grad drehen und sie dann horizontal stapeln, können wir Erklärungen für einen gesamten Datensatz sehen (im Notizbuch ist dieses Diagramm interaktiv):
# visualize all the training set predictions
shap . plots . force ( shap_values [: 500 ])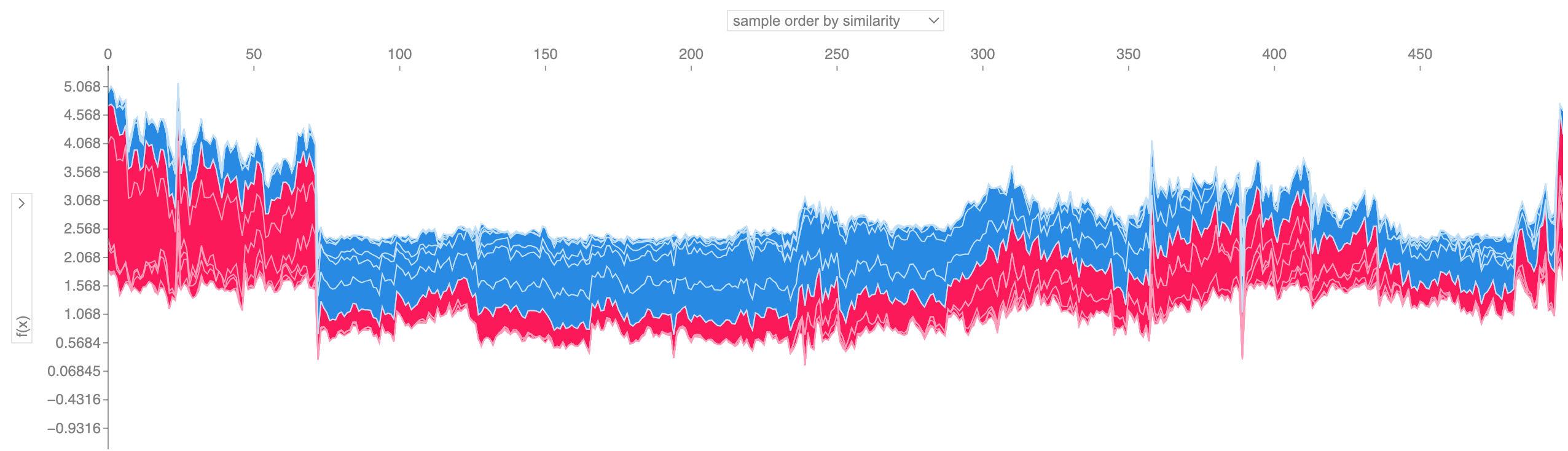
Um zu verstehen, wie sich ein einzelnes Feature auf die Ausgabe des Modells auswirkt, können wir den SHAP-Wert dieses Features im Vergleich zum Wert des Features für alle Beispiele in einem Datensatz grafisch darstellen. Da SHAP-Werte die Verantwortung eines Features für eine Änderung in der Modellausgabe darstellen, stellt das Diagramm unten die Änderung des vorhergesagten Hauspreises dar, wenn sich der Breitengrad ändert. Die vertikale Streuung bei einem einzelnen Breitengradwert stellt Interaktionseffekte mit anderen Merkmalen dar. Um diese Wechselwirkungen besser sichtbar zu machen, können wir ein anderes Merkmal einfärben. Wenn wir den gesamten Erklärungstensor an das color übergeben, wählt das Streudiagramm das beste Merkmal zum Färben aus. In diesem Fall wird der Längengrad ausgewählt.
# create a dependence scatter plot to show the effect of a single feature across the whole dataset
shap . plots . scatter ( shap_values [:, "Latitude" ], color = shap_values )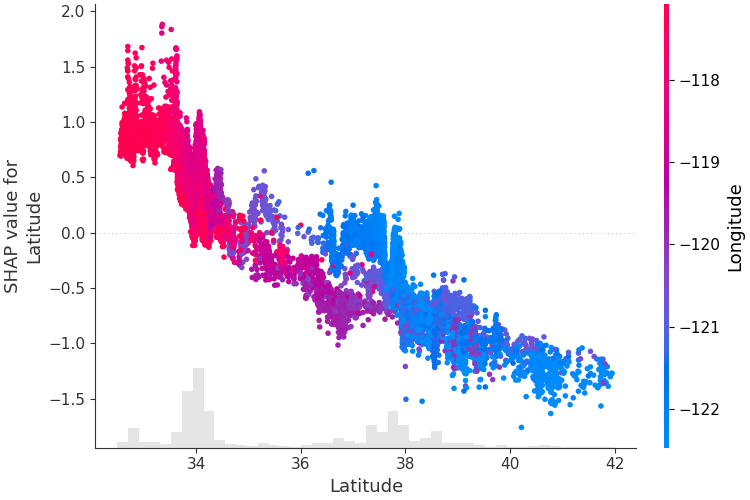
Um einen Überblick darüber zu erhalten, welche Features für ein Modell am wichtigsten sind, können wir die SHAP-Werte jedes Features für jede Stichprobe grafisch darstellen. Das Diagramm unten sortiert Features nach der Summe der SHAP-Wertgrößen über alle Stichproben und verwendet SHAP-Werte, um die Verteilung der Auswirkungen anzuzeigen, die jedes Feature auf die Modellausgabe hat. Die Farbe stellt den Merkmalswert dar (rot hoch, blau niedrig). Dies zeigt beispielsweise, dass ein höheres Durchschnittseinkommen den prognostizierten Immobilienpreis verbessert.
# summarize the effects of all the features
shap . plots . beeswarm ( shap_values )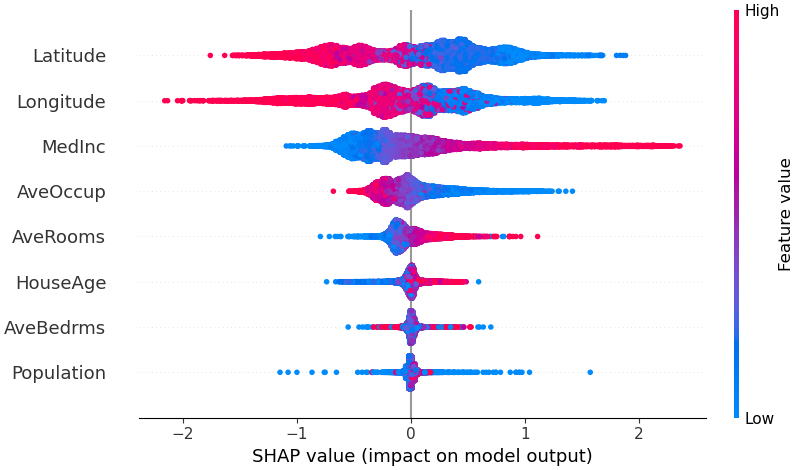
Wir können auch einfach den mittleren Absolutwert der SHAP-Werte für jedes Feature nehmen, um ein Standard-Balkendiagramm zu erhalten (erzeugt gestapelte Balken für Mehrklassenausgaben):
shap . plots . bar ( shap_values )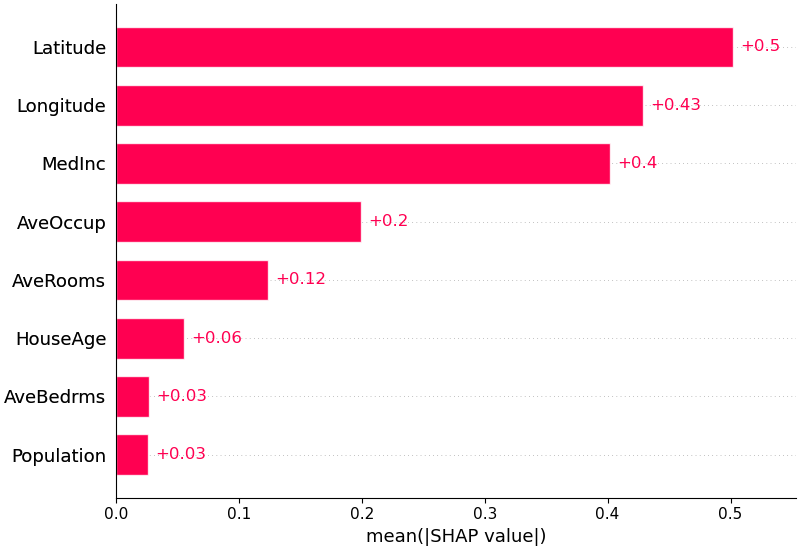
SHAP bietet spezielle Unterstützung für natürliche Sprachmodelle wie die in der Hugging Face-Transformer-Bibliothek. Indem wir den traditionellen Shapley-Werten Koalitionsregeln hinzufügen, können wir Spiele entwickeln, die große moderne NLP-Modelle mit sehr wenigen Funktionsauswertungen erklären. Die Nutzung dieser Funktionalität ist so einfach wie die Übergabe einer unterstützten Transformer-Pipeline an SHAP:
import transformers
import shap
# load a transformers pipeline model
model = transformers . pipeline ( 'sentiment-analysis' , return_all_scores = True )
# explain the model on two sample inputs
explainer = shap . Explainer ( model )
shap_values = explainer ([ "What a great movie! ...if you have no taste." ])
# visualize the first prediction's explanation for the POSITIVE output class
shap . plots . text ( shap_values [ 0 , :, "POSITIVE" ])Deep SHAP ist ein Hochgeschwindigkeits-Approximationsalgorithmus für SHAP-Werte in Deep-Learning-Modellen, der auf einer Verbindung mit DeepLIFT aufbaut, die im SHAP NIPS-Papier beschrieben wird. Die Implementierung hier unterscheidet sich vom ursprünglichen DeepLIFT durch die Verwendung einer Verteilung von Hintergrundproben anstelle eines einzelnen Referenzwerts und die Verwendung von Shapley-Gleichungen zur Linearisierung von Komponenten wie Max, Softmax, Produkten, Divisionen usw. Beachten Sie, dass einige dieser Verbesserungen ebenfalls vorgenommen wurden seitdem in DeepLIFT integriert. TensorFlow-Modelle und Keras-Modelle, die das TensorFlow-Backend verwenden, werden unterstützt (es gibt auch vorläufige Unterstützung für PyTorch):
# ...include code from https://github.com/keras-team/keras/blob/master/examples/demo_mnist_convnet.py
import shap
import numpy as np
# select a set of background examples to take an expectation over
background = x_train [ np . random . choice ( x_train . shape [ 0 ], 100 , replace = False )]
# explain predictions of the model on four images
e = shap . DeepExplainer ( model , background )
# ...or pass tensors directly
# e = shap.DeepExplainer((model.layers[0].input, model.layers[-1].output), background)
shap_values = e . shap_values ( x_test [ 1 : 5 ])
# plot the feature attributions
shap . image_plot ( shap_values , - x_test [ 1 : 5 ])Die obige Darstellung erläutert zehn Ausgaben (Ziffern 0–9) für vier verschiedene Bilder. Rote Pixel erhöhen die Ausgabe des Modells, während blaue Pixel die Ausgabe verringern. Die Eingabebilder werden auf der linken Seite und als nahezu transparente Graustufenhintergründe hinter den einzelnen Erläuterungen angezeigt. Die Summe der SHAP-Werte entspricht der Differenz zwischen der erwarteten Modellausgabe (gemittelt über den Hintergrunddatensatz) und der aktuellen Modellausgabe. Beachten Sie, dass für das „Null“-Bild die leere Mitte wichtig ist, während für das „Vier“-Bild das Fehlen einer Verbindung oben zu einer Vier statt einer Neun führt.
Erwartete Farbverläufe kombinieren Ideen aus integrierten Farbverläufen, SHAP und SmoothGrad in einer einzigen Erwartungswertgleichung. Dies ermöglicht die Verwendung eines gesamten Datensatzes als Hintergrundverteilung (im Gegensatz zu einem einzelnen Referenzwert) und eine lokale Glättung. Wenn wir das Modell mit einer linearen Funktion zwischen jeder Hintergrunddatenprobe und der zu erklärenden aktuellen Eingabe annähern und davon ausgehen, dass die Eingabemerkmale unabhängig sind, berechnen die erwarteten Gradienten ungefähre SHAP-Werte. Im folgenden Beispiel haben wir erklärt, wie sich die 7. Zwischenschicht des VGG16 ImageNet-Modells auf die Ausgabewahrscheinlichkeiten auswirkt.
from keras . applications . vgg16 import VGG16
from keras . applications . vgg16 import preprocess_input
import keras . backend as K
import numpy as np
import json
import shap
# load pre-trained model and choose two images to explain
model = VGG16 ( weights = 'imagenet' , include_top = True )
X , y = shap . datasets . imagenet50 ()
to_explain = X [[ 39 , 41 ]]
# load the ImageNet class names
url = "https://s3.amazonaws.com/deep-learning-models/image-models/imagenet_class_index.json"
fname = shap . datasets . cache ( url )
with open ( fname ) as f :
class_names = json . load ( f )
# explain how the input to the 7th layer of the model explains the top two classes
def map2layer ( x , layer ):
feed_dict = dict ( zip ([ model . layers [ 0 ]. input ], [ preprocess_input ( x . copy ())]))
return K . get_session (). run ( model . layers [ layer ]. input , feed_dict )
e = shap . GradientExplainer (
( model . layers [ 7 ]. input , model . layers [ - 1 ]. output ),
map2layer ( X , 7 ),
local_smoothing = 0 # std dev of smoothing noise
)
shap_values , indexes = e . shap_values ( map2layer ( to_explain , 7 ), ranked_outputs = 2 )
# get the names for the classes
index_names = np . vectorize ( lambda x : class_names [ str ( x )][ 1 ])( indexes )
# plot the explanations
shap . image_plot ( shap_values , to_explain , index_names ) Vorhersagen für zwei Eingabebilder werden in der obigen Darstellung erläutert. Rote Pixel stellen positive SHAP-Werte dar, die die Wahrscheinlichkeit der Klasse erhöhen, während blaue Pixel negative SHAP-Werte darstellen, die die Wahrscheinlichkeit der Klasse verringern. Durch die Verwendung von ranked_outputs=2 erklären wir nur die beiden wahrscheinlichsten Klassen für jede Eingabe (dies erspart uns die Erklärung aller 1.000 Klassen).
Kernel SHAP verwendet eine speziell gewichtete lokale lineare Regression, um SHAP-Werte für jedes Modell zu schätzen. Nachfolgend finden Sie ein einfaches Beispiel zur Erläuterung einer SVM mit mehreren Klassen für den klassischen Iris-Datensatz.
import sklearn
import shap
from sklearn . model_selection import train_test_split
# print the JS visualization code to the notebook
shap . initjs ()
# train a SVM classifier
X_train , X_test , Y_train , Y_test = train_test_split ( * shap . datasets . iris (), test_size = 0.2 , random_state = 0 )
svm = sklearn . svm . SVC ( kernel = 'rbf' , probability = True )
svm . fit ( X_train , Y_train )
# use Kernel SHAP to explain test set predictions
explainer = shap . KernelExplainer ( svm . predict_proba , X_train , link = "logit" )
shap_values = explainer . shap_values ( X_test , nsamples = 100 )
# plot the SHAP values for the Setosa output of the first instance
shap . force_plot ( explainer . expected_value [ 0 ], shap_values [ 0 ][ 0 ,:], X_test . iloc [ 0 ,:], link = "logit" )Die obige Erklärung zeigt vier Funktionen, die jeweils dazu beitragen, die Modellausgabe vom Basiswert (der durchschnittlichen Modellausgabe über den von uns übergebenen Trainingsdatensatz) in Richtung Null zu verschieben. Wenn es Funktionen gäbe, die die Klassenbezeichnung nach oben verschieben würden, würden diese in Rot angezeigt.
Wenn wir viele Erklärungen wie die oben gezeigte nehmen, sie um 90 Grad drehen und sie dann horizontal stapeln, können wir Erklärungen für einen gesamten Datensatz sehen. Genau das machen wir im Folgenden für alle Beispiele im Iris-Testset:
# plot the SHAP values for the Setosa output of all instances
shap . force_plot ( explainer . expected_value [ 0 ], shap_values [ 0 ], X_test , link = "logit" ) SHAP-Interaktionswerte sind eine Verallgemeinerung von SHAP-Werten auf Interaktionen höherer Ordnung. Eine schnelle exakte Berechnung paarweiser Interaktionen wird für Baummodelle mit shap.TreeExplainer(model).shap_interaction_values(X) implementiert. Dadurch wird für jede Vorhersage eine Matrix zurückgegeben, bei der die Haupteffekte auf der Diagonale und die Interaktionseffekte außerhalb der Diagonale liegen. Diese Werte offenbaren häufig interessante versteckte Zusammenhänge, beispielsweise wie das erhöhte Sterberisiko bei Männern im Alter von 60 Jahren seinen Höhepunkt erreicht (Einzelheiten finden Sie im NHANES-Notizbuch):
Die folgenden Notebooks veranschaulichen verschiedene Anwendungsfälle für SHAP. Schauen Sie im Notebook-Verzeichnis des Repositorys nach, wenn Sie selbst versuchen möchten, mit den Original-Notebooks zu spielen.
Eine Implementierung von Tree SHAP, einem schnellen und exakten Algorithmus zur Berechnung von SHAP-Werten für Bäume und Baumensembles.
NHANES-Überlebensmodell mit XGBoost- und SHAP-Interaktionswerten – Dieses Notizbuch zeigt anhand von Mortalitätsdaten aus 20 Jahren Follow-up, wie man XGBoost und shap verwendet, um komplexe Risikofaktorbeziehungen aufzudecken.
Klassifizierung des Volkszählungseinkommens mit LightGBM – Unter Verwendung des standardmäßigen Einkommensdatensatzes für die Volkszählung von Erwachsenen trainiert dieses Notebook ein Gradientenverstärkungsbaummodell mit LightGBM und erklärt dann Vorhersagen mithilfe von shap .
League of Legends-Siegvorhersage mit XGBoost – Anhand eines Kaggle-Datensatzes von 180.000 Ranglistenspielen aus League of Legends trainieren und erklären wir mit XGBoost ein Gradient-Boosting-Tree-Modell, um vorherzusagen, ob ein Spieler sein Match gewinnen wird.
Eine Implementierung von Deep SHAP, einem schnelleren (aber nur ungefähren) Algorithmus zur Berechnung von SHAP-Werten für Deep-Learning-Modelle, der auf Verbindungen zwischen SHAP und dem DeepLIFT-Algorithmus basiert.
MNIST-Ziffernklassifizierung mit Keras – Mithilfe des MNIST-Handschrifterkennungsdatensatzes trainiert dieses Notebook ein neuronales Netzwerk mit Keras und erklärt dann Vorhersagen mithilfe von shap .
Keras LSTM für die IMDB-Sentiment-Klassifizierung – Dieses Notebook trainiert ein LSTM mit Keras anhand des IMDB-Datensatzes zur Text-Sentiment-Analyse und erklärt dann Vorhersagen mithilfe von shap .
Eine Implementierung erwarteter Gradienten zur Annäherung an SHAP-Werte für Deep-Learning-Modelle. Es basiert auf Verbindungen zwischen SHAP und dem Integrated Gradients-Algorithmus. GradientExplainer ist langsamer als DeepExplainer und geht von anderen Näherungsannahmen aus.
Für ein lineares Modell mit unabhängigen Merkmalen können wir die genauen SHAP-Werte analytisch berechnen. Wir können auch die Merkmalskorrelation berücksichtigen, wenn wir bereit sind, die Merkmalskovarianzmatrix zu schätzen. LinearExplainer unterstützt beide Optionen.
Eine Implementierung von Kernel SHAP, einer modellunabhängigen Methode zur Schätzung von SHAP-Werten für jedes Modell. Da keine Annahmen über den Modelltyp getroffen werden, ist KernelExplainer langsamer als die anderen modelltypspezifischen Algorithmen.
Klassifizierung des Volkszählungseinkommens mit scikit-learn – Unter Verwendung des standardmäßigen Einkommensdatensatzes für die Volkszählung von Erwachsenen trainiert dieses Notebook mithilfe von scikit-learn einen Klassifikator für k-nächste Nachbarn und erklärt dann Vorhersagen mithilfe von shap .
ImageNet VGG16-Modell mit Keras – Erklären Sie die Vorhersagen des klassischen VGG16-Faltungs-Neuronalen Netzwerks für ein Bild. Dies funktioniert durch die Anwendung der modellunabhängigen Kernel-SHAP-Methode auf ein Superpixel-segmentiertes Bild.
Irisklassifizierung – Eine grundlegende Demonstration unter Verwendung des Datensatzes beliebter Irisarten. Es erklärt Vorhersagen aus sechs verschiedenen Modellen in scikit-learn mithilfe von shap .
Diese Notizbücher demonstrieren umfassend die Verwendung bestimmter Funktionen und Objekte.
shap.decision_plot und shap.multioutput_decision_plot
shap.dependence_plot
LIME: Ribeiro, Marco Tulio, Sameer Singh und Carlos Guestrin. „Warum sollte ich Ihnen vertrauen?: Die Vorhersagen eines Klassifikators erklären.“ Tagungsband der 22. ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. ACM, 2016.
Shapley-Stichprobenwerte: Strumbelj, Erik und Igor Kononenko. „Erklärung von Vorhersagemodellen und einzelnen Vorhersagen mit Feature-Beiträgen.“ Wissens- und Informationssysteme 41.3 (2014): 647-665.
DeepLIFT: Shrikumar, Avanti, Peyton Greenside und Anshul Kundaje. „Erlernen wichtiger Funktionen durch die Verbreitung von Aktivierungsunterschieden.“ arXiv-Vorabdruck arXiv:1704.02685 (2017).
QII: Datta, Anupam, Shayak Sen und Yair Zick. „Algorithmische Transparenz durch quantitative Inputbeeinflussung: Theorie und Experimente mit lernenden Systemen.“ Sicherheit und Datenschutz (SP), IEEE-Symposium 2016 zum Thema. IEEE, 2016.
Schichtweise Relevanzausbreitung: Bach, Sebastian et al. „Über pixelweise Erklärungen für nichtlineare Klassifikatorentscheidungen durch schichtweise Relevanzausbreitung.“ PloS one 10.7 (2015): e0130140.
Shapley-Regressionswerte: Lipovetsky, Stan und Michael Conklin. „Analyse der Regression im spieltheoretischen Ansatz.“ Angewandte stochastische Modelle in Wirtschaft und Industrie 17.4 (2001): 319-330.
Bauminterpret: Saabas, Ando. Interpretation zufälliger Wälder. http://blog.datadive.net/interpreting-random-forests/
Die in diesem Paket verwendeten Algorithmen und Visualisierungen stammen hauptsächlich aus Forschungen in Su-In Lees Labor an der University of Washington und Microsoft Research. Wenn Sie SHAP in Ihrer Forschung verwenden, würden wir uns über eine Zitierung der entsprechenden Arbeit(en) freuen:
force_plot -Visualisierungen und medizinische Anwendungen können Sie unser Nature Biomedical Engineering-Papier (bibtex; freier Zugang) lesen/zitieren.