Mit greta.gam können Sie die glatteren Funktionen und Formelsyntax von mgcv verwenden, um glatte Begriffe für die Verwendung in einem Greta-Modell zu definieren. Anschließend können Sie Ihre eigene Wahrscheinlichkeit für die Vervollständigung des Modells definieren und es von MCMC anpassen.
Dies ist in Arbeit!
Hier ist ein einfaches Beispiel aus der mgcv ?gam Hilfedatei:
In mgcv :
library( mgcv )
# > Loading required package: nlme
# > This is mgcv 1.9-1. For overview type 'help("mgcv-package")'.
set.seed( 2 )
# simulate some data...
dat <- gamSim( 1 , n = 400 , dist = " normal " , scale = 0.3 )
# > Gu & Wahba 4 term additive model
# fit a model using gam()
b <- gam( y ~ s( x2 ), data = dat ) Jetzt passt das gleiche Modell in greta :
library( greta.gam )
# > Loading required package: greta
# >
# > Attaching package: 'greta'
# > The following objects are masked from 'package:stats':
# >
# > binomial, cov2cor, poisson
# > The following objects are masked from 'package:base':
# >
# > %*%, apply, backsolve, beta, chol2inv, colMeans, colSums, diag,
# > eigen, forwardsolve, gamma, identity, rowMeans, rowSums, sweep,
# > tapply
set.seed( 2024 - 02 - 09 )
# setup the linear predictor for the smooth
z <- smooths( ~ s( x2 ), data = dat )
# > ℹ Initialising python and checking dependencies, this may take a moment.
# > ✔ Initialising python and checking dependencies ... done!
# set the distribution of the response
distribution( dat $ y ) <- normal( z , 1 )
# make some prediction data
pred_dat <- data.frame ( x2 = seq( 0 , 1 , length.out = 100 ))
# z_pred stores the predictions
z_pred <- evaluate_smooths( z , newdata = pred_dat )
# build model
m <- model( z_pred )
# draw from the posterior
draws <- mcmc( m , n_samples = 200 )
# > running 4 chains simultaneously on up to 8 CPU cores
#> warmup 0/1000 | eta: ?s warmup == 50/1000 | eta: 30s warmup ==== 100/1000 | eta: 17s warmup ====== 150/1000 | eta: 12s warmup ======== 200/1000 | eta: 10s warmup ========== 250/1000 | eta: 8s warmup =========== 300/1000 | eta: 7s warmup ============= 350/1000 | eta: 6s warmup =============== 400/1000 | eta: 5s warmup ================= 450/1000 | eta: 5s warmup =================== 500/1000 | eta: 4s warmup ===================== 550/1000 | eta: 4s warmup ======================= 600/1000 | eta: 3s warmup ========================= 650/1000 | eta: 3s warmup =========================== 700/1000 | eta: 2s warmup ============================ 750/1000 | eta: 2s warmup ============================== 800/1000 | eta: 1s warmup ================================ 850/1000 | eta: 1s warmup ================================== 900/1000 | eta: 1s warmup ==================================== 950/1000 | eta: 0s warmup ====================================== 1000/1000 | eta: 0s
# > sampling 0/200 | eta: ?s sampling ========== 50/200 | eta: 1s sampling =================== 100/200 | eta: 0s sampling ============================ 150/200 | eta: 0s sampling ====================================== 200/200 | eta: 0s
# plot the mgcv fit
plot( b , scheme = 1 , shift = coef( b )[ 1 ])
# add in a line for each posterior sample
apply( draws [[ 1 ]], 1 , lines , x = pred_dat $ x2 , col = " blue " )
# > NULL
# plot the data
points( dat $ x2 , dat $ y , pch = 19 , cex = 0.2 )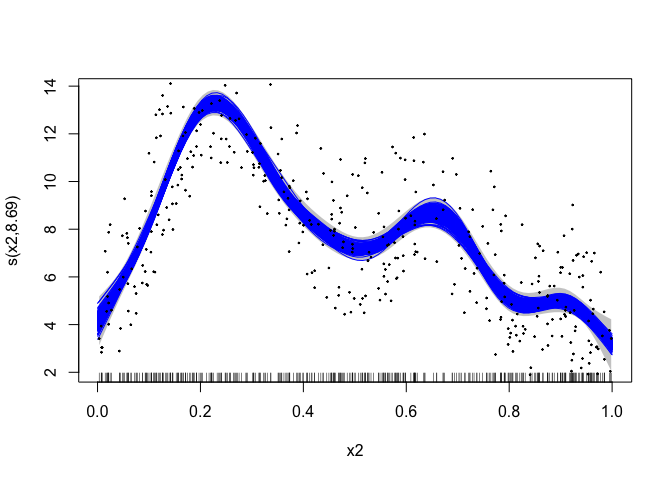
greta.gam nutzt ein paar Tricks aus der jagam -Routine (Wood, 2016) in mgcv um die Dinge zum Laufen zu bringen. Hier einige kurze Details für diejenigen, die sich für die internen Abläufe interessieren …
GAMs sind Modelle mit bayesianischer Interpretation (auch wenn sie mit „frequentistischen“ Methoden angepasst werden). Man kann sich die glattere Strafmatrix als Prior-Präzisionsmatrix in einem Bayes'schen Zufallseffektmodell vorstellen. Designmatrizen werden genau wie im Fall des Frequentismus konstruiert. Weitere Hintergrundinformationen hierzu finden Sie in Miller (2021).
Bei der bayesianischen Interpretation des GAM besteht eine leichte Schwierigkeit darin, dass die Prioren in ihrer naiven Form als Nullraum der Strafe ungeeignet sind (im 1D-Fall normalerweise der lineare Term). Um richtige Priors zu erhalten, können wir einen der in Marra & Wood (2011) verwendeten „Tricks“ anwenden – nämlich die Teile der Strafe, die zum falschen Prior führen, irgendwie zu bestrafen. Wir nutzen die von jagam bereitgestellte Option und erstellen eine zusätzliche Strafmatrix für diese Terme (aus einer Eigenzerlegung der Strafmatrix; siehe Marra & Wood, 2011).
Marra, G und Wood, SN (2011) Praktische Variablenauswahl für verallgemeinerte additive Modelle. Computerstatistik und Datenanalyse, 55, 2372–2387.
Miller DL (2021). Bayesianische Ansichten zur verallgemeinerten additiven Modellierung. arXiv.
Wood, SN (2016) Just Another Gibbs Additive Modeler: Interfacing JAGS and mgcv. Journal of Statistical Software 75, Nr. 7