Repository mit Daten und Code für das Guar-Genotypisierungs- und GWAS-Projekt ( Cyamopsis tetragonoloba (L.) Taub.). Eine kurze Beschreibung des Repository-Inhalts:
Das Skript make_consensus_vcf.py wurde verwendet, um den endgültigen Variantendatensatz mit rohen GATK-HC-, NGSEP- und TASSEL 5-Variantenaufrufen zu generieren. Das Skript make_snp_stats.py wurde mit denselben Eingaben verwendet, um die Qualitätsstatistiken der Variantenaufrufe zu untersuchen.
Das Notizbuch callset_refinement_filtering.ipynb wurde verwendet, um die Variantenqualitätskontrolle durchzuführen und den endgültigen gefilterten Datensatz gängiger Varianten zu erstellen.
Mit dem Notebook genetic_analysis.ipynb wurden die meisten genetischen Analysen durchgeführt, darunter PCA, Kopplungsungleichgewichtsanalyse und verallgemeinerte lineare modellbasierte Assoziationsanalyse.
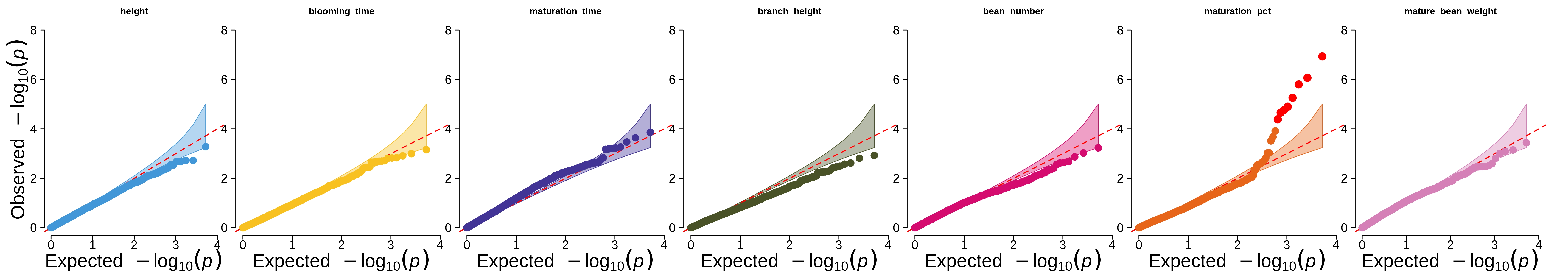
Das Skript prepare_farmcpu.py wurde zum Formatieren der Eingabedaten für das FarmCPU-Tool verwendet (das QQ-Diagramm der resultierenden p-Werte ist im Artikel und unten dargestellt).
Die Datei admix_file_create.py wurde verwendet, um Genotypen für die ADMIXTURE-Analyse vorzuverarbeiten (ein Teil davon wurde mit Hail in der Datei genetic_analysis.ipynb durchgeführt).
Das Skript guar_stat.R wurde verwendet, um eine statistische Analyse der endgültigen Daten durchzuführen, eine genomweite Assoziationsanalyse mit dem FarmCPU-Tool durchzuführen und die Hauptzahlen darzustellen
Das Skript parse_vcf_fcpu.py wurde verwendet, um die endgültige SNP-Genotyptabelle zu formatieren, die als Datei genotypes.xlsx in diesem Repository verfügbar ist.