Dieses Paket stellt trainierte U-Net-Modelle für die Lungensegmentierung bereit. Derzeit sind vier Modelle verfügbar:
U-net (R231): Dieses Modell wurde anhand eines großen und vielfältigen Datensatzes trainiert, der ein breites Spektrum visueller Variabilität abdeckt. Das Modell führt eine Segmentierung in einzelnen Schnitten durch und extrahiert die rechte und linke Lunge separat, einschließlich Lufttaschen, Tumoren und Ergüssen. Die Luftröhre wird nicht in die Lungensegmentierung einbezogen. https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes): Dieses Modell wurde auf einer Teilmenge des LTRC-Datensatzes trainiert. Das Modell führt eine Segmentierung einzelner Lungenlappen durch, liefert jedoch eine begrenzte Leistung, wenn dichte Pathologien vorhanden sind oder wenn Risse nicht in jeder Schicht sichtbar sind.
U-net(LTRCLobes_R231): Dadurch werden das R231- und LTRCLobes-Modell ausgeführt und die Ergebnisse zusammengeführt. Falsch negative Ergebnisse von LTRCLobes werden durch R231-Vorhersagen aufgefüllt und einem Nachbarlabel zugeordnet. Falsch positive Ergebnisse von LTRCLobes werden entfernt. Der Fusionsprozess ist rechenintensiv und kann je nach Daten und Ergebnissen bis zu mehreren Minuten pro Band dauern.
U-net(R231CovidWeb)
Beispiele der beiden angewandten Modelle . Links: U-net (R231), unterscheidet zwischen linker und rechter Lunge und umfasst sehr dichte Bereiche wie Ergüsse (dritte Reihe), Tumor oder schwere Fibrose (vierte Reihe). Rechts: U-net(LTRLobes), unterscheidet zwischen Lungenlappen, schließt aber keine sehr dichten Bereiche ein. LTRCLobes_R231 führt LTRCLobes- und R231-Ergebnisse zusammen. R231CovidWeb wird mit zusätzlichen COVID-19-Daten trainiert.
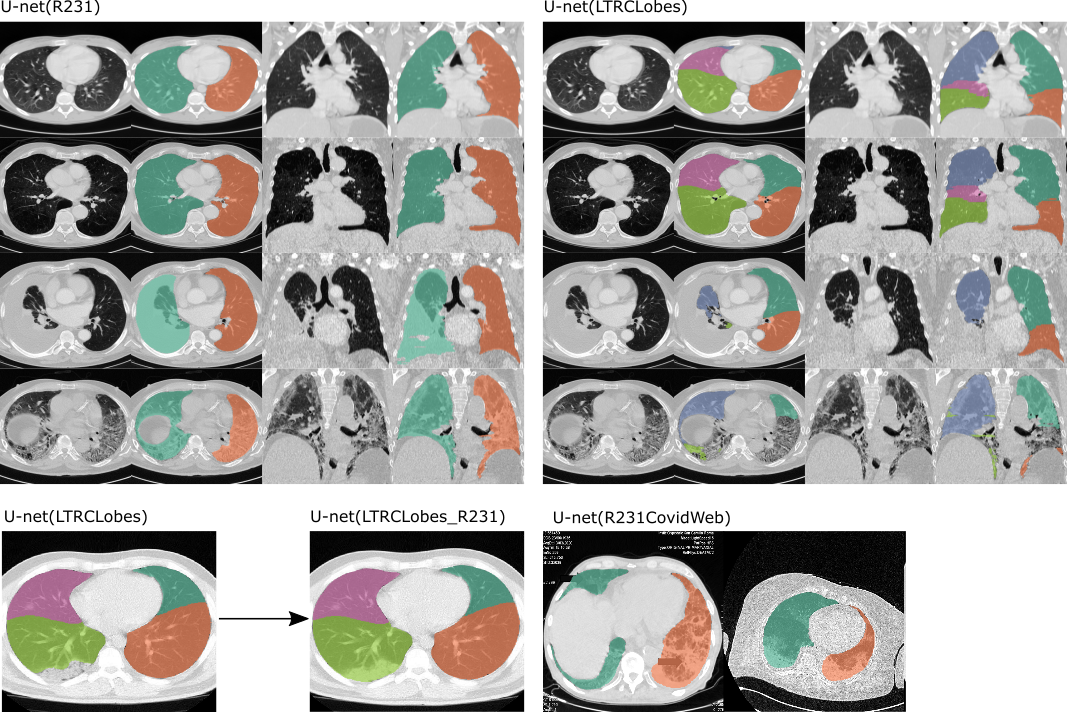
Semantik der Ausgabe :
Zwei Etikettenmodelle (Links-Rechts):
1 = Rechte Lunge
2 = Linke Lunge
Fünf Etikettenmodelle (Lungenlappen):
1 = Linker Oberlappen
2 = Linker Unterlappen
3 = Rechter Oberlappen
4 = Rechter Mittellappen
5 = Rechter Unterlappen
Weitere spannende Forschungsergebnisse zu Lungen-CT-Daten finden Sie auf der Website unserer Forschungsgruppe: https://www.cir.meduniwien.ac.at/research/lung/
Wenn Sie diesen Code oder eines der trainierten Modelle in Ihrer Arbeit verwenden, lesen Sie bitte Folgendes:
Hofmanninger, J., Prayer, F., Pan, J. et al. Die automatische Lungensegmentierung in der Routinebildgebung ist in erster Linie ein Problem der Datenvielfalt und kein methodisches Problem. Eur Radiol Exp 4, 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
Dieses Papier enthält eine detaillierte Beschreibung des verwendeten Datensatzes, eine gründliche Bewertung des U-net(R231)-Modells und einen Vergleich mit Referenzmethoden.
pip install lungmask
oder
pip install git+https://github.com/JoHof/lungmask
Unter Windows kann es je nach Setup erforderlich sein, Torch vorher zu installieren: https://pytorch.org
Die Laufzeit zwischen reiner CPU- und GPU-unterstützter Inferenz variiert stark. Mit der GPU dauert die Verarbeitung eines Volumes nur wenige Sekunden, mit der reinen CPU dauert es mehrere Minuten. Um die GPU nutzen zu können, stellen Sie sicher, dass Ihre Brennerinstallation CUDA-Unterstützung bietet. Im Falle von cuda-out-of-memory-Fehlern reduzieren Sie die Batchgröße mit dem optionalen Argument --batchsize 1 auf 1
lungmask INPUT OUTPUT
Wenn INPUT auf eine Datei verweist, wird die Datei verarbeitet. Wenn INPUT auf ein Verzeichnis zeigt, wird das Verzeichnis nach DICOM-Serien durchsucht. Das größte gefundene Volumen (in Bezug auf die Anzahl der Voxel) wird zur Berechnung der Lungenmaske verwendet. OUTPUT ist der Name der Ausgabedatei. Alle ITK-Formate werden unterstützt.
Wählen Sie ein Modell:
Als Standard wird das U-Net (R231) verwendet. Sie können jedoch ein alternatives Modell angeben, z. B. LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
Geben Sie für zusätzliche Optionen Folgendes ein:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image muss ein SimpleITK-Objekt sein.
Laden Sie ein alternatives Modell wie folgt:
inferer = LMInferer(modelname="R231CovidWeb")
Um die Modellfusionsfunktion für (z. B. LTRCLobes_R231) zu verwenden, verwenden Sie:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
Ab Version 0.2.9 werden Numpy-Arrays als Eingabevolumes unterstützt. In diesem Modus wird davon ausgegangen, dass das Eingabe-Numpy-Array für jede Achse das folgende Format hat:
Das Modell funktioniert nur mit vollständigen Slices. Der zu verarbeitende Schnitt muss die gesamte Lunge zeigen und die Lunge muss von Gewebe umgeben sein, um segmentiert zu werden. Allerdings ist das Modell bei beschnittenem Sichtfeld recht stabil, solange die Lunge von Gewebe umgeben ist.
lungmask INPUT OUTPUT --modelname R231CovidWeb
Das reguläre U-net(R231)-Modell eignet sich sehr gut für COVID-19-CT-Scans. Sammlungen von Schnitten und Fallberichten aus dem Internet werden jedoch häufig zugeschnitten, mit Anmerkungen versehen oder in regulären Bildformaten kodiert, sodass die ursprünglichen Werte der Hounsfield-Einheit (HU) nur geschätzt werden können. Die Trainingsdaten des U-net(R231CovidWeb)-Modells wurden mit COVID-19-Schnitten ergänzt, die von regulären Bildformaten auf HU zurückgebildet wurden. Die Daten wurden von MedSeg (http://medicalsegmentation.com/covid19/) gesammelt und aufbereitet. Während das reguläre U-net (R231) für diese Bilder sehr gute Ergebnisse zeigte, kann es Fälle geben, in denen dieses Modell leicht verbesserte Segmentierungen liefert. Beachten Sie, dass Sie Bilder wieder HU zuordnen müssen, wenn Sie Bilder aus dem Internet verwenden. Dieser Blogbeitrag beschreibt, wie Sie das tun können. Alternativ können Sie das Tag --noHU setzen. 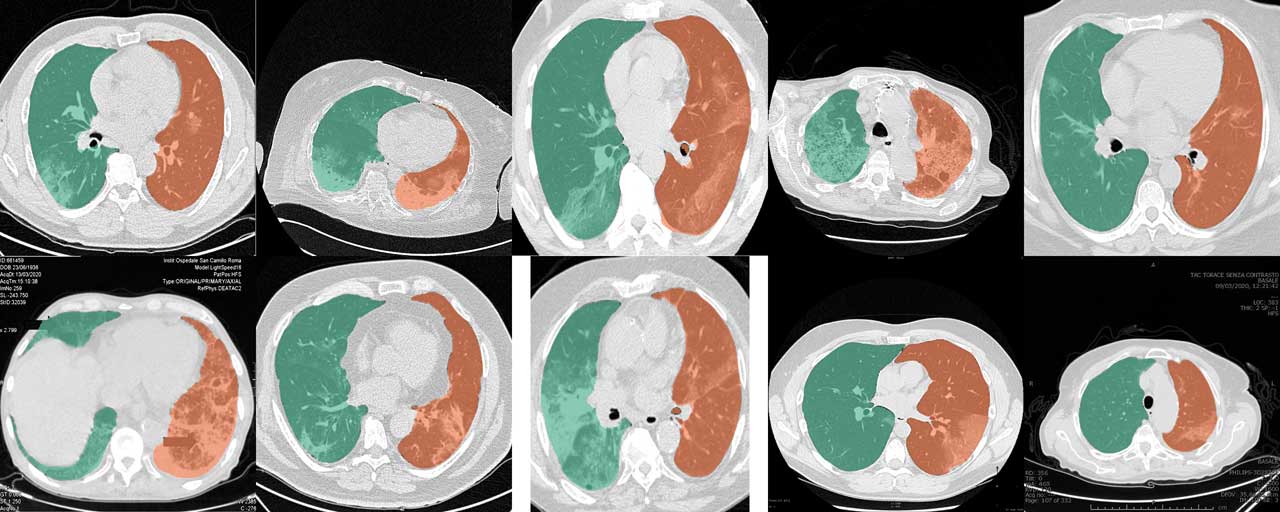
Diese Funktion ist nur in den Versionen zwischen 0.2.5 und 0.2.14 verfügbar. Ab Version 0.2.5 werden diese Bilder unterstützt. Verwenden Sie das Tag --noHU , wenn Sie Bilder verarbeiten, die nicht in HU kodiert sind. Bedenken Sie, dass die Modelle anhand korrekter, in HU kodierter CT-Scans trainiert wurden. Die Ergebnisse bei beschnittenen, kommentierten Bildern mit sehr hoher und sehr geringer Intensitätsverschiebung sind möglicherweise nicht sehr zuverlässig. Bei Verwendung des Tags --noHU können nur einzelne Slices verarbeitet werden.