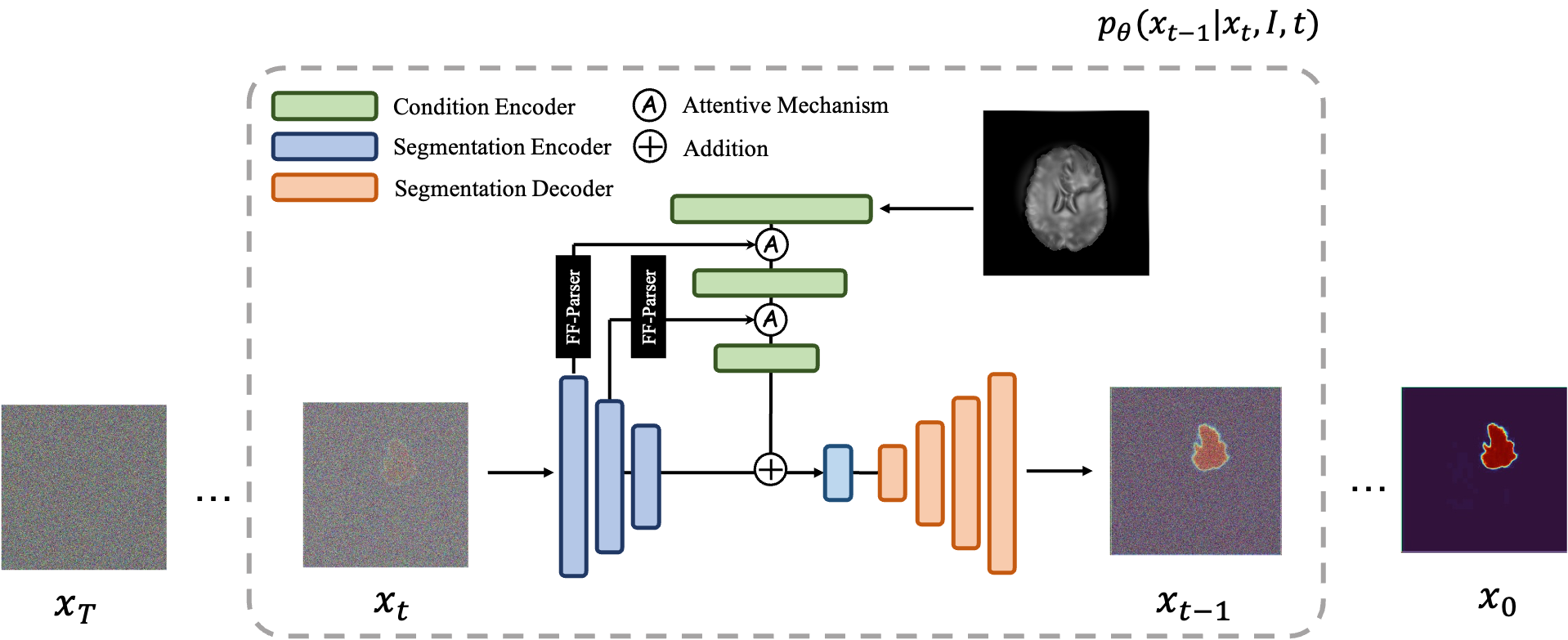
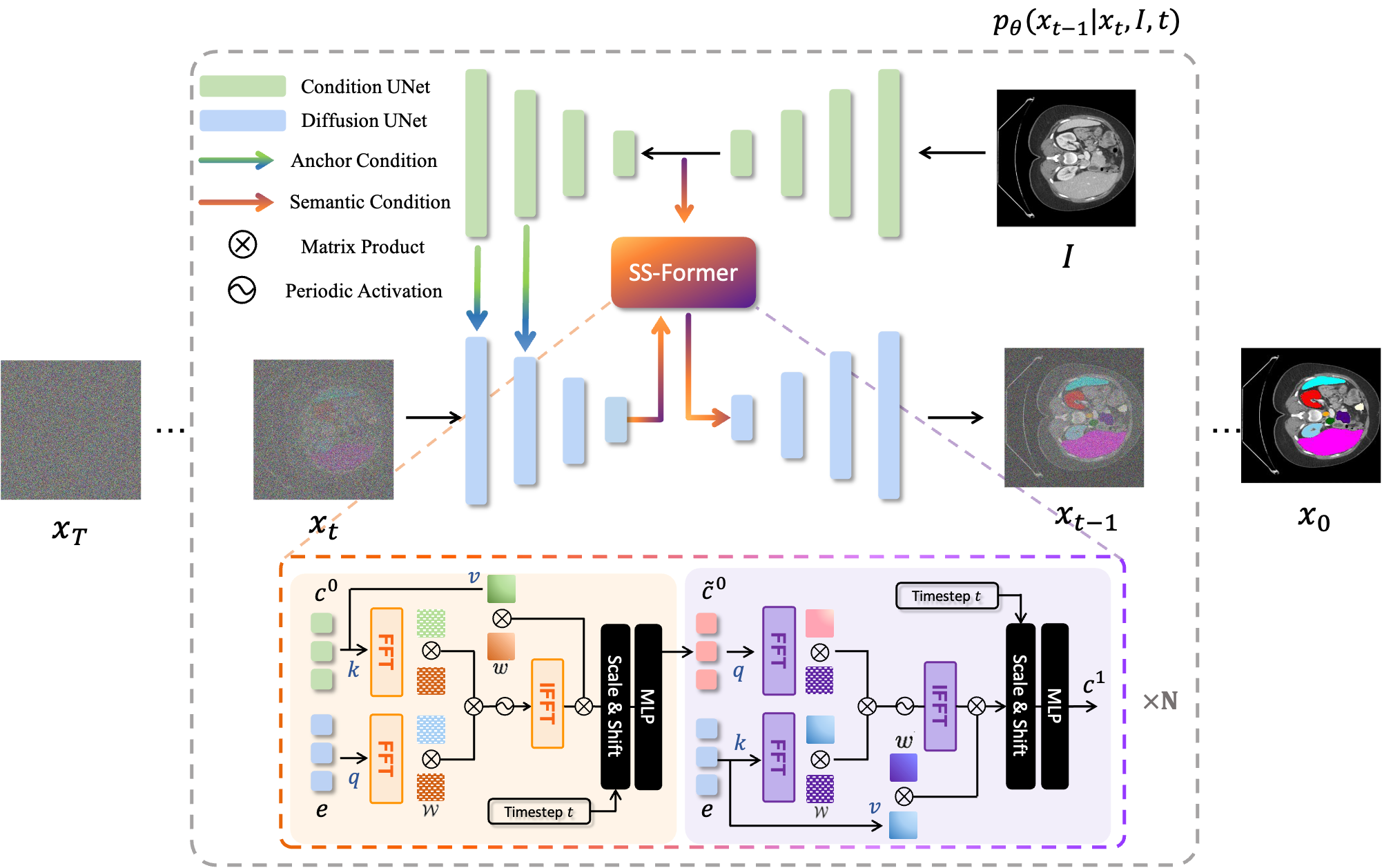
MedSegDiff ist ein auf dem Diffusion Probabilistic Model (DPM) basierendes Framework für die medizinische Bildsegmentierung. Der Algorithmus wird in unserem Artikel MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model und MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer näher erläutert.
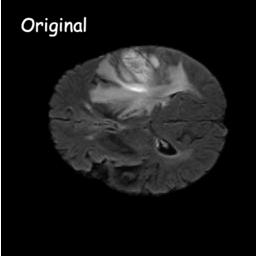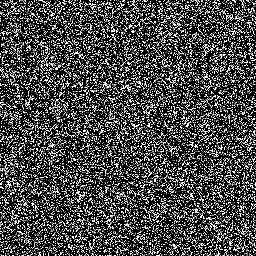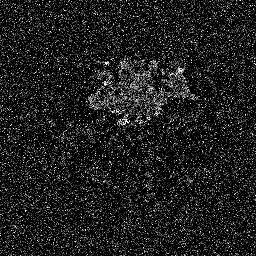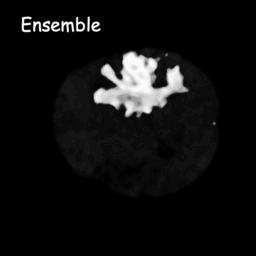 Diffusionsmodelle funktionieren, indem sie Trainingsdaten durch sukzessive Hinzufügung von Gauß-Rauschen zerstören und dann lernen, die Daten durch Umkehrung dieses Rauschprozesses wiederherzustellen. Nach dem Training können wir das Diffusionsmodell verwenden, um Daten zu generieren, indem wir einfach zufällig abgetastetes Rauschen durch den erlernten Entrauschungsprozess leiten. In diesem Projekt erweitern wir diese Idee auf die Segmentierung medizinischer Bilder. Wir verwenden das Originalbild als Bedingung und generieren aus zufälligen Geräuschen mehrere Segmentierungskarten und führen dann eine Zusammenfügung durch, um das Endergebnis zu erhalten. Dieser Ansatz erfasst die Unsicherheit in medizinischen Bildern und übertrifft frühere Methoden bei mehreren Benchmarks.
Diffusionsmodelle funktionieren, indem sie Trainingsdaten durch sukzessive Hinzufügung von Gauß-Rauschen zerstören und dann lernen, die Daten durch Umkehrung dieses Rauschprozesses wiederherzustellen. Nach dem Training können wir das Diffusionsmodell verwenden, um Daten zu generieren, indem wir einfach zufällig abgetastetes Rauschen durch den erlernten Entrauschungsprozess leiten. In diesem Projekt erweitern wir diese Idee auf die Segmentierung medizinischer Bilder. Wir verwenden das Originalbild als Bedingung und generieren aus zufälligen Geräuschen mehrere Segmentierungskarten und führen dann eine Zusammenfügung durch, um das Endergebnis zu erhalten. Dieser Ansatz erfasst die Unsicherheit in medizinischen Bildern und übertrifft frühere Methoden bei mehreren Benchmarks.
 |  |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True setzen.python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
Führen Sie zum Training Folgendes aus: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Führen Sie zum Sampling Folgendes aus: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
Führen Sie zur Auswertung python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
Standardmäßig werden die Proben unter ./results/ gespeichert.
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
Führen Sie zum Training Folgendes aus: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Führen Sie zum Sampling Folgendes aus: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
Es ist einfach, MedSegDiff für die anderen Datensätze auszuführen. Schreiben Sie einfach eine weitere Datenladedatei nach ./guided_diffusion/isicloader.py oder ./guided_diffusion/bratsloader.py . Willkommen bei offenen Problemen, wenn Sie auf ein Problem stoßen. Wir würden uns freuen, wenn Sie Ihre Datensatzerweiterungen beisteuern könnten. Im Gegensatz zu natürlichen Bildern variieren medizinische Bilder je nach Aufgabenstellung stark. Die Erweiterung der Verallgemeinerung einer Methode erfordert die Anstrengung aller.
Um ein feines Modell zu trainieren, z. B. MedSegDiff-B in der Arbeit, legen Sie die Modellhyperparameter wie folgt fest:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
Diffusionshyperparameter als:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
Um die Probenahme zu beschleunigen:
--diffusion_steps 50 --dpm_solver True
auf mehreren GPUs laufen:
--multi-gpu 0,1,2 (for example)
Hyperparameter trainieren als:
--lr 5e-5 --batch_size 8
und setze --num_ensemble 5 im Sampling.
Das Ausführen von etwa 100.000 Schritten im Training wird in den meisten Datensätzen konvergiert. Beachten Sie, dass sich der Verlust in den meisten späteren Schritten zwar nicht verringert, die Qualität der Ergebnisse jedoch immer noch verbessert wird. Ein solcher Prozess wird auch bei anderen DPM-Anwendungen beobachtet, beispielsweise bei der Bildgenerierung. Ich hoffe, jemand, der schlau ist, kann mir sagen, warum?
Aus Vergleichsgründen werde ich demnächst die Leistung unter kleinerer Batchgröße (geeignet für die Ausführung auf einer 24-GB-GPU) veröffentlichen.
Eine Einstellung, um sein ganzes Potenzial freizusetzen, ist (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
Trainieren Sie es dann mit der Batchgröße --batch_size 64 und probieren Sie es mit der Ensemblenummer --num_ensemble 25 aus.
Willkommen bei MedSegDiff. Wir freuen uns über jede Technik, die die Leistung verbessern oder den Algorithmus beschleunigen kann. Ich schreibe MedSegDiff V2 mit dem Ziel, Nature-Zeitschriften/CVPR-ähnliche Veröffentlichungen zu veröffentlichen. Ich freue mich, die Mitwirkenden als meine Co-Autoren aufzuführen.
Code wurde häufig von openai/improved-diffusion, WuJunde/MrPrism, WuJunde/DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Diffusion-based-Segmentation, hojonathanho/diffusion,guided-diffusion, bigmb/Unit-Segmentation-Pytorch-Nest kopiert -of-Unets, nnUnet, lucidrains/vit-pytorch
Bitte zitieren
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu