Ein wunderbares Tool für maschinelles Lernen, mit dem Sie Modelle trainieren/anpassen, testen und verwenden können, ohne Code schreiben zu müssen
Notiz
Ich arbeite auch an einer GUI-Desktop-App für igel, basierend auf den Anfragen der Leute. Sie finden es unter Igel-UI.
Inhaltsverzeichnis
Das Ziel des Projekts besteht darin, maschinelles Lernen für alle bereitzustellen, sowohl für technische als auch für nichttechnische Benutzer.
Manchmal brauchte ich ein Tool, mit dem ich schnell einen Prototyp für maschinelles Lernen erstellen kann. Egal, ob Sie einen Machbarkeitsnachweis erstellen, einen schnellen Modellentwurf erstellen möchten, um einen Punkt zu beweisen, oder Auto ML verwenden möchten. Ich stecke oft beim Schreiben von Standardcode fest und denke zu viel darüber nach, wo ich anfangen soll. Deshalb habe ich beschlossen, dieses Tool zu erstellen.
igel basiert auf anderen ML-Frameworks. Es bietet eine einfache Möglichkeit, maschinelles Lernen zu nutzen, ohne eine einzige Codezeile schreiben zu müssen. Igel ist hochgradig anpassbar , aber nur, wenn Sie es möchten. Igel zwingt Sie nicht, etwas anzupassen. Neben Standardwerten kann igel auch Auto-ML-Funktionen verwenden, um ein Modell zu ermitteln, das hervorragend mit Ihren Daten funktioniert.
Sie benötigen lediglich eine Yaml- (oder JSON- )Datei, in der Sie beschreiben müssen, was Sie tun möchten. Das ist es!
Igel unterstützt Regression, Klassifizierung und Clustering. Igel unterstützt Auto-ML-Funktionen wie ImageClassification und TextClassification
Igel unterstützt die am häufigsten verwendeten Datensatztypen im Bereich Data Science. Ihr Eingabedatensatz kann beispielsweise eine CSV-, TXT-, Excel-Tabellen-, JSON- oder sogar HTML-Datei sein, die Sie abrufen möchten. Wenn Sie Auto-ML-Funktionen verwenden, können Sie sogar Rohdaten an igel weiterleiten und es wird herausfinden, wie es damit umgeht. Mehr dazu später in den Beispielen.
$ pip install -U igel Von Igel unterstützte Modelle:
+--------------------+----------------------------+-------------------------+
| regression | classification | clustering |
+--------------------+----------------------------+-------------------------+
| LinearRegression | LogisticRegression | KMeans |
| Lasso | Ridge | AffinityPropagation |
| LassoLars | DecisionTree | Birch |
| BayesianRegression | ExtraTree | AgglomerativeClustering |
| HuberRegression | RandomForest | FeatureAgglomeration |
| Ridge | ExtraTrees | DBSCAN |
| PoissonRegression | SVM | MiniBatchKMeans |
| ARDRegression | LinearSVM | SpectralBiclustering |
| TweedieRegression | NuSVM | SpectralCoclustering |
| TheilSenRegression | NearestNeighbor | SpectralClustering |
| GammaRegression | NeuralNetwork | MeanShift |
| RANSACRegression | PassiveAgressiveClassifier | OPTICS |
| DecisionTree | Perceptron | KMedoids |
| ExtraTree | BernoulliRBM | ---- |
| RandomForest | BoltzmannMachine | ---- |
| ExtraTrees | CalibratedClassifier | ---- |
| SVM | Adaboost | ---- |
| LinearSVM | Bagging | ---- |
| NuSVM | GradientBoosting | ---- |
| NearestNeighbor | BernoulliNaiveBayes | ---- |
| NeuralNetwork | CategoricalNaiveBayes | ---- |
| ElasticNet | ComplementNaiveBayes | ---- |
| BernoulliRBM | GaussianNaiveBayes | ---- |
| BoltzmannMachine | MultinomialNaiveBayes | ---- |
| Adaboost | ---- | ---- |
| Bagging | ---- | ---- |
| GradientBoosting | ---- | ---- |
+--------------------+----------------------------+-------------------------+Für Auto-ML:
Der Befehl „help“ ist sehr nützlich, um unterstützte Befehle und entsprechende Argumente/Optionen zu überprüfen
$ igel --helpSie können auch Hilfe zu Unterbefehlen ausführen, zum Beispiel:
$ igel fit --helpIgel ist hochgradig anpassbar. Wenn Sie wissen, was Sie wollen und Ihr Modell manuell konfigurieren möchten, lesen Sie die nächsten Abschnitte, die Ihnen zeigen, wie Sie eine Yaml- oder JSON-Konfigurationsdatei schreiben. Danach müssen Sie igel nur noch mitteilen, was zu tun ist und wo sich Ihre Daten- und Konfigurationsdatei befindet. Hier ist ein Beispiel:
$ igel fit --data_path ' path_to_your_csv_dataset.csv ' --yaml_path ' path_to_your_yaml_file.yaml 'Sie können aber auch die Auto-ML-Funktionen nutzen und igel alles für Sie erledigen lassen. Ein gutes Beispiel hierfür wäre die Bildklassifizierung. Stellen Sie sich vor, Sie haben bereits einen Datensatz mit Rohbildern, der in einem Ordner namens „images“ gespeichert ist
Alles, was Sie tun müssen, ist Folgendes auszuführen:
$ igel auto-train --data_path ' path_to_your_images_folder ' --task ImageClassificationDas ist es! Igel liest die Bilder aus dem Verzeichnis, verarbeitet den Datensatz (Konvertierung in Matrizen, Neuskalierung, Aufteilung usw.) und beginnt mit dem Training/Optimierung eines Modells, das gut mit Ihren Daten funktioniert. Wie Sie sehen, ist das ganz einfach: Sie müssen lediglich den Pfad zu Ihren Daten und die Aufgabe angeben, die Sie ausführen möchten.
Notiz
Diese Funktion ist rechenintensiv, da igel viele verschiedene Modelle ausprobieren und deren Leistung vergleichen würde, um das „beste“ Modell zu finden.
Sie können den Befehl „help“ ausführen, um Anweisungen zu erhalten. Sie können auch Hilfe für Unterbefehle ausführen!
$ igel --helpDer erste Schritt besteht darin, eine Yaml-Datei bereitzustellen (Sie können bei Bedarf auch JSON verwenden).
Sie können dies manuell tun, indem Sie eine .yaml-Datei erstellen (konventionell igel.yaml genannt, aber Sie können einen beliebigen Namen vergeben) und sie selbst bearbeiten. Wenn Sie jedoch faul sind (und das sind Sie wahrscheinlich, so wie ich :D), können Sie schnell mit dem Befehl igel init loslegen, der im Handumdrehen eine grundlegende Konfigurationsdatei für Sie erstellt.
"""
igel init --help
Example:
If I want to use neural networks to classify whether someone is sick or not using the indian-diabetes dataset,
then I would use this command to initialize a yaml file n.b. you may need to rename outcome column in .csv to sick:
$ igel init -type " classification " -model " NeuralNetwork " -target " sick "
"""
$ igel initNach der Ausführung des Befehls wird im aktuellen Arbeitsverzeichnis eine igel.yaml-Datei für Sie erstellt. Sie können es ausprobieren und bei Bedarf ändern, ansonsten können Sie auch alles von Grund auf neu erstellen.
# model definition
model :
# in the type field, you can write the type of problem you want to solve. Whether regression, classification or clustering
# Then, provide the algorithm you want to use on the data. Here I'm using the random forest algorithm
type : classification
algorithm : RandomForest # make sure you write the name of the algorithm in pascal case
arguments :
n_estimators : 100 # here, I set the number of estimators (or trees) to 100
max_depth : 30 # set the max_depth of the tree
# target you want to predict
# Here, as an example, I'm using the famous indians-diabetes dataset, where I want to predict whether someone have diabetes or not.
# Depending on your data, you need to provide the target(s) you want to predict here
target :
- sickIm obigen Beispiel verwende ich eine Zufallsstruktur, um abhängig von einigen Merkmalen im Datensatz zu klassifizieren, ob jemand Diabetes hat oder nicht. In diesem Beispieldatensatz habe ich den berühmten indischen Diabetes verwendet.
Beachten Sie, dass ich n_estimators und max_depth als zusätzliche Argumente an das Modell übergeben habe. Wenn Sie keine Argumente angeben, wird der Standardwert verwendet. Sie müssen sich nicht die Argumente für jedes Modell merken. Sie können jederzeit igel models in Ihrem Terminal ausführen. Dadurch gelangen Sie in den interaktiven Modus, in dem Sie aufgefordert werden, das Modell einzugeben, das Sie verwenden möchten, sowie den Typ des Problems, das Sie lösen möchten. Igel zeigt Ihnen dann Informationen zum Modell und einen Link an, über den Sie eine Liste der verfügbaren Argumente und deren Verwendung einsehen können.
Führen Sie diesen Befehl im Terminal aus, um ein Modell anzupassen/zu trainieren. Geben Sie dabei den Pfad zu Ihrem Datensatz und den Pfad zur Yaml-Datei an
$ igel fit --data_path ' path_to_your_csv_dataset.csv ' --yaml_path ' path_to_your_yaml_file.yaml '
# or shorter
$ igel fit -dp ' path_to_your_csv_dataset.csv ' -yml ' path_to_your_yaml_file.yaml '
"""
That's it. Your "trained" model can be now found in the model_results folder
(automatically created for you in your current working directory).
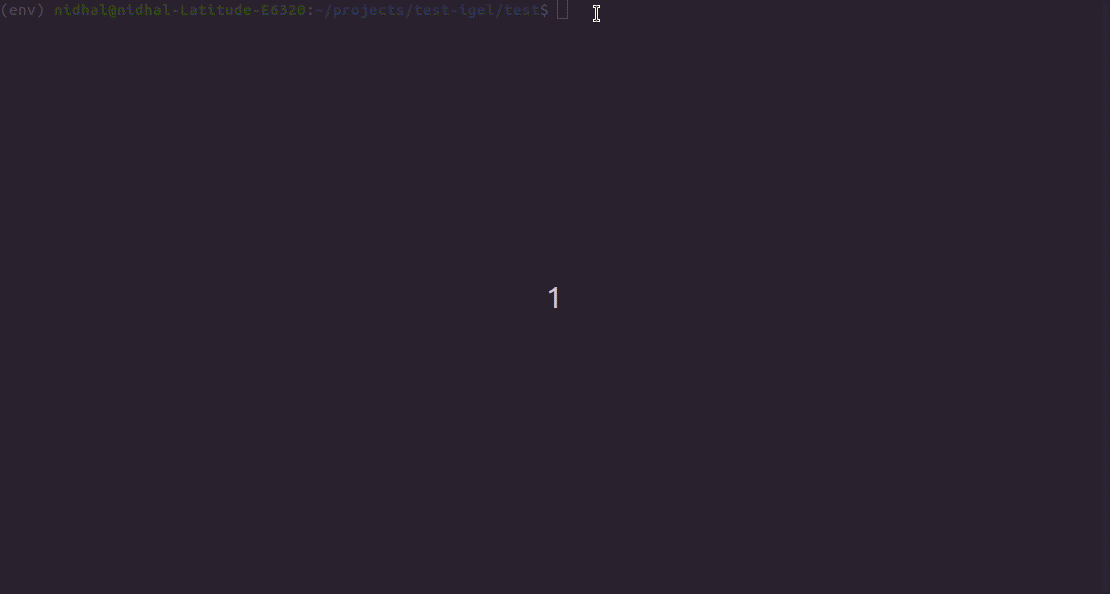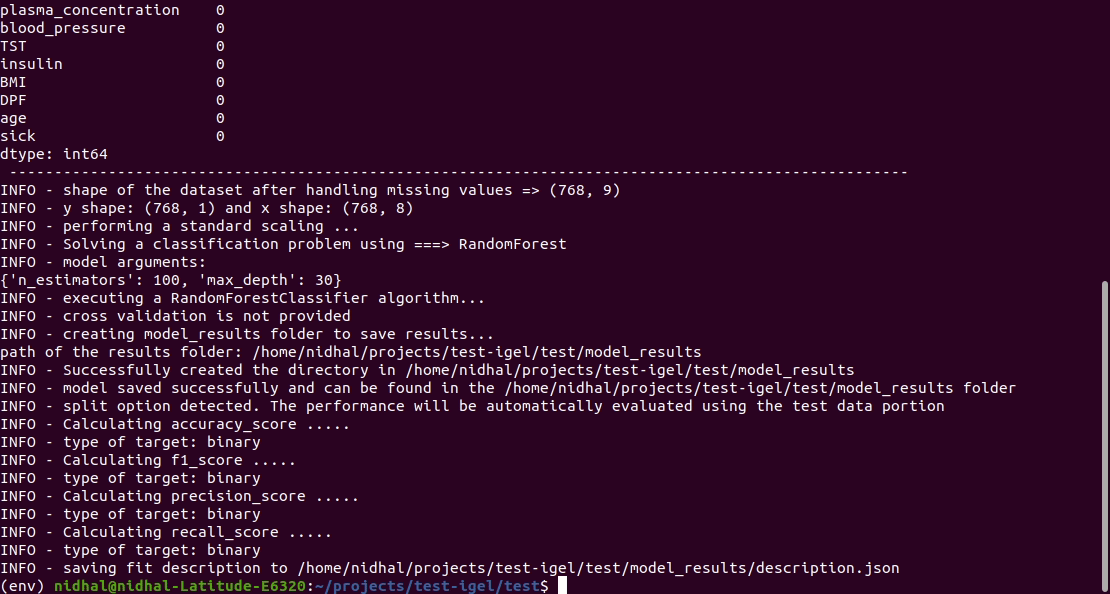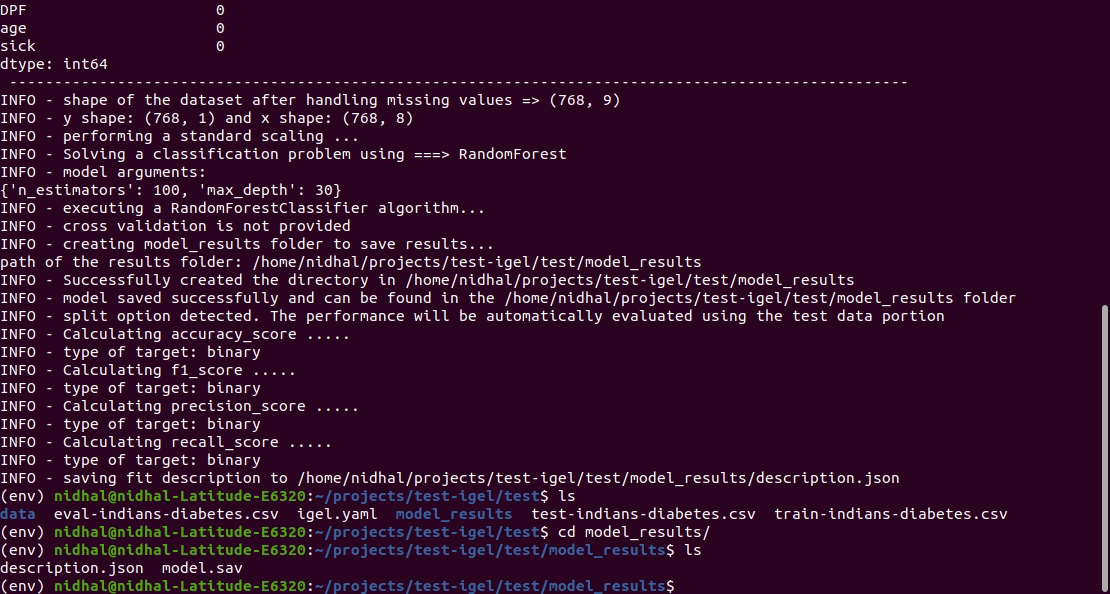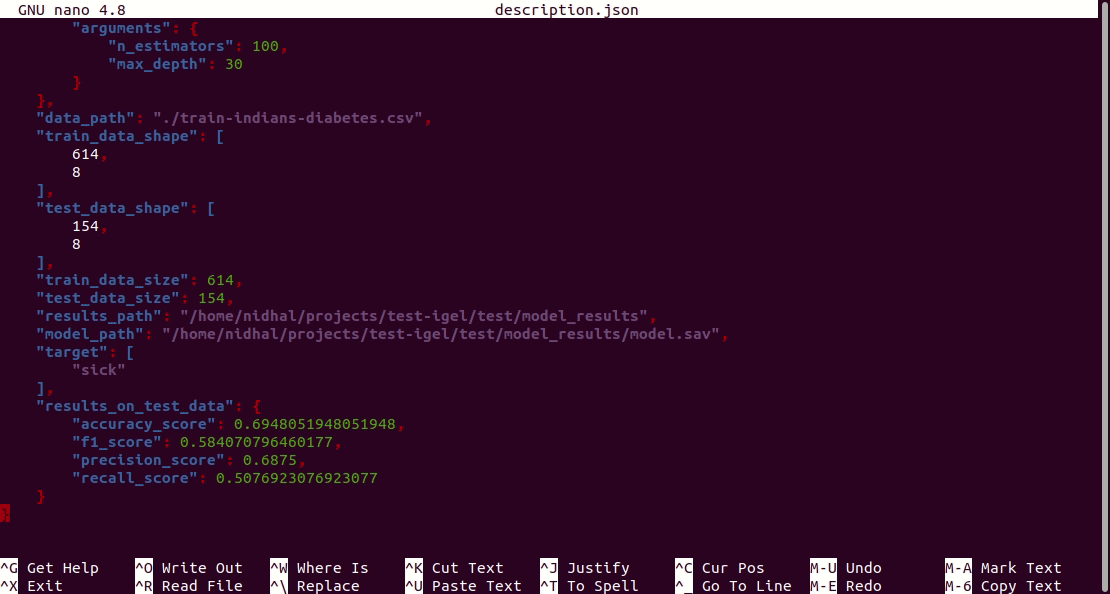
Furthermore, a description can be found in the description.json file inside the model_results folder.
"""
Anschließend können Sie das trainierte/vorangepasste Modell bewerten:
$ igel evaluate -dp ' path_to_your_evaluation_dataset.csv '
"""
This will automatically generate an evaluation.json file in the current directory, where all evaluation results are stored
"""
Schließlich können Sie das trainierte/vorangepasste Modell verwenden, um Vorhersagen zu treffen, wenn Sie mit den Bewertungsergebnissen zufrieden sind:
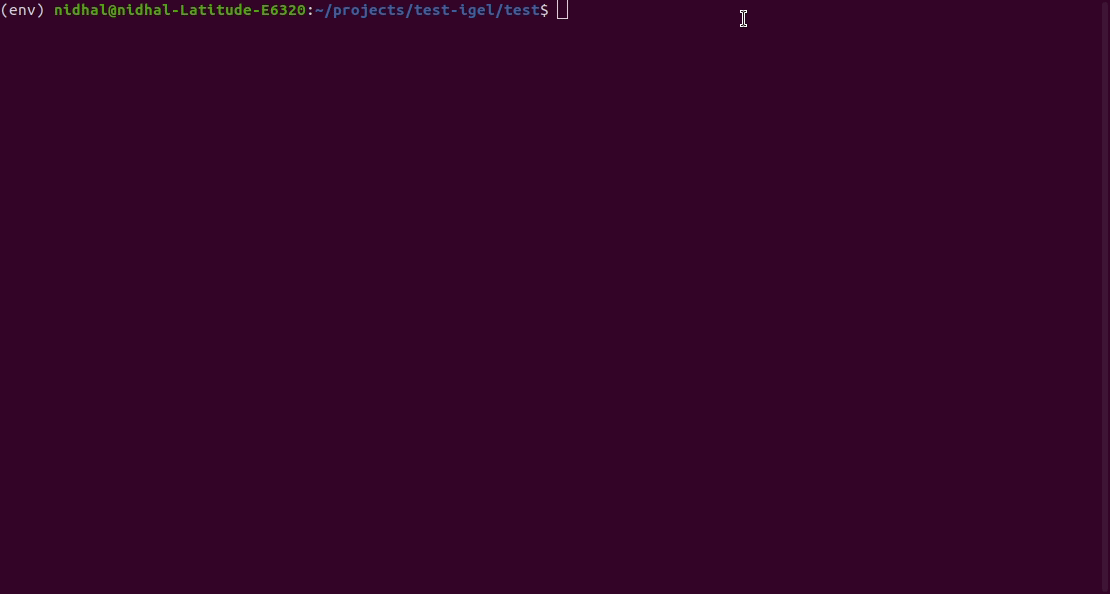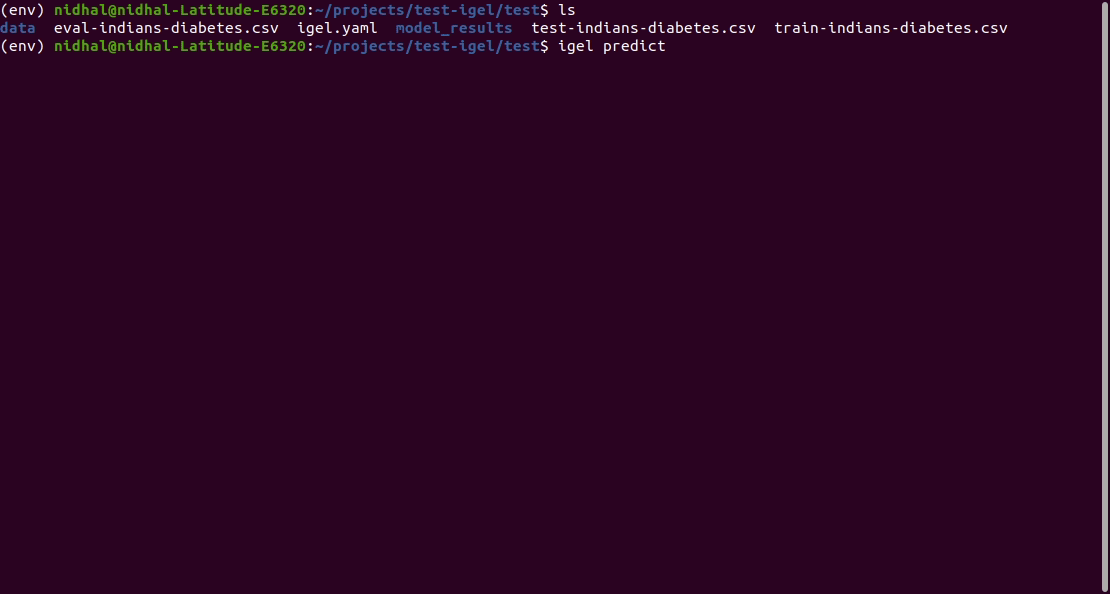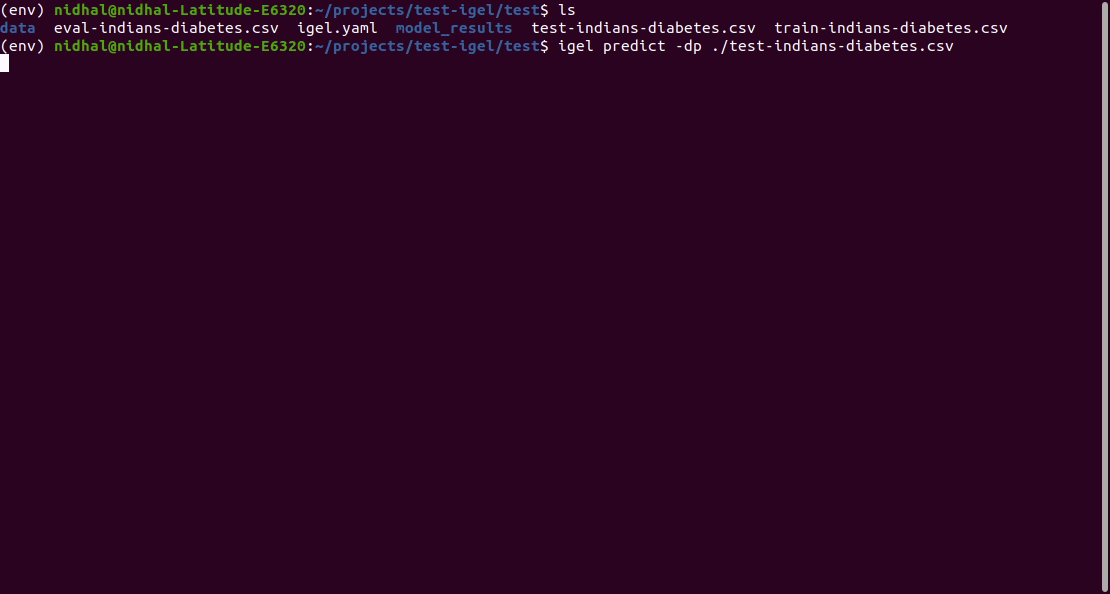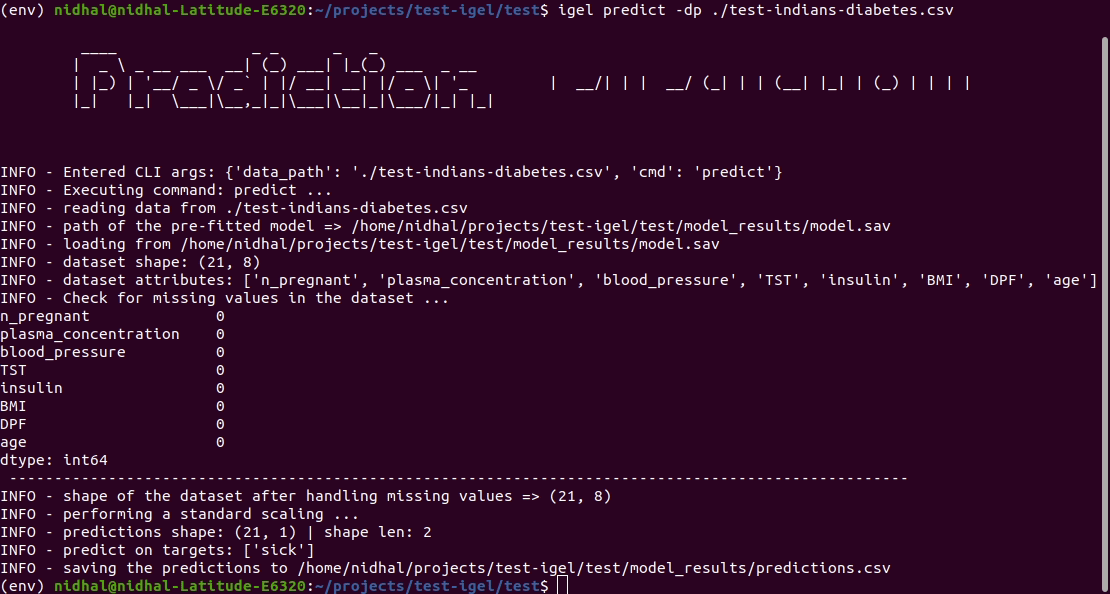
$ igel predict -dp ' path_to_your_test_dataset.csv '
"""
This will generate a predictions.csv file in your current directory, where all predictions are stored in a csv file
"""
Sie können die Phasen Trainieren, Auswerten und Vorhersagen mit einem einzigen Befehl namens Experiment kombinieren:
$ igel experiment -DP " path_to_train_data path_to_eval_data path_to_test_data " -yml " path_to_yaml_file "
"""
This will run fit using train_data, evaluate using eval_data and further generate predictions using the test_data
"""
Sie können das trainierte/vorangepasste Sklearn-Modell in ONNX exportieren:
$ igel export -dp " path_to_pre-fitted_sklearn_model "
"""
This will convert the sklearn model into ONNX
""" from igel import Igel
Igel ( cmd = "fit" , data_path = "path_to_your_dataset" , yaml_path = "path_to_your_yaml_file" )
"""
check the examples folder for more
"""Der nächste Schritt besteht darin, Ihr Modell in der Produktion zu verwenden. Igel hilft Ihnen auch bei dieser Aufgabe, indem es den Befehl „serve“ bereitstellt. Wenn Sie den Befehl „serve“ ausführen, wird igel angewiesen, Ihr Modell bereitzustellen. Genauer gesagt erstellt igel automatisch einen REST-Server und stellt Ihr Modell auf einem bestimmten Host und Port bereit, den Sie konfigurieren können, indem Sie diese als CLI-Optionen übergeben.
Der einfachste Weg ist, Folgendes auszuführen:
$ igel serve --model_results_dir " path_to_model_results_directory "Beachten Sie, dass igel die CLI-Option --model_results_dir oder kurz -res_dir benötigt, um das Modell zu laden und den Server zu starten. Standardmäßig stellt igel Ihr Modell auf localhost:8000 bereit. Sie können dies jedoch leicht überschreiben, indem Sie eine Host- und eine Port-CLI-Option angeben.
$ igel serve --model_results_dir " path_to_model_results_directory " --host " 127.0.0.1 " --port 8000Igel verwendet FastAPI zum Erstellen des REST-Servers, einem modernen Hochleistungs-Framework, und Uvicorn, um ihn unter der Haube auszuführen.
Dieses Beispiel wurde mit einem vorab trainierten Modell (erstellt durch Ausführen von igel init --target sick -type classification) und dem indischen Diabetes-Datensatz unter Beispiele/Daten erstellt. Die Überschriften der Spalten in der ursprünglichen CSV lauten „preg“, „plas“, „pres“, „skin“, „test“, „mass“, „pedi“ und „age“.
CURL:
$ curl -X POST localhost:8080/predict --header " Content-Type:application/json " -d ' {"preg": 1, "plas": 180, "pres": 50, "skin": 12, "test": 1, "mass": 456, "pedi": 0.442, "age": 50} '
Outputs: {"prediction":[[0.0]]}$ curl -X POST localhost:8080/predict --header " Content-Type:application/json " -d ' {"preg": [1, 6, 10], "plas":[192, 52, 180], "pres": [40, 30, 50], "skin": [25, 35, 12], "test": [0, 1, 1], "mass": [456, 123, 155], "pedi": [0.442, 0.22, 0.19], "age": [50, 40, 29]} '
Outputs: {"prediction":[[1.0],[0.0],[0.0]]}Vorbehalte/Einschränkungen:
Beispielhafte Verwendung des Python-Clients:
from python_client import IgelClient
# the client allows additional args with defaults:
# scheme="http", endpoint="predict", missing_values="mean"
client = IgelClient ( host = 'localhost' , port = 8080 )
# you can post other types of files compatible with what Igel data reading allows
client . post ( "my_batch_file_for_predicting.csv" )
Outputs : < Response 200 > : { "prediction" :[[ 1.0 ],[ 0.0 ],[ 0.0 ]]}Das Hauptziel von igel besteht darin, Ihnen eine Möglichkeit zu bieten, Modelle zu trainieren/anzupassen, zu bewerten und zu verwenden, ohne Code schreiben zu müssen. Stattdessen müssen Sie lediglich in einer einfachen Yaml-Datei angeben/beschreiben, was Sie tun möchten.
Grundsätzlich stellen Sie Beschreibungen bzw. Konfigurationen in der Yaml-Datei als Schlüssel-Wert-Paare bereit. Hier ist eine Übersicht aller (vorerst) unterstützten Konfigurationen:
# dataset operations
dataset :
type : csv # [str] -> type of your dataset
read_data_options : # options you want to supply for reading your data (See the detailed overview about this in the next section)
sep : # [str] -> Delimiter to use.
delimiter : # [str] -> Alias for sep.
header : # [int, list of int] -> Row number(s) to use as the column names, and the start of the data.
names : # [list] -> List of column names to use
index_col : # [int, str, list of int, list of str, False] -> Column(s) to use as the row labels of the DataFrame,
usecols : # [list, callable] -> Return a subset of the columns
squeeze : # [bool] -> If the parsed data only contains one column then return a Series.
prefix : # [str] -> Prefix to add to column numbers when no header, e.g. ‘X’ for X0, X1, …
mangle_dupe_cols : # [bool] -> Duplicate columns will be specified as ‘X’, ‘X.1’, …’X.N’, rather than ‘X’…’X’. Passing in False will cause data to be overwritten if there are duplicate names in the columns.
dtype : # [Type name, dict maping column name to type] -> Data type for data or columns
engine : # [str] -> Parser engine to use. The C engine is faster while the python engine is currently more feature-complete.
converters : # [dict] -> Dict of functions for converting values in certain columns. Keys can either be integers or column labels.
true_values : # [list] -> Values to consider as True.
false_values : # [list] -> Values to consider as False.
skipinitialspace : # [bool] -> Skip spaces after delimiter.
skiprows : # [list-like] -> Line numbers to skip (0-indexed) or number of lines to skip (int) at the start of the file.
skipfooter : # [int] -> Number of lines at bottom of file to skip
nrows : # [int] -> Number of rows of file to read. Useful for reading pieces of large files.
na_values : # [scalar, str, list, dict] -> Additional strings to recognize as NA/NaN.
keep_default_na : # [bool] -> Whether or not to include the default NaN values when parsing the data.
na_filter : # [bool] -> Detect missing value markers (empty strings and the value of na_values). In data without any NAs, passing na_filter=False can improve the performance of reading a large file.
verbose : # [bool] -> Indicate number of NA values placed in non-numeric columns.
skip_blank_lines : # [bool] -> If True, skip over blank lines rather than interpreting as NaN values.
parse_dates : # [bool, list of int, list of str, list of lists, dict] -> try parsing the dates
infer_datetime_format : # [bool] -> If True and parse_dates is enabled, pandas will attempt to infer the format of the datetime strings in the columns, and if it can be inferred, switch to a faster method of parsing them.
keep_date_col : # [bool] -> If True and parse_dates specifies combining multiple columns then keep the original columns.
dayfirst : # [bool] -> DD/MM format dates, international and European format.
cache_dates : # [bool] -> If True, use a cache of unique, converted dates to apply the datetime conversion.
thousands : # [str] -> the thousands operator
decimal : # [str] -> Character to recognize as decimal point (e.g. use ‘,’ for European data).
lineterminator : # [str] -> Character to break file into lines.
escapechar : # [str] -> One-character string used to escape other characters.
comment : # [str] -> Indicates remainder of line should not be parsed. If found at the beginning of a line, the line will be ignored altogether. This parameter must be a single character.
encoding : # [str] -> Encoding to use for UTF when reading/writing (ex. ‘utf-8’).
dialect : # [str, csv.Dialect] -> If provided, this parameter will override values (default or not) for the following parameters: delimiter, doublequote, escapechar, skipinitialspace, quotechar, and quoting
delim_whitespace : # [bool] -> Specifies whether or not whitespace (e.g. ' ' or ' ') will be used as the sep
low_memory : # [bool] -> Internally process the file in chunks, resulting in lower memory use while parsing, but possibly mixed type inference.
memory_map : # [bool] -> If a filepath is provided for filepath_or_buffer, map the file object directly onto memory and access the data directly from there. Using this option can improve performance because there is no longer any I/O overhead.
random_numbers : # random numbers options in case you wanted to generate the same random numbers on each run
generate_reproducible : # [bool] -> set this to true to generate reproducible results
seed : # [int] -> the seed number is optional. A seed will be set up for you if you didn't provide any
split : # split options
test_size : 0.2 # [float] -> 0.2 means 20% for the test data, so 80% are automatically for training
shuffle : true # [bool] -> whether to shuffle the data before/while splitting
stratify : None # [list, None] -> If not None, data is split in a stratified fashion, using this as the class labels.
preprocess : # preprocessing options
missing_values : mean # [str] -> other possible values: [drop, median, most_frequent, constant] check the docs for more
encoding :
type : oneHotEncoding # [str] -> other possible values: [labelEncoding]
scale : # scaling options
method : standard # [str] -> standardization will scale values to have a 0 mean and 1 standard deviation | you can also try minmax
target : inputs # [str] -> scale inputs. | other possible values: [outputs, all] # if you choose all then all values in the dataset will be scaled
# model definition
model :
type : classification # [str] -> type of the problem you want to solve. | possible values: [regression, classification, clustering]
algorithm : NeuralNetwork # [str (notice the pascal case)] -> which algorithm you want to use. | type igel algorithms in the Terminal to know more
arguments : # model arguments: you can check the available arguments for each model by running igel help in your terminal
use_cv_estimator : false # [bool] -> if this is true, the CV class of the specific model will be used if it is supported
cross_validate :
cv : # [int] -> number of kfold (default 5)
n_jobs : # [signed int] -> The number of CPUs to use to do the computation (default None)
verbose : # [int] -> The verbosity level. (default 0)
hyperparameter_search :
method : grid_search # method you want to use: grid_search and random_search are supported
parameter_grid : # put your parameters grid here that you want to use, an example is provided below
param1 : [val1, val2]
param2 : [val1, val2]
arguments : # additional arguments you want to provide for the hyperparameter search
cv : 5 # number of folds
refit : true # whether to refit the model after the search
return_train_score : false # whether to return the train score
verbose : 0 # verbosity level
# target you want to predict
target : # list of strings: basically put here the column(s), you want to predict that exist in your csv dataset
- put the target you want to predict here
- you can assign many target if you are making a multioutput prediction Notiz
Igel verwendet Pandas unter der Haube, um die Daten zu lesen und zu analysieren. Daher finden Sie diese optionalen Datenparameter auch in der offiziellen Pandas-Dokumentation.
Nachfolgend finden Sie eine detaillierte Übersicht über die Konfigurationen, die Sie in der Yaml- (oder JSON-)Datei bereitstellen können. Beachten Sie, dass Sie sicherlich nicht alle Konfigurationswerte für den Datensatz benötigen. Sie sind optional. Im Allgemeinen wird igel herausfinden, wie Ihr Datensatz gelesen wird.
Sie können jedoch Abhilfe schaffen, indem Sie mithilfe dieses Abschnitts „read_data_options“ zusätzliche Felder bereitstellen. Einer der hilfreichen Werte ist meiner Meinung nach beispielsweise „sep“, der definiert, wie Ihre Spalten im CSV-Datensatz getrennt sind. Im Allgemeinen werden CSV-Datensätze durch Kommas getrennt, was auch hier der Standardwert ist. In Ihrem Fall kann es jedoch sein, dass es durch ein Semikolon getrennt wird.
Daher können Sie dies in den read_data_options angeben. Fügen Sie einfach das sep: ";" hinzu. unter read_data_options.
| Parameter | Typ | Erläuterung |
|---|---|---|
| sep | str, Standard ',' | Zu verwendendes Trennzeichen. Wenn sep „None“ ist, kann die C-Engine das Trennzeichen nicht automatisch erkennen, die Python-Parsing-Engine jedoch schon, was bedeutet, dass letztere verwendet wird und das Trennzeichen automatisch von Pythons integriertem Sniffer-Tool csv.Sniffer erkennt. Darüber hinaus werden Trennzeichen, die länger als 1 Zeichen sind und sich von „s+“ unterscheiden, als reguläre Ausdrücke interpretiert und erzwingen ebenfalls die Verwendung der Python-Parsing-Engine. Beachten Sie, dass Regex-Trennzeichen dazu neigen, Daten in Anführungszeichen zu ignorieren. Regex-Beispiel: 'rt'. |
| Trennzeichen | Standard: Keine | Alias für sep. |
| Kopfzeile | int, Liste von int, Standard 'infer' | Zeilennummern, die als Spaltennamen verwendet werden sollen, und der Beginn der Daten. Das Standardverhalten besteht darin, die Spaltennamen abzuleiten: Wenn keine Namen übergeben werden, ist das Verhalten identisch mit header=0 und Spaltennamen werden aus der ersten Zeile der Datei abgeleitet. Wenn Spaltennamen explizit übergeben werden, ist das Verhalten identisch mit header=None . Übergeben Sie explizit header=0, um vorhandene Namen ersetzen zu können. Der Header kann eine Liste von Ganzzahlen sein, die Zeilenpositionen für einen Multiindex in den Spalten angeben, z. B. [0,1,3]. Dazwischenliegende Zeilen, die nicht angegeben sind, werden übersprungen (z. B. 2 in diesem Beispiel wird übersprungen). Beachten Sie, dass dieser Parameter kommentierte Zeilen und leere Zeilen ignoriert, wenn skip_blank_lines=True, sodass header=0 die erste Datenzeile und nicht die erste Zeile der Datei bezeichnet. |
| Namen | arrayartig, optional | Liste der zu verwendenden Spaltennamen. Wenn die Datei eine Kopfzeile enthält, sollten Sie header=0 explizit übergeben, um die Spaltennamen zu überschreiben. Duplikate in dieser Liste sind nicht zulässig. |
| index_col | int, str, Sequenz von int/str oder False, Standardwert: Keine | Spalte(n), die als Zeilenbeschriftungen des DataFrame verwendet werden sollen, entweder als Zeichenfolgenname oder als Spaltenindex angegeben. Wenn eine Folge von int/str angegeben ist, wird ein MultiIndex verwendet. Hinweis: index_col=False kann verwendet werden, um Pandas zu zwingen, die erste Spalte nicht als Index zu verwenden, z. B. wenn Sie eine fehlerhafte Datei mit Trennzeichen am Ende jeder Zeile haben. |
| usecols | listenartig oder aufrufbar, optional | Gibt eine Teilmenge der Spalten zurück. Wenn es sich um eine Liste handelt, müssen alle Elemente entweder positionell sein (dh ganzzahlige Indizes in den Dokumentspalten) oder Zeichenfolgen sein, die Spaltennamen entsprechen, die entweder vom Benutzer in Namen angegeben oder aus der/den Kopfzeile(n) des Dokuments abgeleitet werden. Ein gültiger listenartiger usecols-Parameter wäre beispielsweise [0, 1, 2] oder ['foo', 'bar', 'baz']. Die Reihenfolge der Elemente wird ignoriert, daher ist usecols=[0, 1] dasselbe wie [1, 0]. Um einen DataFrame aus Daten mit beibehaltener Elementreihenfolge zu instanziieren, verwenden Sie pd.read_csv(data, usecols=['foo', 'bar'])[['foo', 'bar']] für Spalten in ['foo', 'bar' '] order oder pd.read_csv(data, usecols=['foo', 'bar'])[['bar', 'foo']] für ['bar', 'foo'] Befehl. Wenn sie aufrufbar ist, wird die aufrufbare Funktion anhand der Spaltennamen ausgewertet und gibt Namen zurück, bei denen die aufrufbare Funktion „True“ ergibt. Ein Beispiel für ein gültiges aufrufbares Argument wäre lambda x: x.upper() in ['AAA', 'BBB', 'DDD']. Die Verwendung dieses Parameters führt zu einer viel schnelleren Analysezeit und einer geringeren Speichernutzung. |
| quetschen | bool, Standardwert False | Wenn die analysierten Daten nur eine Spalte enthalten, wird eine Serie zurückgegeben. |
| Präfix | str, optional | Präfix zum Hinzufügen zu Spaltennummern, wenn keine Überschrift vorhanden ist, z. B. „X“ für X0, X1, … |
| mangle_dupe_cols | bool, Standardwert True | Doppelte Spalten werden als „X“, „X.1“, … „X.N“ und nicht als „X“ … „X“ angegeben. Die Übergabe von False führt dazu, dass Daten überschrieben werden, wenn in den Spalten doppelte Namen vorhanden sind. |
| dtype | {'c', 'python'}, optional | Zu verwendende Parser-Engine. Die C-Engine ist schneller, während die Python-Engine derzeit über einen umfassenderen Funktionsumfang verfügt. |
| Konverter | Diktat, optional | Diktat von Funktionen zum Konvertieren von Werten in bestimmten Spalten. Schlüssel können entweder Ganzzahlen oder Spaltenbezeichnungen sein. |
| wahre_Werte | Liste, optional | Als wahr zu betrachtende Werte. |
| falsche_Werte | Liste, optional | Als falsch zu betrachtende Werte. |
| überspringeninitialspace | bool, Standardwert False | Überspringen Sie Leerzeichen nach dem Trennzeichen. |
| Skiprows | listenartig, int oder aufrufbar, optional | Zu überspringende Zeilennummern (0-indiziert) oder Anzahl der zu überspringenden Zeilen (int) am Anfang der Datei. Wenn sie aufrufbar ist, wird die aufrufbare Funktion anhand der Zeilenindizes ausgewertet und gibt „True“ zurück, wenn die Zeile übersprungen werden soll, andernfalls „False“. Ein Beispiel für ein gültiges aufrufbares Argument wäre Lambda x: x in [0, 2]. |
| Skipfooter | int, Standard 0 | Anzahl der Zeilen am Ende der Datei, die übersprungen werden sollen (nicht unterstützt mit engine='c'). |
| nrows | int, optional | Anzahl der zu lesenden Dateizeilen. Nützlich zum Lesen großer Dateiteile. |
| na_values | Skalar, str, listenartig oder dict, optional | Zusätzliche Zeichenfolgen zur Erkennung als NA/NaN. Wenn das Diktat übergeben wird, spezifische NA-Werte pro Spalte. Standardmäßig werden die folgenden Werte als NaN interpretiert: '', '#N/A', '#N/AN/A', '#NA', '-1.#IND', '-1.#QNAN', '-NaN', '-nan', '1.#IND', '1.#QNAN', '<NA>', 'N/A', 'NA', 'NULL', 'NaN', 'n /a', 'nan', 'null'. |
| keep_default_na | bool, Standardwert True | Ob beim Parsen der Daten die Standard-NaN-Werte einbezogen werden sollen oder nicht. Abhängig davon, ob na_values übergeben wird, ist das Verhalten wie folgt: Wenn keep_default_na True ist und na_values angegeben sind, wird na_values an die für die Analyse verwendeten Standard-NaN-Werte angehängt. Wenn keep_default_na True ist und na_values nicht angegeben sind, werden nur die Standard-NaN-Werte zum Parsen verwendet. Wenn keep_default_na False ist und na_values angegeben sind, werden nur die angegebenen NaN-Werte na_values zum Parsen verwendet. Wenn keep_default_na „False“ ist und keine „na_values“ angegeben sind, werden keine Zeichenfolgen als NaN geparst. Beachten Sie, dass die Parameter keep_default_na und na_values ignoriert werden, wenn na_filter als False übergeben wird. |
| na_filter | bool, Standardwert True | Erkennen Sie fehlende Wertmarkierungen (leere Zeichenfolgen und den Wert von na_values). Bei Daten ohne NAs kann die Übergabe von na_filter=False die Leistung beim Lesen einer großen Datei verbessern. |
| ausführlich | bool, Standardwert False | Geben Sie die Anzahl der NA-Werte an, die in nicht numerischen Spalten platziert sind. |
| überspringen_leere_Zeilen | bool, Standardwert True | Wenn True, überspringen Sie Leerzeilen, anstatt sie als NaN-Werte zu interpretieren. |
| parse_dates | bool oder Liste von int oder Namen oder Liste von Listen oder dict, Standardwert False | Das Verhalten ist wie folgt: boolean. Wenn True -> versuchen Sie, den Index zu analysieren. Liste von int oder Namen. Beispiel: Wenn [1, 2, 3] -> Versuchen Sie, die Spalten 1, 2, 3 jeweils als separate Datumsspalte zu analysieren. Liste von Listen. zB Wenn [[1, 3]] -> Spalten 1 und 3 kombinieren und als einzelne Datumsspalte analysieren. dict, zB {'foo' : [1, 3]} -> Analysieren Sie die Spalten 1, 3 als Datum und rufen Sie das Ergebnis 'foo' auf. Wenn eine Spalte oder ein Index nicht als Array von Datums- und Uhrzeitangaben dargestellt werden kann, beispielsweise aufgrund eines nicht analysierbaren Werts oder Bei einer Mischung aus Zeitzonen wird die Spalte oder der Index unverändert als Objektdatentyp zurückgegeben. |
| infer_datetime_format | bool, Standardwert False | Wenn „True“ und „parse_dates“ aktiviert sind, versucht Pandas, das Format der Datums-/Uhrzeitzeichenfolgen in den Spalten abzuleiten, und wenn es abgeleitet werden kann, wechselt es zu einer schnelleren Methode zum Parsen. In einigen Fällen kann dies die Parsing-Geschwindigkeit um das 5- bis 10-fache erhöhen. |
| keep_date_col | bool, Standardwert False | Wenn „True“ und „parse_dates“ die Kombination mehrerer Spalten angibt, behalten Sie die ursprünglichen Spalten bei. |
| date_parser | Funktion, optional | Funktion zum Konvertieren einer Folge von Zeichenfolgenspalten in ein Array von Datetime-Instanzen. Standardmäßig wird dateutil.parser.parser für die Konvertierung verwendet. Pandas versucht, date_parser auf drei verschiedene Arten aufzurufen und geht zum nächsten über, wenn eine Ausnahme auftritt: 1) Übergeben Sie ein oder mehrere Arrays (wie durch parse_dates definiert) als Argumente; 2) Verketten Sie (zeilenweise) die Zeichenfolgenwerte aus den durch parse_dates definierten Spalten in einem einzigen Array und übergeben Sie dieses; und 3) rufen Sie date_parser einmal für jede Zeile auf und verwenden Sie dabei eine oder mehrere Zeichenfolgen (entsprechend den durch parse_dates definierten Spalten) als Argumente. |
| am ersten Tag | bool, Standardwert False | Datumsangaben im TT/MM-Format, internationales und europäisches Format. |
| Cache_Daten | bool, Standardwert True | Wenn True, verwenden Sie einen Cache mit eindeutigen, konvertierten Datumsangaben, um die Datum/Uhrzeit-Konvertierung anzuwenden. Kann beim Parsen doppelter Datumszeichenfolgen zu einer erheblichen Beschleunigung führen, insbesondere bei solchen mit Zeitzonenversätzen. |
| Tausende | str, optional | Tausendertrennzeichen. |
| dezimal | str, Standard '.' | Zeichen, das als Dezimalpunkt erkannt werden soll (z. B. „,“ für europäische Daten verwenden). |
| Linienterminator | str (Länge 1), optional | Zeichen zum Aufteilen der Datei in Zeilen. Nur gültig mit C-Parser. |
| escapechar | str (Länge 1), optional | Eine aus einem Zeichen bestehende Zeichenfolge, die als Escapezeichen für andere Zeichen verwendet wird. |
| Kommentar | str, optional | Gibt an, dass der Rest der Zeile nicht analysiert werden soll. Wenn es am Anfang einer Zeile steht, wird die Zeile vollständig ignoriert. |
| Codierung | str, optional | Codierung, die beim Lesen/Schreiben für UTF verwendet werden soll (z. B. „utf-8“). |
| Dialekt | str oder csv.Dialect, optional | Falls angegeben, überschreibt dieser Parameter die Werte (Standard oder nicht) für die folgenden Parameter: Trennzeichen, doppeltes Anführungszeichen, Escapechar, Skipinitialspace, Anführungszeichen und Anführungszeichen |
| low_memory | bool, Standardwert True | Verarbeiten Sie die Datei intern in Blöcken, was zu einer geringeren Speichernutzung beim Parsen, aber möglicherweise zu gemischten Typrückschlüssen führt. Um sicherzustellen, dass keine gemischten Typen vorhanden sind, legen Sie entweder „False“ fest oder geben Sie den Typ mit dem Parameter „dtype“ an. Beachten Sie, dass die gesamte Datei unabhängig davon in einen einzelnen DataFrame eingelesen wird. |
| Speicherkarte | bool, Standardwert False | Ordnen Sie das Dateiobjekt direkt dem Speicher zu und greifen Sie von dort aus direkt auf die Daten zu. Die Verwendung dieser Option kann die Leistung verbessern, da kein E/A-Overhead mehr entsteht. |
In diesem Abschnitt wird eine vollständige End-to-End-Lösung bereitgestellt, um die Leistungsfähigkeit von igel unter Beweis zu stellen. Wie bereits erläutert, müssen Sie eine Yaml-Konfigurationsdatei erstellen. Hier ist ein End-to-End-Beispiel für die Vorhersage, ob jemand Diabetes hat oder nicht, mithilfe des Entscheidungsbaumalgorithmus . Der Datensatz befindet sich im Beispielordner.
model :
type : classification
algorithm : DecisionTree
target :
- sick $ igel fit -dp path_to_the_dataset -yml path_to_the_yaml_fileJetzt passt igel das Modell für Sie an und speichert es im Ordner „model_results“ in Ihrem aktuellen Verzeichnis.
Bewerten Sie das vorgefertigte Modell. Igel lädt das vorangepasste Modell aus dem Verzeichnis model_results und wertet es für Sie aus. Sie müssen lediglich den Befehl „evaluieren“ ausführen und den Pfad zu Ihren Bewertungsdaten angeben.
$ igel evaluate -dp path_to_the_evaluation_datasetDas ist es! Igel wertet das Modell aus und speichert Statistiken/Ergebnisse in einer Evaluation.json- Datei im Ordner „model_results“.
Verwenden Sie das vorangepasste Modell, um anhand neuer Daten Vorhersagen zu treffen. Dies wird von igel automatisch durchgeführt. Sie müssen lediglich den Pfad zu Ihren Daten angeben, für die Sie die Vorhersage verwenden möchten.
$ igel predict -dp path_to_the_new_datasetDas ist es! Igel verwendet das vorangepasste Modell, um Vorhersagen zu treffen und es in einer Datei „predictions.csv“ im Ordner „model_results“ zu speichern
Sie können auch einige Vorverarbeitungsmethoden oder andere Vorgänge ausführen, indem Sie diese in der Yaml-Datei bereitstellen. Hier ist ein Beispiel, bei dem die Daten zu 80 % für das Training und zu 20 % für die Validierung/Tests aufgeteilt werden. Außerdem werden die Daten beim Teilen gemischt.
Darüber hinaus werden die Daten vorverarbeitet, indem fehlende Werte durch den Mittelwert ersetzt werden (Sie können auch Median, Modus usw. verwenden). Weitere Informationen finden Sie unter diesem Link
# dataset operations
dataset :
split :
test_size : 0.2
shuffle : True
stratify : default
preprocess : # preprocessing options
missing_values : mean # other possible values: [drop, median, most_frequent, constant] check the docs for more
encoding :
type : oneHotEncoding # other possible values: [labelEncoding]
scale : # scaling options
method : standard # standardization will scale values to have a 0 mean and 1 standard deviation | you can also try minmax
target : inputs # scale inputs. | other possible values: [outputs, all] # if you choose all then all values in the dataset will be scaled
# model definition
model :
type : classification
algorithm : RandomForest
arguments :
# notice that this is the available args for the random forest model. check different available args for all supported models by running igel help
n_estimators : 100
max_depth : 20
# target you want to predict
target :
- sickAnschließend können Sie das Modell anpassen, indem Sie den igel-Befehl ausführen, wie in den anderen Beispielen gezeigt
$ igel fit -dp path_to_the_dataset -yml path_to_the_yaml_fileZur Auswertung
$ igel evaluate -dp path_to_the_evaluation_datasetFür die Produktion
$ igel predict -dp path_to_the_new_dataset Im Beispielordner im Repository finden Sie einen Datenordner, in dem der berühmte Indian-Diabetes-, Iris-Datensatz und der Linnerud-Datensatz (von Sklearn) gespeichert sind. Darüber hinaus gibt es in jedem Ordner End-to-End-Beispiele mit Skripten und Yaml-Dateien, die Ihnen den Einstieg erleichtern.
Der Ordner „indian-diabetes-example“ enthält zwei Beispiele, die Ihnen den Einstieg erleichtern:
Der Ordner „iris-example“ enthält ein Beispiel für eine logistische Regression , bei dem eine Vorverarbeitung (eine Hot-Codierung) an der Zielspalte durchgeführt wird, um Ihnen mehr über die Möglichkeiten von igel zu zeigen.
Darüber hinaus enthält das Multioutput-Beispiel ein Multioutput-Regressionsbeispiel . Schließlich enthält das cv-Beispiel ein Beispiel, das den Ridge-Klassifikator mit Kreuzvalidierung verwendet.
Im Ordner finden Sie auch Beispiele für eine Kreuzvalidierung und eine Hyperparametersuche.
Ich schlage vor, dass Sie mit den Beispielen und igel cli herumspielen. Wenn Sie möchten, können Sie jedoch auch fit.py, equal.py und Predict.py direkt ausführen.
Erstellen oder ändern Sie zunächst einen Datensatz mit Bildern, die basierend auf der Bildbezeichnung/-klasse in Unterordner kategorisiert werden. Wenn Sie beispielsweise Bilder von Hunden und Katzen haben, benötigen Sie zwei Unterordner:
Angenommen, diese beiden Unterordner befinden sich in einem übergeordneten Ordner namens „images“, geben Sie einfach die Daten an igel ein:
$ igel auto-train -dp ./images --task ImageClassificationIgel übernimmt alles von der Vorverarbeitung der Daten bis zur Optimierung der Hyperparameter. Am Ende wird das beste Modell im aktuellen Arbeitsverzeichnis gespeichert.
Erstellen oder ändern Sie zunächst einen Textdatensatz, der basierend auf der Textbezeichnung/-klasse in Unterordner kategorisiert wird. Wenn Sie beispielsweise über einen Textdatensatz mit positiven und negativen Rückmeldungen verfügen, benötigen Sie zwei Unterordner:
Angenommen, diese beiden Unterordner befinden sich in einem übergeordneten Ordner namens „texts“, geben Sie einfach Daten an igel ein:
$ igel auto-train -dp ./texts --task TextClassificationIgel übernimmt alles von der Vorverarbeitung der Daten bis zur Optimierung der Hyperparameter. Am Ende wird das beste Modell im aktuellen Arbeitsverzeichnis gespeichert.
Sie können die igel-Benutzeroberfläche auch ausführen, wenn Sie mit dem Terminal nicht vertraut sind. Installieren Sie igel einfach wie oben beschrieben auf Ihrem Computer. Führen Sie dann diesen einzelnen Befehl in Ihrem Terminal aus
$ igel guiDadurch wird die GUI geöffnet, die sehr einfach zu verwenden ist. Beispiele dafür, wie die GUI aussieht und wie man sie verwendet, finden Sie hier: https://github.com/nidhaloff/igel-ui
Sie können das Image zuerst vom Docker Hub abrufen
$ docker pull nidhaloff/igelDann nutzen Sie es:
$ docker run -it --rm -v $( pwd ) :/data nidhaloff/igel fit -yml ' your_file.yaml ' -dp ' your_dataset.csv 'Sie können igel in Docker ausführen, indem Sie zunächst das Image erstellen:
$ docker build -t igel .Und dann führen Sie es aus und hängen Ihr aktuelles Verzeichnis (es muss nicht das igel-Verzeichnis sein) als /data (das Arbeitsverzeichnis) innerhalb des Containers an:
$ docker run -it --rm -v $( pwd ) :/data igel fit -yml ' your_file.yaml ' -dp ' your_dataset.csv ' Wenn Sie auf Probleme stoßen, können Sie gerne ein Problem eröffnen. Darüber hinaus können Sie für weitere Informationen/Fragen Kontakt mit dem Autor aufnehmen.
Magst du igel? Sie können die Entwicklung dieses Projekts jederzeit unterstützen, indem Sie:
Sie halten dieses Projekt für nützlich und möchten neue Ideen, neue Funktionen, Fehlerbehebungen und eine Erweiterung der Dokumentation einbringen?
Beiträge sind jederzeit willkommen. Stellen Sie sicher, dass Sie zuerst die Richtlinien lesen
MIT-Lizenz
Copyright (c) 2020-heute, Nidhal Baccouri