
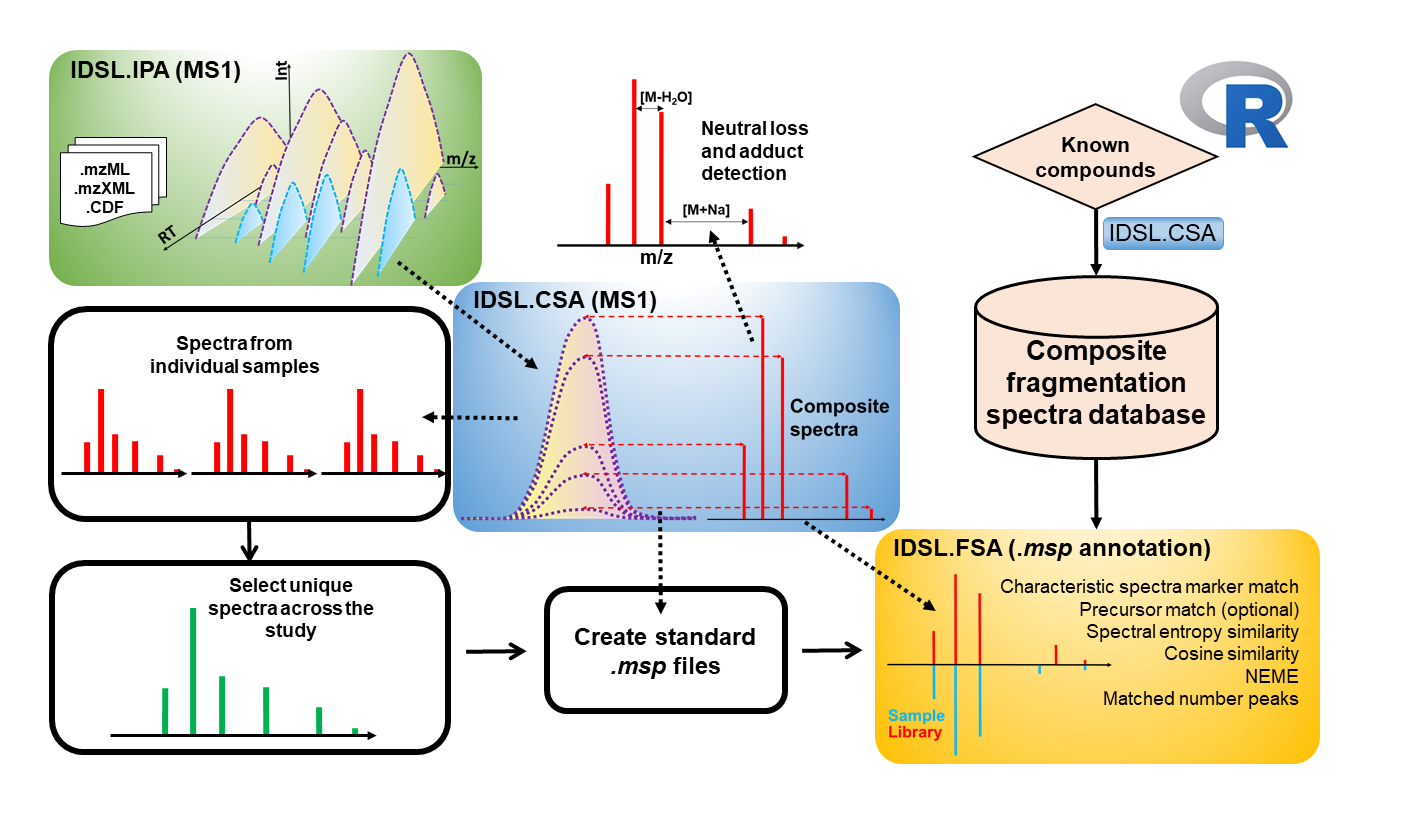
Das Komposit -Spektrenanalyse (IDSL.CSA) R für die Analyse von Massenspektrometriedaten wurde vom integrierten Data Science Laboratory für Metabolomics and Exposomics (IDSL.ME) entwickelt. Dieses Paket kann für die Entfaltung von Fragmentierungsspektren verwendet werden, die durch verschiedene Analysemethoden wie MS1-Verbundspektren-Dekonvolution-Analyse ( CSA ), datenabhängige Erfassung ( DDA ) und eine verschiedene datenunabhängige Erfassungsmethoden (MS E) erhalten werden , All-Ionen-Fragmentierung (AIF) und Swath-MS-Analysen. Ziel des IDSL.CSA -Pakets ist es, den Datenanalyseprozess zu optimieren und die Annotation der chemischen Struktur in den Bereichen Metabolomik und Exposomik zu verbessern.
Start in der Parameter -Tabelle von IDSL.CSA. install.packages("IDSL.CSA")
Vor der Verarbeitung Ihrer Massenspektrometriedaten ( MZXML , MZML , NETCDF ) unter Verwendung des IDSL.CSA-Workflows sollten Massenspektrometriedaten mit dem IDSL.IPA-Workflow verarbeitet werden, um chromatographische Informationen der Peaks ( M/Z-RT ) zu erfassen. Wenn die chromatographischen Informationen von individuellen und aggregierten ausgerichteten Peaklists unter Verwendung des IDSL.IPA -Workflows generiert wurden, laden Sie die Parameter -Tabelle von IDSL.CSA herunter und wählen Sie die Parameter entsprechend aus und verwenden Sie diese Tabelle als Eingabe für den IDSL.CSA -Workflow:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Befolgen Sie diese Schritte für eine schnelle Fallstudie (n = 33) ST002263 mit thermo-q-exakten HF-Hybrid-Orbitrap-Daten, die in den Hilic-ESI-Pos/NEG-Modi gesammelt wurden.
Prozessdaten für Rohmassenspektrometrie und chromatographische Informationen unter Verwendung der für IDSL.IPA beschriebenen Methode
Die zusammengesetzte Spektrenanalyse erfordert 39 Parameter, die in 5 separate Abschnitte verteilt sind, um eine vollständige Analyse in vollem Umfang zu erhalten. Verwenden Sie für diese Studie Standardparameterwerte, die in der Parameter -Tabelle von idsl.csa angegeben sind. Geben Sie als nächstes Informationen an
2.1. Wählen Sie in der Registerkarte Start Ja für Param0001 aus, um den CSA -Workflow nur zu verarbeiten.
2.2. CSA0005 für die HRMS -Datenstandortadresse (MS1 -HRMS -Daten auf MS1 -Ebene)
2.3. CSA0008 für die Adresse des vom IDSL.IPA -Workflow generierten peaklists -Verzeichnisse
2.4. CSA0009 für die Adresse des vom IDSL.IPA -Workflow generierten peak_alignment -Verzeichnis
2.5. CSA0011 für Ausgabestandort (.MSP -Dateien und EICs)
2.6. Sie können auch die Anzahl der Verarbeitungs -Threads mit CSA0004 gemäß Ihrer Rechenleistung erhöhen
Führen Sie diesen Befehl in R/Rstudio -Konsole oder Terminal aus:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Sie können die Ergebnisse an der Adresse analysieren, die Sie für CSA0011 angegeben haben.
4.1. CSA_MSP enthält eine .msp -Datei
4.2. Csa_adduct_annotation enthält Peaklists mit potenziellen Adduktinformationen
4.3. Peak_Alignment_Subset enthält Teilmengen aus ausgerichteten Peaktabellen für die Hauptionen in jedem CSA -Cluster
4.4. Aligned_spectra_table enthält Informationen für die CSA -Aggregation in der ausgerichteten Tabelle
[1] Fakouri Baygi, S., Kumar, Y. Barupal, DK IDSL.CSA: Verbundspektrenanalyse zur chemischen Annotation von nicht zielgeruchten Metabolomics -Datensätzen. Analytical Chemistry , 2023 , 95 (25) , 9480–9487.
[2] Fakouri Baygi, S., Kumar, Y. Barupal, DK IDSL. IPA charakterisiert den organischen chemischen Raum in nicht gezielten LC/HRMS -Datensätzen. Journal of Proteome Research , 2022 , 21 (6) , 1485-1494.