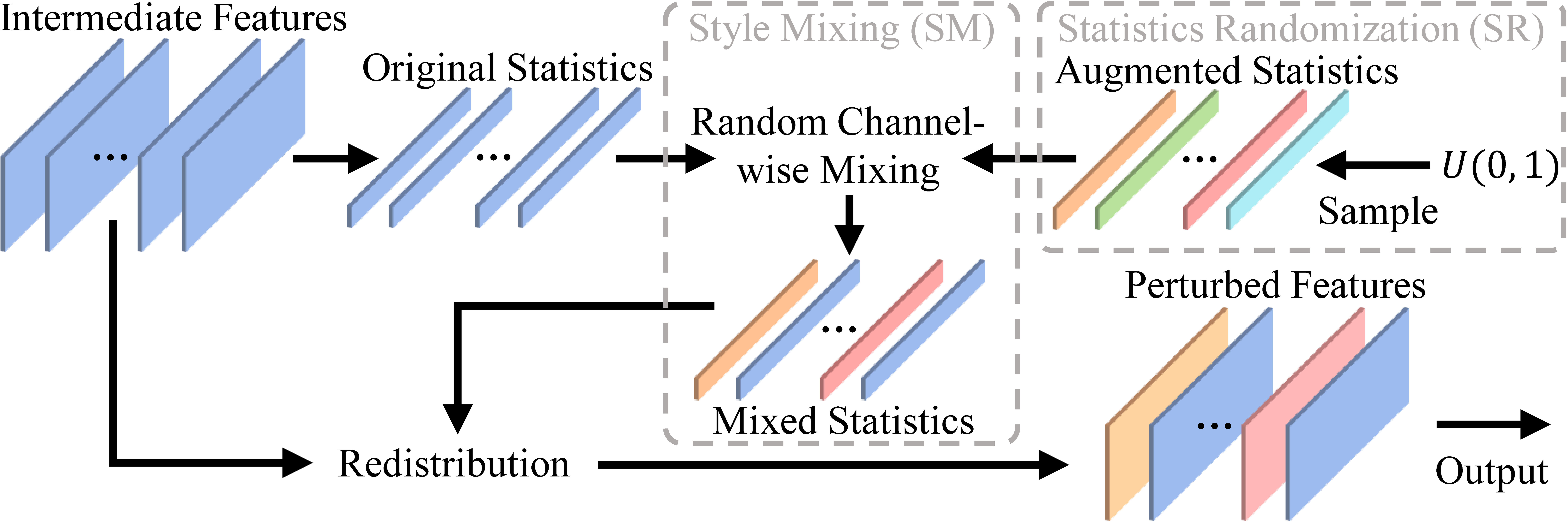
Dies ist die offizielle Pytorch-Implementierung unseres MICCAI 2023-Papiers "Schatz in der Verteilung: eine Domänen-Randomisierungsbasis-Basis-Verallgemeinerung der Multi-Source-Domänen für die Segmentierung von medizinischen Bild von 2D". In diesem Artikel schlagen wir eine Multi-Source-Domänenverallgemeinerungsmethode vor, die als Schatz in Distribution (TRID) bezeichnet wird, um einen beispiellosen Suchraum für die Randomisierung der Feature-Space-Domäne zu erstellen.
Python 3.7
Pytorch 1.8.0
git clone https://github.com/Chen-Ziyang/TriD.git
cd TriD/TriD-master
OD/OC -Segmentierung
Prostata -Segmentierung
Wir nehmen das Szenario als Beispiel mit Binrushed (Zieldomäne) und anderen vier Datensätzen (Quelldomänen) auf.
cd OPTIC
# Training
CUDA_VISIBLE_DEVICES=0 python main.py --mode train_DG --mixstyle_layers layer1 layer2 --random_type TriD --Target_Dataset BinRushed --Source_Dataset Magrabia REFUGE ORIGA Drishti_GS
# Test
CUDA_VISIBLE_DEVICES=0 python main.py --mode single_test --load_time TIME_OF_MODEL --Target_Dataset BinRushed
Wir nehmen das Szenario mit BMC (Zieldomäne) und anderen fünf Datensätzen (Quelldomänen) als Beispiel.
cd PROSTATE
# Training
CUDA_VISIBLE_DEVICES=0 python main.py --mode train_DG --mixstyle_layers layer1 layer2 --random_type TriD --Target_Dataset BMC --Source_Dataset BIDMC HK I2CVB RUNMC UCL
# Test
CUDA_VISIBLE_DEVICES=0 python main.py --mode single_test --load_time TIME_OF_MODEL --Target_Dataset BMC
Ein Teil des Codes wird aus der Pytorch -Implementierung von DOCR überarbeitet.
Wenn Sie dieses Repo für Ihre Forschung nützlich finden, sollten Sie das Papier wie folgt zitieren:
@inproceedings{chen2023treasure,
title={Treasure in distribution: a domain randomization based multi-source domain generalization for 2d medical image segmentation},
author={Chen, Ziyang and Pan, Yongsheng and Ye, Yiwen and Cui, Hengfei and Xia, Yong},
booktitle={International Conference on Medical Image Computing and Computer-Assisted Intervention},
pages={89--99},
year={2023},
organization={Springer}
}