
Bitte beachten Sie, dass der Code von Poseidon gerade an den nächsten Flow übertragen wurde, sodass es möglicherweise noch einige Fehler gibt. Bitte zögern Sie nicht, Probleme zu melden!
Hier präsentieren wir Poseidon, eine Pipeline, um signifikant positive ausgewählte Stellen und mögliche Rekombinationsereignisse in einer Ausrichtung mehrerer Proteinkodierungssequenzen zu erkennen. Standorte, die sich einer positiven Selektion unterziehen , liefern Einblicke in die Evolutionsgeschichte Ihrer Sequenzen, z.
Poseidon basiert auf einer Vielzahl verschiedener Dritter-Tools (siehe unten). Aber keine Sorge, wir haben jedes Tool in seinem eigenen Docker -Container verkapiniert und es im Workflow -Management -System Nextflow verbunden.
Gehen Sie direkt zu einem kleinen Beispiel für das Poseidon-Ausgang für das SARS-CoV-2-Spike-Protein im Vergleich zu einer kürzlich durchgeführten Studie von Zhou et al . 2020.
Sie benötigen nur NextFlow (Version 20.+) und Docker, um die Pipeline auszuführen. Alle Abhängigkeiten werden automatisch gezogen.
Entweder Run Poseidon, indem Sie dieses Repository klonieren:
Git Clone https://github.com/hoelzer/pinidon.gitcd poseidon NEXTFLOW RUN Poseidon.nf -Help
oder lass Nextflow den Zug machen
NEXTFLOW Pull Hoelzer/Poseidon
Wir empfehlen die Verwendung einer bestimmten Freisetzung von Poseidon über
#PULLNEXTFLOW Pull Hoelzer/Poseidon -r v1.0.1#Runnextflow Run Hoelzer/Poseidon -r v1.0.1 -Help
Aktualisieren Sie die Pipeline abhängig von Ihrer Installationsverfahren über git pull oder nextflow pull hoelzer/poseidon .
Wichtig: Poseidon benötigt Nukleotidsequenzen mit einem korrekten offenen Leserahmen als Eingabe. Darüber hinaus hängen die Ergebnisse stark von Ihrer Auswahl von Sequenzen ab. Daher können Sie die Pipeline mehrmals mit unterschiedlichen Stichproben Ihrer Eingangssequenzen ausführen. Auch die Pipeline kann nicht mit zu vielen Sequenzen funktionieren, da in ihrem Kernposeidon Codeml aus der PAML -Suite verwendet, die für> 100 Sequenzen nicht integriert ist. Eine detaillierte Beschreibung der folgenden Eingabeparameter und Einstellungen finden Sie unter.
NEXTFLOW kann leicht in verschiedenen Umgebungen wie Ihrer lokalen Maschine, einem Hochleistungscluster oder der Cloud ausgeführt werden. Unterschiedliche -profile werden verwendet, um den nächsten Flow zu sagen, welches System verwendet werden soll. Für lokale Ausführung -profile local,docker verwendet werden (und ist auch der Standard). Sie können Poseidon auch auf einem HPC mit Singularität über -profile lsf,singularity , -profile slurm,singularity oder -profile sge,singularity leiten. In solchen Fällen können Sie auch in Betracht ziehen, um --cachedir zu zeigen, wo Sie Singularity -Bilder auf Ihrem Cluster speichern können. Der Parameter --workdir ist möglicherweise auch hilfreich, um anpassen, wo temporäre Arbeitsverzeichnisse gespeichert werden sollen (z. B. Verwendung /scratch anstelle von /tmp abhängig von Ihrer HPC -Konfiguration.)
Nehmen wir nun an, dass Sie den nächsten Flow verwendet haben, um den Poseidon -Code zu ziehen, und die Pipeline auf einem lokalen Computer mithilfe des Standardprofils -profile local,docker ausführen.
# Help Nextflow Run Hoelzer/Poseidon anzeigen -HELP # LEGEN SMENDE BEISCHLIESSE auf einem lokalen Computer mit # (zum ersten Mal benötigt dies noch einige Zeit, da die Docker -Container heruntergeladen werden) NEXTFLOW RUN HOELZER/POSEIDON -R V1.0.1 -FASTA ~/.Nextflow/assets/Hoelzer/poseidon/test_data/bats_mx1_small.fasta -CORES 4# Nehmen Sie einen gebrochenen Runnextflow -Lauf Hoelzer/Poseidon -r v1.0.1 --Fasta ~/.Nextflow/Assets/Hoelzer/Poseidon/test_data/bats_mx1_small.fasta -CORES 4 -RESume# anstatt alle verfügbaren Kerne zu verwenden, verwenden nur eine maximale Menge für den lokalen Maschinenextflow -Lauf Hoelzer/Poseidon -r v1.0.1 --Fasta ~/.Nextflow/assets/Hoelzer/poseidon/test_data/batsmall.mall.fastafastafastafastafastafasta.fastafasta.fastafasta.fastafasta. -max_cores 8 -korte 4
Um die positiven Selektionsergebnisse in Fuchs et al . (2017), Journal of Virology Run:
NEXTFLOW RUN HOELZER/POSEIDON -R V1.0.0 -FASTA ~/.NEXTFLOW/ASSETS/Hoelzer/Poseidon/test_data/bats_mx1.fasta -Cores 4-KH-Outgroup "Pteropus_Aalecto, Eidolon_Helvum, Rousettus_aegyptiacus, Hypsignatus_Monstrosus"-Referenz "myotis_daubentonii" "
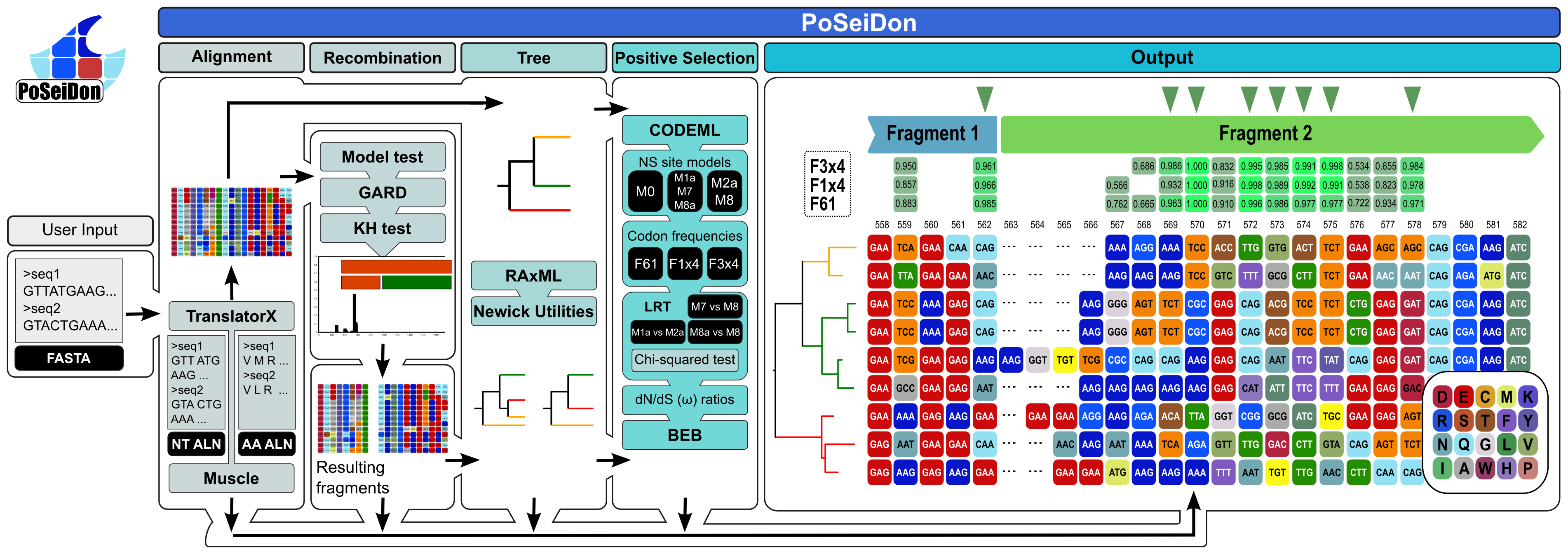
Die Poseidon-Pipeline umfasst die Ausrichtung homologischer Proteinkodiersequenzen, den Nachweis mutmaßlicher Rekombinationsereignisse und evolutionäre Haltepunkte, phylogenetische Rekonstruktionen und den Nachweis positiv ausgewählter Stellen in der vollen Ausrichtung und aller möglichen Fragmente. Schließlich werden alle Ergebnisse auf einer benutzerfreundlichen und löschlichen HTML-Webseite kombiniert und visualisiert. Die resultierenden Ausrichtungsfragmente sind mit farbigen Balken in der HTML -Ausgabe angezeigt.
Translatorx (v1.1), Abascal et al . (2010); 20435676
Muskel (v3.8.31), Edgar (2004); 15034147
Raxml (V8.0.25), Stamatakis (2014); 24451623
Newick Utilities (v1.6), Junier und Zdobnov (2010); 20472542
Modeltest, Posada und Crandall (1998); 9918953
Hyphy (v2.2), Pond et al . (2005); 15509596
Gard, Pond et al. (2006); 17110367
PAML/Codeml (v4.8), Yang (2007); 17483113
Ruby (v2.3.1)
Inkscape (v1.0)
pdftex (v3.14)
Die meisten Poseidon -Parameter sind optional und werden nachstehend im Detail erläutert.
--fasta
Obligatorisch. Ihre Eingabe -Fasta -Datei muss dem Format folgen:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA... >Myotis_davidii Mx1 Gene ATGGCGGTCGAGATAAGATACGTT...
Alle Sequenzen müssen einen korrekten offenen Leserahmen haben, dürfen nur Nukleotidzeichen [a | c | g | t] und kein internes Stop -Codon enthalten.
Sequenz -IDs müssen bis zum ersten Auftreten eines Raums eindeutig sein.
--reference
Optional. Standard: Verwenden Sie die erste Sequenz -ID als Referenz. Sie können eine Arten -ID aus Ihrer Mehrfachfasta -Datei als Referenzart definieren. Positiv ausgewählte Stellen und entsprechende Aminosäuren werden in Bezug auf diese Spezies gezogen. Die ID muss mit dem Fasta -Header übereinstimmen, bis der erste Raum vorliegt. Wenn Sie beispielsweise Myotis lucifugus als Ihre Referenzart möchten und Ihre Fasta -Datei enthält:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA...
verwenden
--reference "Myotis_lucifugus"
als Parameter zum Festlegen der Referenzspezies. Per Standard wird die erste ID in der Mehrfacher Fasta -Datei verwendet.
--outgroup
Optional. Standardeinstellung: Bäume sind ungewurzelt. Sie können eine oder mehrere (Comma getrennte) Arten -IDs als Outgroup definieren. Alle phylogenetischen Bäume werden nach dieser Art verwurzelt. Zum Beispiel, wenn Ihre mehrfache Fasta -Datei enthält
ATGGCGATCGAGATACGATACGTA...
>Myotis_davidii Mx1 Gene
ATGGCGGTCGAGATAAGATACGTT...
>Pteropus_vampyrus Mx1 Gene
ATGGCCGTAGAGATTAGATACTTT...
>Eidolon_helvum Mx1 Gene
ATGCCCGTAGAGAATAGATACTTT...Sie können definieren:
--outgroup "Pteropus_vampyrus,Eidolon_helvum"
Alle Bäume in Bezug auf diese beiden Arten zu verwurzeln.
--kh
Optional. Standard: Falsch. Mit diesem Parameter können Sie entscheiden, ob unbedeutende Haltepunkte berücksichtigt werden sollten. Alle Haltepunkte werden unter Verwendung eines Kashino Hasegawa (KH) -Testkishino, H. und Hasegawa, M. (1989) auf einen signifikanten topologischen Inkongruenz getestet. KH-insidezierende Haltepunkte entstehen am häufigsten aus Variationen der Zweiglängen zwischen den Segmenten. Dennoch könnte es interessant sein, kh-in-signifikante Haltepunkte zu berücksichtigen, da wir bereits mutitativ ausgewählte Stellen in Fragmenten ohne signifikanten topologischen Inkongruenz beobachtet haben. KH-insidesische Fragmente sind in der endgültigen Ausgabe markiert, da sie möglicherweise nicht aus echten Rekombinationsereignissen auftreten.
Pro Standard werden nur signifikante Haltepunkte für weitere Berechnungen verwendet.
Beachten Sie auch, dass die Verwendung auch unbedeutender Haltepunkte die Laufzeit von Poseidon von Minuten auf Stunden verlängern kann, abhängig von der Anzahl der erkannten Haltepunkte.
Bitte beachten Sie die --help für andere Parameter (Gard, Raxml, ...) und teilen Sie uns mit, ob Sie eine weitere Anpassung benötigen!
Wenn Poseidon Ihnen hilft, zitieren Sie bitte:
Martin Hölzer und Manja Marz, "Poseidon: Eine nächste Flow -Pipeline für die Erkennung von Evolutionsrekombinationsereignissen und positive Selektion", OUP Bioinformatics (2020)