Wir freuen uns, Ihnen mitteilen zu können, dass die Indexgröße auf der Festplatte um das 8-fache und im Speicher um viermal gesunken ist von Suffix -Array. Zum Beispiel ist für das menschliche Genom die Indexgröße auf der Festplatte auf ~ 10 GB ab ~ 80 GB und der Speicher Fußabdruck auf ~ 10 GB von ~ 40 GB zurückgegangen. Aufgrund der Reduzierung und kaum leistungsfähigen Auswirkungen auf die Lesezuordnung gibt es eine erhebliche Verringerung der Index -IO -Zeit. Aufgrund dieser Änderung der Indexstruktur (in Commit #4B59796, 10. Oktober 2020) müssen Sie den Index wieder aufbauen.
MC -Flag in der Ausgabe -SAM -Datei in Commit A591E22 hinzugefügt. Die Ausgabe sollte mit der ursprünglichen BWA-MEM-Version 0.7.17 übereinstimmen.
Nach dem Commit E0AC59E haben wir ein Git -Submodul Safestringlib. Um dies zu erhalten, verwenden Sie -rekursiv beim Klonen oder Verwenden von "Git Submodule Init" und "Git Submodule Update" in einem bereits klonierten Repository (siehe unten für weitere Details).
# Vorkompilierte Binärdateien verwenden (empfohlen) curl -l https://github.com/bwa-mem2/bwa-mem2/releases/download/v2.2.1/bwa-mem2-2.2.1_x64-linux.tar.bz2 | Teer Jxf - BWA-MEM2-2.2.1_X64-Linux/BWA-MEM2 INDEX REF.FA BWA-MEM2-2.2.1_X64-Linux/BWA-MEM2 MEM REF.FA Read1.fq Read2.fq> out.sam# kompilieren Sie aus der Quelle (nicht für allgemeine Benutzer empfohlen)# Holen Sie sich den SourceGit-Klon-recursive https: // github.com/bwa-mem2/bwa-mem2cd bwa-mem2# orgit klone https://github.com/bwa-mem2/bwa-mem2cd bwa-mem2 Git Submodule init Git Submodule Update# Compile und Runmake ./BWA-MEM2
Das Werkzeug BWA-MEM2 ist die nächste Version des BWA-MEM-Algorithmus in BWA. Es erzeugt eine Ausrichtung mit BWA und beträgt ~ 1,3-3.1x schneller, je nach Anwendungsfall, Datensatz und laufender Maschine.
Die ursprüngliche BWA wurde von Heng Li (@lh3) entwickelt. Die Leistungsverbesserung in BWA-MEM2 wurde hauptsächlich von Vasimuddin MD (@YUK12) und Sanchit Misra (@Sanchit-Misra) von Parallel Computing Lab, Intel, durchgeführt. BWA-MEM2 wird unter der MIT-Lizenz verteilt.
Für allgemeine Benutzer wird empfohlen, die vorkompilierten Binärdateien auf der Release -Seite zu verwenden. Diese Binärdateien wurden mit dem Intel-Compiler zusammengestellt und läuft schneller als GCC-kompilierte Binärdateien. Die vorkompilierten Binärdateien unterstützen auch indirekt den CPU -Versand. Die bwa-mem2 Binärin kann automatisch die effizienteste Implementierung basierend auf dem SIMD-Befehlssatz auswählen, der auf der laufenden Maschine verfügbar ist. Vorkompilierte Binärdateien wurden auf einer CentOS7 -Maschine unter Verwendung der folgenden Befehlszeile erzeugt:
Machen Sie CXX = ICPC Multi
Die Verwendung ist genau das gleiche wie das ursprüngliche BWA -MEM -Werkzeug. Hier sind kurze Synops. Ausführen ./BWA-MEM2 für verfügbare Befehle.
# Indexierung der Referenzsequenz (erfordert 28N GB Speicher, wobei n die Größe der Referenzsequenz ist). <In.fasta> ist der Pfad zur Referenzsequenz Fasta -Datei und <prefix> ist das Präfix der Namen der Dateien, die den resultierenden Index speichern. Standard ist in.fasta. # Mapping # run "./bwa-mem2 mem", um alle Optionen zu erhalten Wobei <prefix> das Präfix ist, das beim Erstellen des Index oder des Pfades zur Referenz -Fasta -Datei angegeben ist, falls kein Präfix bereitgestellt wurde.
Datensätze:
Referenzgenom: Human_g1k_v37.fasta
| Alias | Datensatzquelle | Anzahl der Lesevorgänge | Lesen Sie Länge |
|---|---|---|---|
| D1 | Breites Institut | 2 x 2,5 m bp | 151bp |
| D2 | SRA: SRR7733443 | 2 x 2,5 m bp | 151bp |
| D3 | SRA: SRR9932168 | 2 x 2,5 m bp | 151bp |
| D4 | SRA: SRX6999918 | 2 x 2,5 m bp | 151bp |
Maschinendetails:
Prozessor: Intel (R) Xeon (R) 8280 CPU @ 2,70 GHz
OS: CentOS Linux Release 7.6.1810
Speicher: 100 GB
Wir befolgten die folgenden Schritte, um die Leistungsergebnisse zu sammeln:
A. Daten herunterladen Schritte:
Laden Sie das SRA-Toolkit von https://trace.ncbi.nlm.nih.gov/traces/sra/sra.cgi?view=software#header-global herunter
tar xfzv sratoolkit.2.10.5-centos_linux64.tar.gz
Download D2: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump-split-files srr7733443
Laden Sie D3 herunter:
Laden Sie D4 herunter:
B. Ausrichtungsschritte:
Git Clone https://github.com/bwa-mem2/bwa-mem2.git
CD BWA-MEM2
make CXX=icpc (mit Intel C/C ++ - Compiler)
oder make (mit GCC -Compiler)
./BWA-MEM2 INDEX <REF.FA>
./bwa-mem2 mem [-t <#Threads>] <ref.fa> <in_1.fastq> [<in_2.fastq>]> <output.sam>
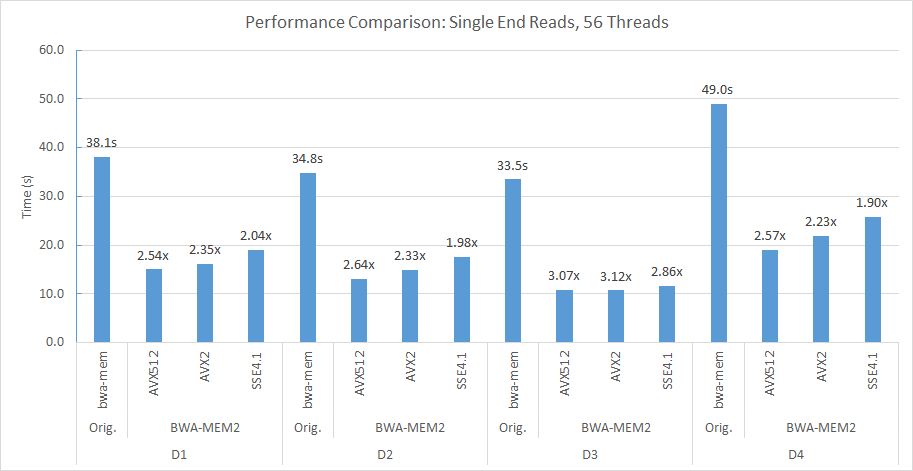
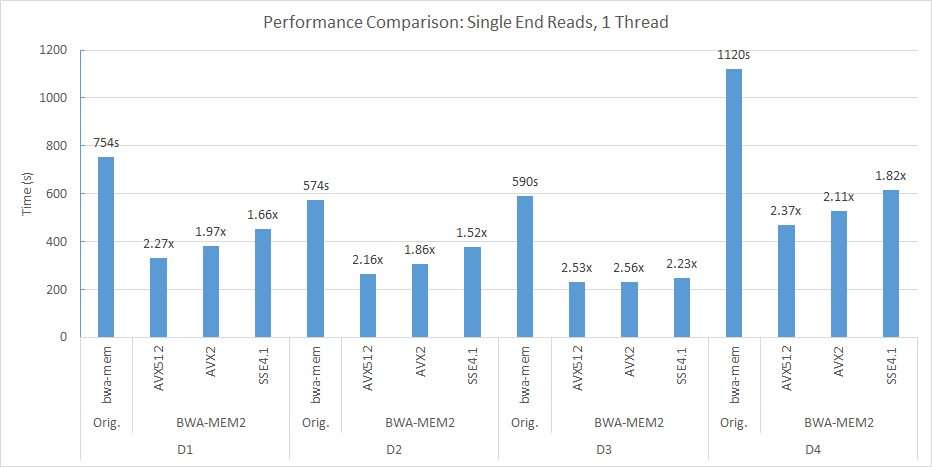
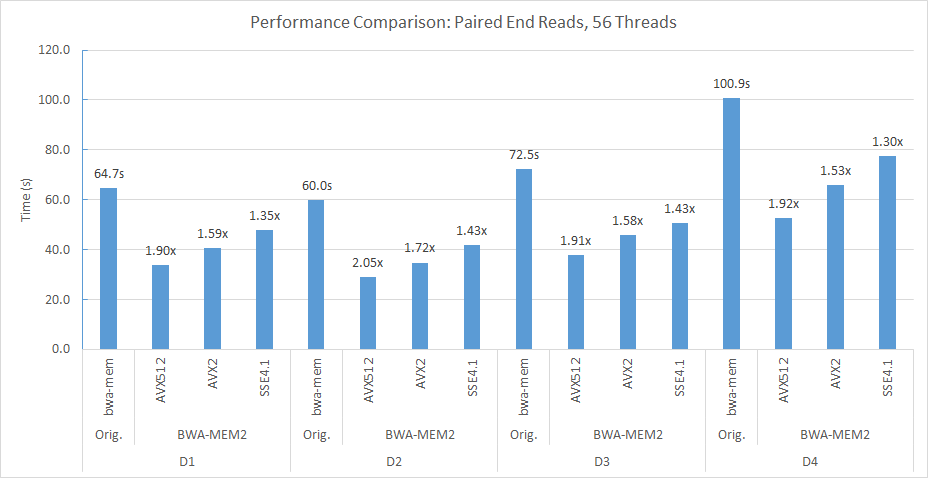
In unserer Doppelsteck (jeweils 56 Threads) und Double Numa Compute -Knoten haben wir beispielsweise die folgende Befehlszeile verwendet, um D2 auf Human_G1K_V37.Fasta Referenzgenom auszurichten.
numactl -m 0 -C 0-27,56-83 ./bwa-mem2 index human_g1k_v37.fasta numactl -m 0 -C 0-27,56-83 ./bwa-mem2 mem -t 56 human_g1k_v37.fasta SRR7733443_1.fastq SRR7733443_2.fastq > d2_align.sam

BWA-MEM2-LISA ist eine beschleunigte Version von BWA-MEM2, in der wir gelernte Indexes in die Seeding-Phase anwenden. BWA-MEM2-LISA-Zweig enthält den Quellcode der Implementierung. Im Folgenden finden Sie die Merkmale von BWA-MEM2-LISA:
Genau die gleiche Ausgabe wie BWA-MEM2.
Alle Befehlslinien zum Erstellen eines Index und der Lesezuordnung sind genauso wie BWA-MEM2.
BWA-MEM2-LISA beschleunigt die Seeding-Phase (einer der Haupttipps in BWA-MEM2) um bis zu 4,5x im Vergleich zu BWA-MEM2.
Der Gedächtnisfußabdruck des BWA-MEM2-LISA-Index beträgt ~ 120 GB für menschliches Genom.
Der Code ist in BWA-MEM2-lisa Branch vorhanden: https://github.com/bwa-mem2/bwa-mem2/tree/bwa-mem2-lisa
Der ERT-Zweig von BWA-MEM2-Repository enthält eine Codebasis von Enterated Radix-Baumbasis von BWA-MEM2. Der ERT-Code ist auf BWA-MEM2 aufgebaut (dank der harten Arbeit von @arun-sub). Im Folgenden finden Sie die Highlights des ERT-basierten BWA-MEM2-Tools:
Exakt gleiche Ausgabe wie BWA-MEM (2)
Das Tool verfügt über zwei zusätzliche Flags, um die Verwendung der ERT-Lösung (für die Erstellung und Mapping in der Index) zu ermöglichen.
Es verwendet 1 zusätzliches Flag, um den ERT-Index (anders als BWA-MEM2-Index) und 1 zusätzliches Flag für die Verwendung dieses ERT-Index (siehe ReadMe of ERT-Zweig) zu erstellen.
Die ERT -Lösung ist 10% -30% schneller (getestet an der oben genannten Maschinenkonfiguration) im Vergleich zu Vanilla bwa -MEM2 -Benutzer werden empfohlen, Option -K 1000000 zu verwenden, um die Beschleunigungen anzuzeigen
Der Speicherfußdruck des ERT -Index beträgt ~ 60 GB
Der Code ist in der ERT-Filiale vorhanden: https://github.com/bwa-mem2/bwa-mem2/tree/ert
Vasimuddin MD, Sanchit Misra, Heng Li, Srinivas Aluru. Effiziente Architekturbeschleunigung von BWA-MEM für Multicore-Systeme. IEEE Parallel und verteiltes Verarbeitungssymposium (IPDPS), 2019. 10.1109/IPDPs.2019.00041