Eine R -Schnittstelle zur Meme Suite -Familie von Tools, die mehrere Dienstprogramme zur Durchführung der Motivanalyse zu DNA-, RNA- und Proteinsequenzen bietet. Memes arbeitet, indem er eine lokale Installation der Meme -Suite erfasst, die Befehle ausgeführt und dann die Ergebnisse direkt in R importiert.
Memes ist derzeit in der Bioconductor devel Branch erhältlich:
if ( ! requireNamespace( " BiocManager " , quietly = TRUE ))
install.packages( " BiocManager " )
# The following initializes usage of Bioc devel
BiocManager :: install( version = ' devel ' )
BiocManager :: install( " memes " )Sie können die Entwicklungsversion von Memes aus GitHub installieren mit:
if ( ! requireNamespace( " remotes " , quietly = TRUE ))
install.packages( " remotes " )
remotes :: install_github( " snystrom/memes " )
# To temporarily bypass the R version 4.1 requirement, you can pull from the following branch:
remotes :: install_github( " snystrom/memes " , ref = " no-r-4 " ) # Get development version from dockerhub
docker pull snystrom/memes_docker:devel
# the -v flag is used to mount an analysis directory,
# it can be excluded for demo purposes
docker run -e PASSWORD= < password > -p 8787:8787 -v < path > / < to > / < project > :/mnt/ < project > snystrom/memes_docker:develMemes stützt sich auf eine lokale Installation der Meme -Suite. Für Installationsanweisungen für die MEME -Suite finden Sie im Installationshandbuch der Meme Suite.
Memes muss den Standort des meme/bin/ Verzeichnisses auf Ihrer lokalen Maschine kennen. Sie können Memes den Ort Ihrer Meme -Suite auf 4 Arten erkennen. Memes bevorzugen immer die spezifischere Definition, wenn es sich um einen gültigen Pfad handelt. Hier werden sie von den meisten bis am wenigsten spezifisch eingestuft:
meme_path aller Memes -Funktionen übergebenoptions(meme_bin = "/path/to/meme/bin/") in Ihrem R -Skript einMEME_BIN=/path/to/meme/bin/ in Ihrer .Renviron -Datei~/meme/bin/Wenn Memes Ihre Installation nicht an der angegebenen Stelle erfasst, fällt sie auf die nächste Option zurück.
Um zu überprüfen, ob Memes Ihre Meme -Installation erkennen können, verwenden Sie check_meme_install() , bei dem die obige Suche nach einer gültigen Meme -Installation verwendet wird. Es wird berichtet, ob Werkzeuge fehlen, und den Pfad zu Meme ausdrucken, den es sieht. Dies kann nützlich sein, um Probleme mit Ihrer Installation zu beheben.
library( memes )
# Verify that memes detects your meme install
# (returns all green checks if so)
check_meme_install()
# > checking main install
# > ✓ /opt/meme/bin
# > checking util installs
# > ✓ /opt/meme/bin/dreme
# > ✓ /opt/meme/bin/ame
# > ✓ /opt/meme/bin/fimo
# > ✓ /opt/meme/bin/tomtom
# > ✓ /opt/meme/bin/meme
# > x /opt/meme/bin/streme # You can manually input a path to meme_path
# If no meme/bin is detected, will return a red X
check_meme_install( meme_path = ' bad/path ' )
# > checking main install
# > x bad/path | Funktionsname | Verwenden | Sequenzeingabe | Motiveingabe | Ausgabe |
|---|---|---|---|---|
runStreme() | Motiv -Entdeckung (kurze Motive) | Ja | NEIN | universalmotif_df |
runDreme() | Motiv -Entdeckung (kurze Motive) | Ja | NEIN | universalmotif_df |
runAme() | Motivanreicherung | Ja | Ja | Data.Frame (Optional: sequences Spalte) |
runFimo() | Motivscannen | Ja | Ja | Grang von Motivpositionen |
runTomTom() | Motivvergleich | NEIN | Ja | universalmotif_df mit best_match_motif und tomtom -Spalten* |
runMeme() | Motiv -Entdeckung (lange Motive) | Ja | NEIN | universalmotif_df |
* Hinweis: Wenn runTomTom() unter Verwendung eines universalmotif_df ausgeführt wird, werden die Ergebnisse mit den Ergebnissen universalmotif_df als zusätzliche Spalten verbunden. Dies ermöglicht einen einfachen Vergleich von De-Novo- entdeckten Motiven mit ihren Übereinstimmungen.
Sequenzeingänge können eines von: sein:
Biostrings::XStringSet (kann aus Granges mit get_sequence() Helfer -Funktion erzeugt werden)Biostrings::XStringSet -Objekten (generiert von get_sequence() )Motiveingänge können eines der:
universalmotif -Objekt oder eine Liste von universalmotif -ObjektenrunDreme() -Ergebungsobjekt (dies ermöglicht die Ergebnisse von runDreme() direkt an runTomTom() ))list() (z list("path/to/database.meme", "dreme_results" = dreme_res)Ausgangstypen :
runDreme() , runStreme() , runMeme() und runTomTom() Return universalmotif_df -Objekte, die Daten sind, die mit speziellen Spalten sind. Die motif -Spalte enthält ein universalmotif -Objekt mit 1 Eintrag pro Zeile. Die verbleibenden Spalten beschreiben die Eigenschaften jedes zurückgegebenen Motivs. Die folgenden Spaltennamen sind insofern von Bedeutung, als ihre Werte beim Ausführen update_motifs() und to_list() verwendet werden, um die Eigenschaften der in der motif -Spalte gespeicherten Motive zu ändern. Achten Sie darauf, diese Werte zu ändern, da diese Änderungen beim Aufrufen von update_motifs() oder to_list() in die motif -Spalte ausbreiten.
Memes basiert auf dem UniversalMotif -Paket universalmotif das ein Rahmen für die Manipulation von Motiven in R. universalmotif_df to_df() to_list() Dies ermöglicht die Verwendung von memes -Ergebnissen mit allen anderen Biokonduktor -Motiv -Paketen, da universalmotif -Objekte mit convert_motifs() in einen anderen Motivtyp konvertieren können.
runTomTom() gibt eine spezielle Spalte zurück: tomtom , die ein data.frame ist. Rahmen aller Übereinstimmungsdaten für jedes Eingabemotiv. Dies kann mit tidyr::unnest(tomtom_results, "tomtom") erweitert und mit nest_tomtom() renestiert werden. Die von runTomTom() zurückgegebenen Spalten best_match_ geben Werte für das Motiv an, was die beste Übereinstimmung mit dem Eingangsmotiv entsprach.
suppressPackageStartupMessages(library( magrittr ))
suppressPackageStartupMessages(library( GenomicRanges ))
# Example transcription factor peaks as GRanges
data( " example_peaks " , package = " memes " )
# Genome object
dm.genome <- BSgenome.Dmelanogaster.UCSC.dm6 :: BSgenome.Dmelanogaster.UCSC.dm6 Die Funktion get_sequence nimmt eine GRanges oder GRangesList als Eingabe und gibt die Sequenzen als BioStrings::XStringSet oder Liste der XStringSet -Objekte zurück. get_sequence nennt jeden Fasta -Eintrag durch die genomischen Koordinaten. Jede Sequenz stammt aus.
# Generate sequences from 200bp about the center of my peaks of interest
sequences <- example_peaks % > %
resize( 200 , " center " ) % > %
get_sequence( dm.genome ) runDreme() akzeptiert Xstringset oder einen Pfad zu einer Fasta -Datei als Eingabe. Sie können andere Sequenzen oder gemischte Eingangssequenzen als Steuerdatensatz verwenden.
# runDreme accepts all arguments that the commandline version of dreme accepts
# here I set e = 50 to detect motifs in the limited example peak list
# In a real analysis, e should typically be < 1
dreme_results <- runDreme( sequences , control = " shuffle " , e = 50 ) Memes basiert auf dem UniversalMotif -Paket. Die Ergebnisse werden im Format universalmotif_df zurückgegeben, bei dem es sich um ein R -Daten handelt. Der Rahmen kann mit to_list() nahtlos zwischen Daten und universalmotif -Format miteinander verbunden sein, um in das Listenformat universalmotif -Listen zu konvertieren, und to_df() um wieder in das Data.Frame -Format zu konvertieren. Mit to_list() ermöglicht die Verwendung von memes -Ergebnissen mit allen universalmotif -Funktionen wie SO:
library( universalmotif )
dreme_results % > %
to_list() % > %
view_motifs()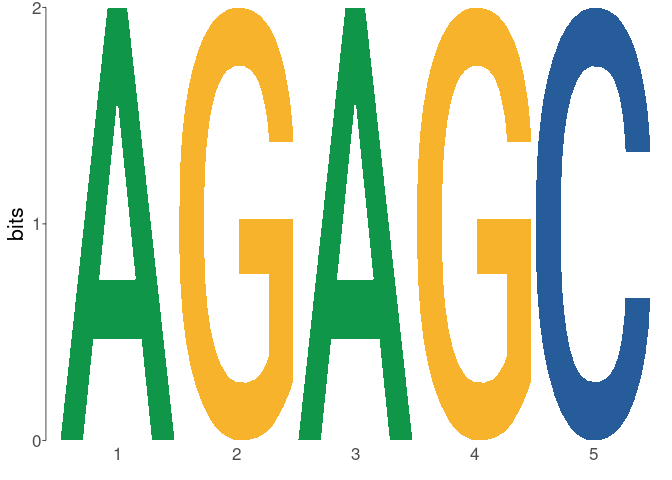
Entdeckte Motive können an bekannte TF -Motive unter Verwendung von runTomTom() angepasst werden, die als Eingabe einen Pfad zu einer .MEME -formatierten Datei, einer universalmotif -Liste oder den Ergebnissen von runDreme() akzeptieren können.
TomTom verwendet eine Datenbank mit bekannten Motiven, die an den database als Pfad zu einer .meme -Formatdatei oder als universalmotif -Objekt übergeben werden kann.
Optional können Sie die Umgebungsvariable MEME_DB in .Renviron auf eine Datei auf der Festplatte festlegen, oder der meme_db -Wert in options in eine gültige .MEME -Formatdatei und Meme verwendet diese Datei als Datenbank. Memes bevorzugen immer die Benutzereingabe für den Funktionsaufruf über eine globale variable Einstellung.
options( meme_db = system.file( " extdata/flyFactorSurvey_cleaned.meme " , package = " memes " ))
m <- create_motif( " CMATTACN " , altname = " testMotif " )
tomtom_results <- runTomTom( m ) tomtom_results
# > motif name altname consensus alphabet strand icscore type
# > 1 <mot:motif> motif testMotif CMATTACN DNA +- 13 PPM
# > bkg best_match_name best_match_altname
# > 1 0.25, 0.25, 0.25, 0.25 prd_FlyReg prd
# > best_db_name best_match_offset best_match_pval best_match_eval
# > 1 flyFactorSurvey_cleaned 0 9.36e-05 0.052
# > best_match_qval best_match_strand
# > 1 0.0353 +
# > best_match_motif
# > 1 <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>
#> tomtom
#> 1 prd_FlyReg, tup_SOLEXA_10, CG13424_Cell, CG11085_Cell, BH2_Cell, CG13424_SOLEXA_2, Tup_Cell, Tup_SOLEXA, Bsh_Cell, Exex_SOLEXA, Odsh_SOLEXA, Unc4_Cell, Ubx_FlyReg, Unc4_SOLEXA, E5_Cell, inv_SOLEXA_5, BH2_SOLEXA, Zen_SOLEXA, CG33980_SOLEXA_2_10, BH1_SOLEXA, CG33980_SOLEXA_2_0, Hgtx_Cell, NK7.1_Cell, Slou_Cell, CG13424_SOLEXA, Zen2_Cell, AbdA_SOLEXA, Antp_SOLEXA, Btn_Cell, Dfd_SOLEXA, Eve_SOLEXA, Ftz_Cell, Hmx_SOLEXA, Hmx_Cell, CG34031_Cell, zen2_SOLEXA_2, En_Cell, Pb_SOLEXA, Slou_SOLEXA, Unpg_Cell, inv_SOLEXA_2, ovo_FlyReg, lim_SOLEXA_2, C15_SOLEXA, Ems_Cell, Btn_SOLEXA, Unpg_SOLEXA, Pb_Cell, Bsh_SOLEXA, Scr_SOLEXA, Zen2_SOLEXA, CG34031_SOLEXA, Eve_Cell, Pph13_Cell, BH1_Cell, CG11085_SOLEXA, CG32532_Cell, en_FlyReg, Dll_SOLEXA, Dfd_Cell, Dr_SOLEXA, Ap_Cell, Ro_Cell, CG4136_SOLEXA, CG33980_SOLEXA, Hbn_SOLEXA, Lbl_Cell, Otp_Cell, Rx_Cell, CG32532_SOLEXA, NK7.1_SOLEXA, Dr_Cell, Odsh_Cell, Al_SOLEXA, Antp_Cell, Hgtx_SOLEXA, Ftz_SOLEXA, Lab_SOLEXA, Dfd_FlyReg, Ap_SOLEXA, Awh_SOLEXA, CG11294_SOLEXA, CG4136_Cell, E5_SOLEXA, Ro_SOLEXA, PhdP_SOLEXA, CG12361_SOLEXA_2, Ind_Cell, Scr_Cell, CG9876_Cell, CG18599_Cell, CG9876_SOLEXA, Otp_SOLEXA, Lbl_SOLEXA, Ubx_Cell, Ubx_SOLEXA, en_SOLEXA_2, Pph13_SOLEXA, Rx_SOLEXA, CG15696_SOLEXA, CG18599_SOLEXA, Ems_SOLEXA, Repo_Cell, Dll_Cell, C15_Cell, CG12361_SOLEXA, Abd-A_FlyReg, Repo_SOLEXA, Zen_Cell, Inv_Cell, En_SOLEXA, Lim3_Cell, Lim1_SOLEXA, CG15696_Cell, Crc_CG6272_SANGER_5, Lab_Cell, CG32105_SOLEXA, Bap_SOLEXA, CG9437_SANGER_5, AbdA_Cell, pho_FlyReg, CG33980_Cell, Cad_SOLEXA, CG4328_SOLEXA, CG4328_Cell, Gsc_Cell, vri_SANGER_5, AbdB_SOLEXA, Xrp1_CG6272_SANGER_5, Al_Cell, Exex_Cell, br-Z4_FlyReg, CG11294_Cell, Aef1_FlyReg, CG7745_SANGER_5, PhdP_Cell, Awh_Cell, prd, tup, lms, CG11085, B-H2, lms, tup, tup, bsh, exex, OdsH, unc-4, Ubx, unc-4, E5, inv, B-H2, zen, CG33980, B-H1, CG33980, HGTX, NK7.1, slou, lms, zen2, abd-A, Antp, btn, Dfd, eve, ftz, Hmx, Hmx, CG34031, zen2, en, pb, slou, unpg, inv, ovo, Lim1, C15, ems, btn, unpg, pb, bsh, Scr, zen2, CG34031, eve, Pph13, B-H1, CG11085, CG32532, en, Dll, Dfd, Dr, ap, ro, CG4136, CG33980, hbn, lbl, otp, Rx, CG32532, NK7.1, Dr, OdsH, al, Antp, HGTX, ftz, lab, Dfd, ap, Awh, CG11294, CG4136, E5, ro, PHDP, CG12361, ind, Scr, CG9876, CG18599, CG9876, otp, lbl, Ubx, Ubx, en, Pph13, Rx, CG15696, CG18599, ems, repo, Dll, C15, CG12361, abd-A, repo, zen, inv, en, Lim3, Lim1, CG15696, crc, lab, CG32105, bap, CG9437, abd-A, pho, CG33980, cad, CG4328, CG4328, Gsc, vri, Abd-B, Xrp1, al, exex, br, CG11294, Aef1, CG7745, PHDP, Awh, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, 0, 1, 0, 1, 1, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 3, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, -1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, -2, 0, 0, 0, -3, 0, 4, 1, 0, 0, 0, 0, -2, 0, -2, 1, 0, -2, 1, 0, 2, -1, 0, 9.36e-05, 0.000129, 0.00013, 0.000194, 0.000246, 0.000247, 0.000319, 0.000364, 0.000427, 0.000435, 0.000473, 0.000473, 0.000556, 0.000643, 0.000694, 0.000728, 0.000748, 0.000748, 0.000785, 0.000828, 0.000849, 0.000868, 0.000868, 0.000868, 0.00102, 0.00113, 0.00116, 0.00116, 0.00116, 0.00116, 0.00116, 0.00116, 0.00118, 0.00126, 0.00134, 0.00141, 0.00144, 0.00145, 0.00145, 0.00154, 0.00161, 0.00165, 0.00173, 0.00177, 0.00177, 0.00179, 0.00179, 0.0019, 0.00191, 0.00191, 0.00191, 0.00203, 0.00203, 0.00203, 0.00212, 0.00214, 0.00219, 0.00232, 0.00242, 0.00247, 0.0026, 0.00264, 0.00264, 0.00268, 0.00282, 0.00282, 0.00282, 0.00282, 0.00282, 0.00286, 0.00286, 0.00296, 0.00305, 0.00316, 0.0032, 0.0032, 0.00326, 0.00326, 0.00341, 0.00348, 0.00348, 0.00348, 0.00348, 0.00348, 0.00348, 0.0035, 0.00359, 0.00359, 0.00359, 0.00364, 0.00372, 0.00372, 0.00372, 0.00387, 0.00387, 0.00412, 0.00415, 0.00424, 0.00424, 0.00439, 0.00452, 0.00452, 0.00467, 0.00483, 0.00497, 0.00497, 0.00528, 0.00549, 0.0059, 0.00597, 0.00624, 0.00635, 0.00709, 0.00761, 0.0079, 0.00856, 0.00859, 0.00912, 0.00935, 0.0097, 0.00974, 0.0101, 0.0109, 0.0116, 0.0123, 0.0123, 0.0127, 0.0138, 0.0138, 0.014, 0.014, 0.0147, 0.0148, 0.0155, 0.0165, 0.0166, 0.0177, 0.052, 0.0718, 0.0725, 0.108, 0.137, 0.137, 0.177, 0.202, 0.238, 0.242, 0.263, 0.263, 0.309, 0.357, 0.386, 0.405, 0.416, 0.416, 0.436, 0.46, 0.472, 0.483, 0.483, 0.483, 0.566, 0.629, 0.646, 0.646, 0.646, 0.646, 0.646, 0.646, 0.654, 0.702, 0.745, 0.785, 0.8, 0.808, 0.808, 0.858, 0.897, 0.92, 0.961, 0.985, 0.985, 0.993, 0.993, 1.05, 1.06, 1.06, 1.06, 1.13, 1.13, 1.13, 1.18, 1.19, 1.22, 1.29, 1.34, 1.38, 1.45, 1.47, 1.47, 1.49, 1.57, 1.57, 1.57, 1.57, 1.57, 1.59, 1.59, 1.65, 1.7, 1.76, 1.78, 1.78, 1.81, 1.81, 1.9, 1.94, 1.94, 1.94, 1.94, 1.94, 1.94, 1.95, 2, 2, 2, 2.02, 2.07, 2.07, 2.07, 2.15, 2.15, 2.29, 2.31, 2.36, 2.36, 2.44, 2.52, 2.52, 2.6, 2.68, 2.76, 2.76, 2.94, 3.05, 3.28, 3.32, 3.47, 3.53, 3.94, 4.23, 4.39, 4.76, 4.77, 5.07, 5.2, 5.39, 5.41, 5.61, 6.06, 6.42, 6.81, 6.81, 7.03, 7.66, 7.65, 7.78, 7.78, 8.15, 8.26, 8.59, 9.18, 9.2, 9.85, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0368, 0.0369, 0.0369, 0.0369, 0.0369, 0.0369, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0379, 0.0379, 0.0381, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0404, 0.0404, 0.0424, 0.0424, 0.0424, 0.0424, 0.0435, 0.0439, 0.0439, 0.0449, 0.046, 0.0464, 0.0464, 0.0489, 0.0504, 0.0536, 0.0538, 0.0557, 0.0562, 0.0622, 0.0662, 0.0681, 0.0727, 0.0727, 0.0765, 0.0778, 0.0797, 0.0797, 0.0819, 0.0877, 0.0923, 0.0964, 0.0964, 0.0987, 0.106, 0.106, 0.106, 0.106, 0.11, 0.111, 0.114, 0.121, 0.121, 0.128, +, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, +, -, -, -, +, -, -, -, -, -, -, -, +, -, +, -, -, -, +, +, +, -, -
# >
# > [Hidden empty columns: family, organism, nsites, bkgsites, pval, qval,
# > eval.] runTomTom() fügt seine Ergebnisse als Spalten zu einem runDreme() -Ergebnisdaten.Frame hinzu.
full_results <- dreme_results % > %
runTomTom() AME wird verwendet, um die Anreicherung bekannter Motive in Zielsequenzen zu testen. runAme() verwendet den MEME_DB -Eintrag in .Renviron oder options(meme_db = "path/to/database.meme") als Motiv -Datenbank. Alternativ akzeptiert es alle gültigen Eingaben ähnlich wie runTomTom() .
# here I set the evalue_report_threshold = 30 to detect motifs in the limited example sequences
# In a real analysis, evalue_report_threshold should be carefully selected
ame_results <- runAme( sequences , control = " shuffle " , evalue_report_threshold = 30 )
ame_results
# > # A tibble: 2 x 17
# > rank motif_db motif_id motif_alt_id consensus pvalue adj.pvalue evalue tests
# > <int> <chr> <chr> <chr> <chr> <dbl> <dbl> <dbl> <int>
# > 1 1 /usr/lo… Eip93F_… Eip93F ACWSCCRA… 5.14e-4 0.0339 18.8 67
# > 2 2 /usr/lo… Cf2-PB_… Cf2 CSSHNKDT… 1.57e-3 0.0415 23.1 27
# > # … with 8 more variables: fasta_max <dbl>, pos <int>, neg <int>,
# > # pwm_min <dbl>, tp <int>, tp_percent <dbl>, fp <int>, fp_percent <dbl> view_tomtom_hits ermöglicht den Vergleich der Eingangsmotive mit den oberen Treffern von TomTom. Die manuelle Überprüfung dieser Übereinstimmungen ist wichtig, da manchmal die obere Übereinstimmung nicht immer die richtige Aufgabe ist. Durch das Ändern von top_n können Sie zusätzliche Übereinstimmungen in absteigender Reihenfolge ihres Ranges anzeigen.
full_results % > %
view_tomtom_hits( top_n = 1 )
# > $m01_AGAGC 
Es kann nützlich sein, die Ergebnisse von runAme() als Heatmap anzusehen. plot_ame_heatmap() kann komplexe Visualisierungen zur Analyse der Anreicherung zwischen verschiedenen Regionstypen erstellen (Einzelheiten siehe Vignetten). Hier ist eine einfache Beispiel -Heatmap.
ame_results % > %
plot_ame_heatmap()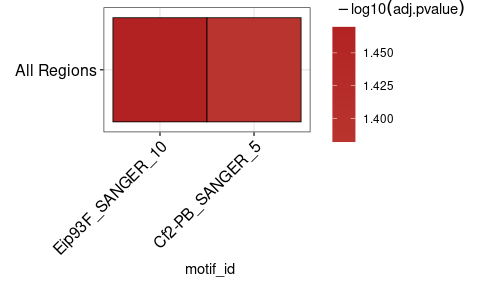
Das FIMO -Tool wird verwendet, um Übereinstimmungen an bekannte Motive zu identifizieren. runFimo gibt diese Treffer als GRanges -Objekt zurück, das die genomischen Koordinaten des Motiv -Matchs enthält.
# Query MotifDb for a motif
e93_motif <- MotifDb :: query( MotifDb :: MotifDb , " Eip93F " ) % > %
universalmotif :: convert_motifs()
# > See system.file("LICENSE", package="MotifDb") for use restrictions.
# Scan for the E93 motif within given sequences
fimo_results <- runFimo( sequences , e93_motif , thresh = 1e-3 )
# Visualize the sequences matching the E93 motif
plot_sequence_heatmap( fimo_results $ matched_sequence ) 
MEMEs unterstützt auch das Import von Ergebnissen, die mit der Meme-Suite außerhalb von R erzeugt werden (z. B. Aufträge auf meme-suite.org oder auf der Befehlszeile ausführen). Dies ermöglicht die Verwendung bereits bestehender Meme -Suite -Ergebnisse mit nachgeschalteten Memes -Funktionen.
| Meme -Werkzeug | Funktionsname | Dateityp |
|---|---|---|
| Streme | importStremeXML() | Streme.xml |
| Dreme | importDremeXML() | dreme.xml |
| Tomtom | importTomTomXML() | tomtom.xml |
| AME | importAme() | AME.TSV* |
| Fimo | importFimo() | fimo.tsv |
| Meme | importMeme() | meme.txt |
* importAME() kann auch die Ausgabe "Sequenzen.tsv" verwenden, wenn AME verwendet wird method = "fisher" , dies ist optional.
Die MEME -Suite unterstützt derzeit keine Windows, kann jedoch unter Cygwin oder dem Windows Linux Subsytem (WSL) installiert werden. Bitte beachten Sie, dass Sie, wenn Meme auf Cygwin oder WSL installiert ist, auch R in Cygwin oder WSL ausführen müssen, um MEMEs zu verwenden.
Eine alternative Lösung besteht darin, Docker zu verwenden, um eine virtuelle Umgebung mit installierter Meme -Suite auszuführen. Wir bieten einen Memes Docker -Container an
Das wird mit der Meme-Suite, dem R-Studio und allen memes Abhängigkeiten vorinstalliert.
Memes ist ein Wrapper für einige ausgewählte Tools aus der Meme -Suite, die von einer anderen Gruppe entwickelt wurden. Zusätzlich zum Zitieren von Memes zitieren bitte die MEME -Suite -Tools, die den von Ihnen verwendeten Werkzeugen entsprechen.
Wenn Sie runDreme() in Ihrer Analyse verwenden, zitieren Sie bitte:
Timothy L. Bailey, „Dreme: Motiv-Entdeckung in Transkriptionsfaktor-Chip-Seq-Daten“, Bioinformatik, 27 (12): 1653-1659, 2011. Volltext
Wenn Sie runTomTom() in Ihrer Analyse verwenden, zitieren Sie bitte:
Shobhit Gupta, Ja Stamatoyannopolous, Timothy Bailey und William Stafford Noble, „Ähnlichkeit zwischen Motiven“, Genome Biology, 8 (2): R24, 2007. Volltext
Wenn Sie runAme() in Ihrer Analyse verwenden, zitieren Sie bitte:
Robert McLeay und Timothy L. Bailey, „Motivanreicherungsanalyse: Ein einheitliches Gerüst und Methodenbewertung“, BMC Bioinformatics, 11: 165, 2010, doi: 10.1186/1471-2105-11-165. Volltext
Wenn Sie runFimo() in Ihrer Analyse verwenden, zitieren Sie bitte:
Charles E. Grant, Timothy L. Bailey und William Stafford Noble, „Fimo: Scannen nach Vorkommen eines bestimmten Motivs“, Bioinformatics, 27 (7): 1017-1018, 2011. Volltext
Die MEME-Suite ist kostenlos für gemeinnützige Verwendung, aber gewinnorientierte Benutzer sollten eine Lizenz erwerben. Weitere Informationen finden Sie auf der Meme Suite Copyright -Seite.