
Isling ist ein Instrument zur Erkennung der Virus- oder Vektorintegration in Paired-End-Lesevorgängen. Bitte lesen Sie unser Papier für alle Details.
Wenn Sie conda und snakemake installiert haben, werden Sie mit den (enthaltenen) Testdaten lokal ausgeführt:
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
Wenn Sie snakemake und singularity installiert haben, können Sie stattdessen:
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
Wenn Sie Docker installiert haben, können Sie alternativ ausführen:
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
Dadurch wird die Konfigurationsdatei und die Daten im Container verwendet. Die Ergebnisse werden in einem in Ihrem aktuellen Arbeitsverzeichnis out Ordner angezeigt. Unter Linux müssen Sie diesen Befehl als Root ausführen, und unter Windows müssen Sie die Bind -Mount -Syntax ( -v -Argumentation) einstellen.
Die Eingabedaten (Lese- und Host- und virale Referenzen) werden als Konfigurationsdatei als eigene Daten angegeben. Sie müssen die Beispielkonfigurationsdatei ( test/config/test.yml ) ändern, um auf Ihre eigenen Daten zu verweisen. Weitere Informationen zum Format der Konfigurationsdatei finden Sie unter configfile.md .
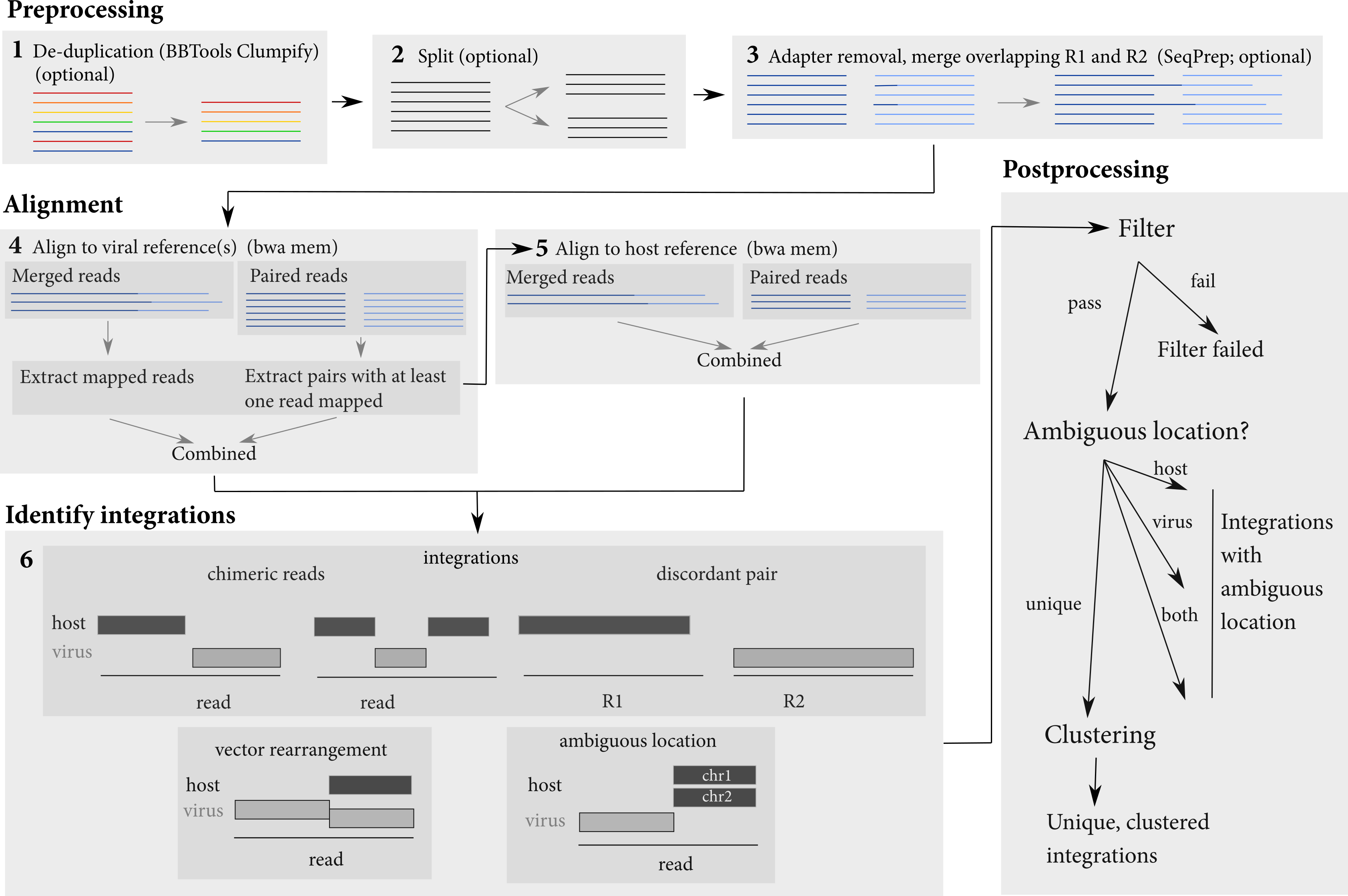
Die Pipeline führt mehrere Schritte aus, um Integrationsstellen zu identifizieren. Es dauert als Eingabedatensätze, die entweder aus Fastq -Dateien oder BAM -Dateien bestehen. Es wird einige Vorverarbeitung der Lesevorgänge durchgeführt (überlappende Lesevorgänge verschmelzen, optional) und sie dann sowohl auf einen Host als auch an eine virale Sequenz ausgerichtet. Die Lesevorgänge sind zuerst auf die Virussequenz ausgerichtet und dann werden ausgerichtete Lesevorgänge extrahiert und mit dem Wirt ausgerichtet. Diese Ausrichtungen werden verwendet, um virale Integrationen zu identifizieren.
Isling erfordert snakemake und entweder singularity (empfohlen) oder conda um Abhängigkeiten zu liefern. Zusätzlich, python Version 3.5 oder höher und pandas sind erforderlich (diese sollten automatisch installiert werden, wenn snakemake mit conda installiert wird.
Alternativley, verwenden Sie die Docker -Version, die Isling und alle Abhängigkeiten enthält.
Die erforderlichen Eingänge sind die Konfigurationsdatei, die die Referenzen des Hosts und der Virus-/Vektor -Referenzen anerkennt, und es sind Lesevorgänge erforderlich. Geben Sie alle Eingaben in einer Konfigurationsdatei an. Isling arbeitet derzeit nur für Paired-End-Lesevorgänge.
Eine Beschreibung des Formats dieser Konfigurationsdatei finden Sie in der Datei configfile.md .
Isling-Ausgänge Integrationsseiten in einem tabendarmen Format in dem in der Konfigurationsdatei angegebenen Ausgabeverzeichnis.
Innerhalb des Ausgabeordners wird für jeden Datensatz in der Konfigurationsdatei ein Ordner erstellt, und für jeden Datensatz finden Sie Integrationen im ints -Verzeichnis. Für jedes Beispiel gibt es einen Satz von Ausgabedateien.
Für jedes Beispiel gibt es eine Reihe von Ausgabedateien, die für bestimmte Anwendungsfälle von Interesse sein können.
<sample>.<host>.<virus>.integrations.txt : Informationen zu allen erkannten Übergängen, unabhängig davon, ob sie die Filter bestanden haben oder nicht<sample>.<host>.<virus>.integrations.post.txt : Informationen zu erkannten Übergängen, die alle Filter bestanden haben<sample>.<host>.<virus>.integrations.post.unique.txt : Informationen zu erkannten Übergängen, die alle Filter übergeben haben und einen eindeutigen Standort sowohl im Host- als auch im Vektor/in Vector/Virus haben<sample>.<host>.<virus>.integrations.post.unique.merged.txt : Das Ergebnis von überlappenden Übergängen oder Kreuzungen mit denselben Koordinaten sowohl im Host als auch im Virus/Vektor. Das Zusammenführen erfolgt nur mit den Integrationsübergängen, die sowohl im Host- als auch im Virus/im Vektor einen eindeutigen Ort haben.<sample>.<host>.<virus>.integrations.post.host_ambig.txt : Informationen zu erkannten Übergängen, die alle Filter übergeben haben und einen eindeutigen Ort Vektor/Virus, aber mehrdeutiger Ort im Host haben<sample>.<host>.<virus>.integrations.post.virus_ambig.txt : Informationen zu erkannten Kreuzungen, die alle Filter übergeben haben und einen eindeutigen Standort -Host, aber mehrdeutiger Ort im Vektor/Virus haben<sample>.<host>.<virus>.integrations.post.both_ambig.txt : Informationen zu erkannten Übergängen, die alle Filter bestanden haben und einen mehrdeutigen Ort in Host- und Vektor/Virus haben Die Ausgabedateien geben den Speicherort der identifizierten Integrationen und deren Eigenschaften an. Koordinaten für Integrationsübergänge werden in Bezug auf ihre mehrdeutigen Grundlagen angegeben. Das heißt, es gibt oft eine Lücke oder Überlappung zwischen dem Wirt und den viralen Teilen einer Read: 
Da der Ort der Integration in diesem Fall nicht eindeutig bestimmt werden kann, gibt Isling die Koordinaten dieser Grundlagen im Host- und Vektor-/Virus -Genom als Ort der Integration aus.
Mit Ausnahme der fusionierten Cluster -Ausgabedatei enthalten alle Dateien diese Spalten:
gap oder overlap für eine schmutzige Lektüre war (Host- und Virussteile, die auf derselben Lektüre identifiziert wurden), ist eine Kreuzung clean , wenn sie keine mehrdeutigen Basen aufweist. Alternativ ist die OverlapType discordant , wenn die Juktion in einem nicht übereinstimmenden Paar identifiziert wurdehv ) oder Virus/Host ( vh )+ oder -bwa ) der Hostausrichtung bearbeitenbwa ) der viralen Ausrichtung bearbeitenOverlapType des Host- gap viralen Bearbeitungsdistnaschimeric Lektüre beobachtet wird, ein discordant Paar oder ist es eine short Integration, in der beide Verbindungen in derselben Lektüre beobachtet wurdenbwa )bwa )'xxx' getrennt Um die Figuren im Isling -Manuskript zu reproduzieren, siehe Readme im benchmarking -Verzeichnis.