Última actualización: 30/09/2024
MSBooster es una herramienta para incorporar predicciones de bibliotecas espectrales en la recuperación de coincidencias de espectro péptido (PSM) en datos proteómicos de espectrometría de masas de cromatografía líquida en tándem ascendente. Se divide aproximadamente en 4 pasos:
Extracción de péptidos de PSM en resultados de búsqueda y formato para archivos de entrada de predictores de aprendizaje automático/aprendizaje profundo (ML/DL)
Llamar a los modelos de predicción y guardar el resultado.
Cálculo de características
Adición de nuevas funciones al archivo de resultados de búsqueda.
MSBooster es compatible con muchos tipos de búsquedas en bases de datos, incluidas inmunopeptidómica HLA, DDA y DIA, y proteómica unicelular. Está incorporado a FragPipe y se incluye en muchos de sus flujos de trabajo. MSBooster se desarrolló teniendo en cuenta otras herramientas FragPipe, como FragPipe-PDV.
MSBooster está equipado para manejar múltiples formatos y modelos de archivos de entrada:
| Salida del espectrómetro de masas |
|---|
| .mzML |
| .mgf |
| Archivo PSM |
|---|
| .alfiler |
| .pepXML (en progreso) |
| Modelo de predicción |
|---|
| DIA-NN |
| Modelos Koina |
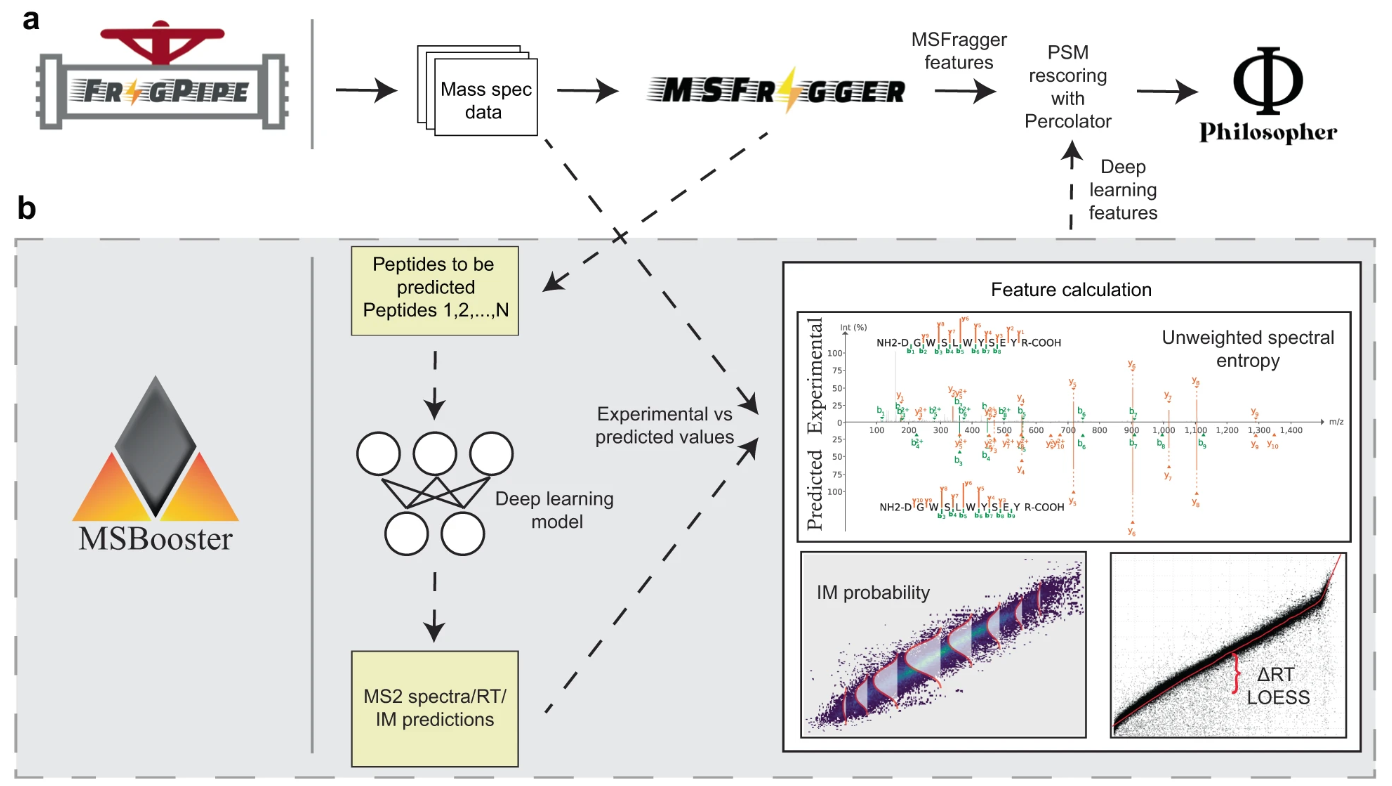
MSBooster se puede ejecutar en sistemas Windows y Linux. Si utiliza FragPipe, no se necesitan otros pasos de instalación además de instalar FragPipe. MSBooster se encuentra en la pestaña "Validación". Elija habilitar las funciones de tiempo de retención con "Predecir RT" y las funciones espectrales de MS/MS con "Predecir espectros". Consulte la documentación de FragPipe para saber cómo ejecutar un análisis.
Si utiliza MSBooster independiente para ejecutar en la línea de comandos, descargue el archivo jar más reciente de Versiones. MSBooster también requiere DIA-NN para predicción de MS/MS y RT. Instale DIA-NN y tome nota de la ruta al ejecutable de DIA-NN (por ejemplo, DiaNN.exe para Windows, diann-1.8.1.8 para Linux).
Puede ejecutar MSBooster usando un comando similar al siguiente:
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
Los parámetros mínimos que deben pasarse son:
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
Si bien puede pasar estos parámetros individualmente, es más fácil colocar uno en cada línea del archivo paramsList. Consulte msbooster_params.txt para obtener una plantilla.
Los parámetros siguientes son para uso general. Los parámetros específicos de Koina se encuentran en la documentación de Koina.
paramsList (String) : ubicación del archivo de texto que contiene los parámetros para esta ejecución
fragger (String) : ruta del archivo fragger.params de la ejecución de MSFragger. MSBooster leerá múltiples parámetros y ajustará los parámetros internos en función de ellos, como la tolerancia al error de masa de fragmentos y las compensaciones de masa.
outputDirectory (String) : dónde generar los nuevos archivos
editedPin (String) : MSBooster nombrará el nuevo archivo según los proporcionados. Por ejemplo, A.pin tendrá una contraparte llamada A_edited.pin. Para cambiar el valor predeterminado de "editado", proporcione una nueva cadena aquí
renamePin (int) : si se debe generar un nuevo archivo pin o reescribir el anterior. El valor predeterminado aquí es 1, que no se sobrescribirá. Establecer esto en 0 sobrescribirá el archivo PIN anterior.
deletePreds (boolean) : si se deben eliminar los archivos que almacenan las predicciones del modelo después de finalizar una ejecución exitosa. De forma predeterminada, se establece en falso. Establezca en verdadero si desea eliminarlos
loadingPercent (int) : con qué frecuencia informar el progreso de las tareas utilizando un informe de progreso. De forma predeterminada, está configurado en 10, lo que significa que se imprimirá una actualización cada 10%.
numThreads (int) : número de subprocesos a utilizar. De forma predeterminada, se establece en 0, que utiliza todos los subprocesos disponibles menos 1.
splitPredInputFile (int) : solo se usa cuando las predicciones DIA-NN fallan debido a un error de falta de memoria (137). De forma predeterminada, establezca en 1, pero puede aumentarlo para especificar en cuántos archivos más pequeños se debe dividir el archivo de entrada DIA-NN. Luego, cada archivo se predecirá secuencialmente, lo que aliviará la carga de memoria.
plotExtension (String) : en qué formato de archivo deben estar los gráficos. png de forma predeterminada, y también se permite pdf
features (String) : lista de características a calcular. Distingue entre mayúsculas y minúsculas, separados por comunicaciones y sin espacios entre ellos. El valor predeterminado es "predRTrealUnits, unweightedSpectralEntropy, deltaRTLOESS"
spectraPredFile (String) : si está reutilizando predicciones espectrales antiguas (por ejemplo, de DIA-NN o Koina), puede especificar la ubicación del archivo aquí
RTPredFile (String) : igual que spectraPredFile, pero para predicciones RT
IMPredFile (String) : igual que spectraPredFile, pero para predicciones de mensajería instantánea
spectraModel (String) : qué modelo de predicción espectral usar
rtModel (String) : igual que spectraModel, pero para RT
imModel (String) : igual que spectraModel, pero para IM
useSpectra (boolean) : si se deben utilizar funciones basadas en predicción espectral. Establecido en verdadero de forma predeterminada
useRT (boolean) : si se deben utilizar funciones basadas en predicciones de RT. Establecido en verdadero de forma predeterminada
useIM (boolean) : si se deben utilizar funciones basadas en predicciones de mensajería instantánea. Establecido en falso de forma predeterminada
ppmTolerance (float) : tolerancia de ppm de error de fragmento (predeterminado 20 ppm)
matchWithDaltons (boolean) : si se deben hacer coincidir los fragmentos previstos y observados en Daltons (falso predeterminado)
DaTolerance (float) : cuántos daltons alrededor del pico previsto buscar para el pico experimental (predeterminado 0,05)
useTopFragments (boolean) : si se debe filtrar la predicción espectral a los N picos de intensidad más altos (verdadero por defecto)
topFragments (int) : hasta cuántos fragmentos predichos se deben usar para el cálculo de características (predeterminado 20). Sólo se aplica si useTopFragments es verdadero
removeRankPeaks (boolean) : establecido en verdadero de forma predeterminada, lo que filtra los fragmentos de los espectros experimentales una vez que coinciden. Si es falso, los fragmentos experimentales pueden coincidir con varios PSM del mismo escaneo
useBasePeak (boolean) : si se debe aplicar un límite inferior a las predicciones de MS2 para usar solo fragmentos con mayor intensidad (verdadero por defecto)
percentBasePeak (float) : porcentaje en el que el fragmento con una intensidad de algún porcentaje de la intensidad máxima base se incluye en el cálculo de similitud. Solo se aplica si useBasePeak es verdadero (predeterminado 1)
loessEscoreCutoff (float) : valor de corte esperado utilizado para el primer paso en la recopilación de PSM para la calibración RT/IM. El valor predeterminado es 10^-3,5, o aproximadamente 0,000316
rtLoessRegressionSize (int) : número máximo de PSM utilizados para la calibración RT LOESS (predeterminado 5000)
imLoessRegressionSize (int) : igual que rtLoessRegressionSize pero para mensajería instantánea (predeterminado 1000)
minLoessRegressionSize (int) : número mínimo de PSM necesarios para intentar la calibración LOESS RT/IM (predeterminado 100). Si hay menos de este número de PSM disponibles, se utiliza la regresión lineal en su lugar.
minLinearRegressionSize (int) : número mínimo de PSM necesarios para intentar la calibración RT/IM de regresión lineal (predeterminado 10). Si hay menos de este número de PSM disponibles, no se intenta realizar ninguna calibración.
loessBandwidth (String) : lista de anchos de banda para probar para la calibración RT/IM LOESS (predeterminado 0.01,0.05,0.1,0.2). Esto debe estar separado por comas y sin espacios entre ellos.
regressionSplits (int) : número de validaciones cruzadas utilizadas para la calibración RT/IM LOESS (predeterminado 5)
massesForLoessCalibration (String) : masas para cambios de masa que deben ajustarse a sus propias curvas de calibración. La lista está separada por comas y sin espacios entre ellas. Las masas deben escribirse con el mismo número de dígitos que en el archivo PIN.
loessScatterOpacity (float) : opacidad de los diagramas de dispersión en las cifras de calibración LOESS, de 0 a 1 (predeterminado 0,35)
Archivo .pin con nuevas funciones. De forma predeterminada, se generarán nuevos archivos pin que terminen en "_edited.pin". Las funciones predeterminadas utilizadas son "unweighted_spectral_entropy", "delta_RT_loess" y "pred_RT_real_units". Si las funciones de movilidad de iones están habilitadas, también se incluirán "delta_IM_loess" y "ion_mobility".
spectraRT.tsv y spectraRT_full.tsv: archivos de entrada para el modelo de predicción DIA-NN
spectraRT.predicted.bin: un archivo binario con predicciones de DIA-NN que MSBooster utilizará para el cálculo de funciones. Si se utiliza FragPipe-PDV, estos archivos se utilizan para generar gráficos espejo de espectros experimentales y predichos.
MSBooster produce múltiples gráficos que se pueden usar para examinar más a fondo cómo se comparan sus datos con las predicciones del modelo.
Carpeta MSBooster_plots:
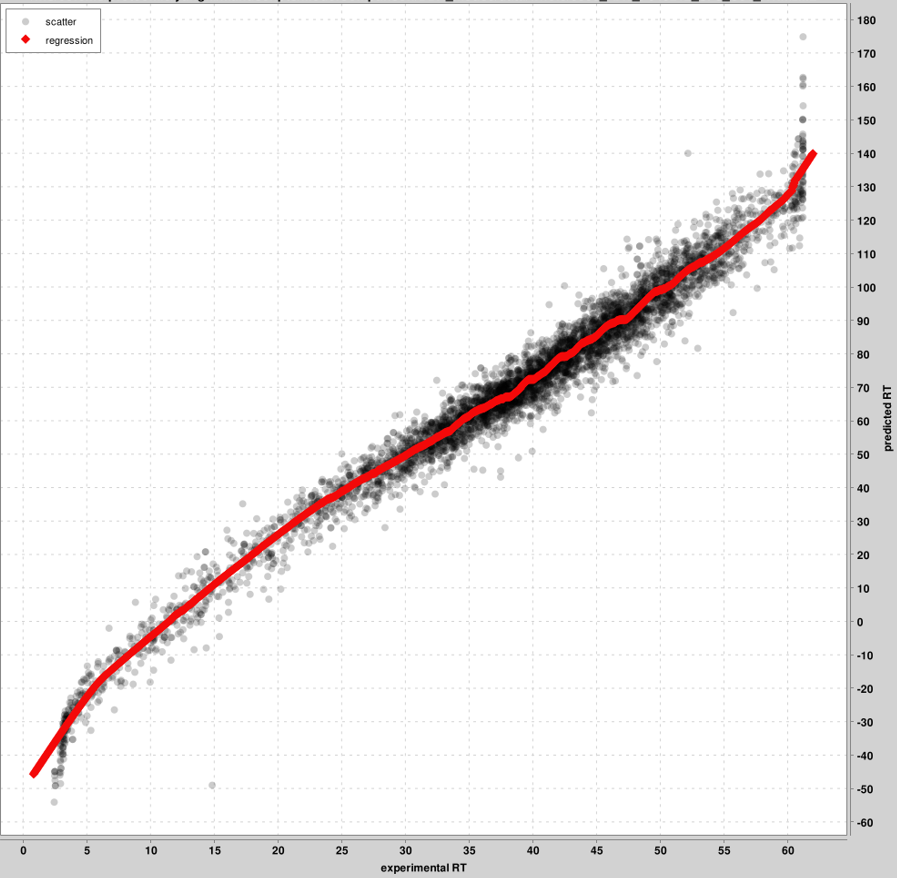
RT_calibration_curves: se utilizarán hasta los 5000 PSM superiores para la calibración entre las escalas RT experimentales y previstas. Estos PSM principales se presentan en el gráfico, no todos los PSM. Se producirá un gráfico por archivo pin
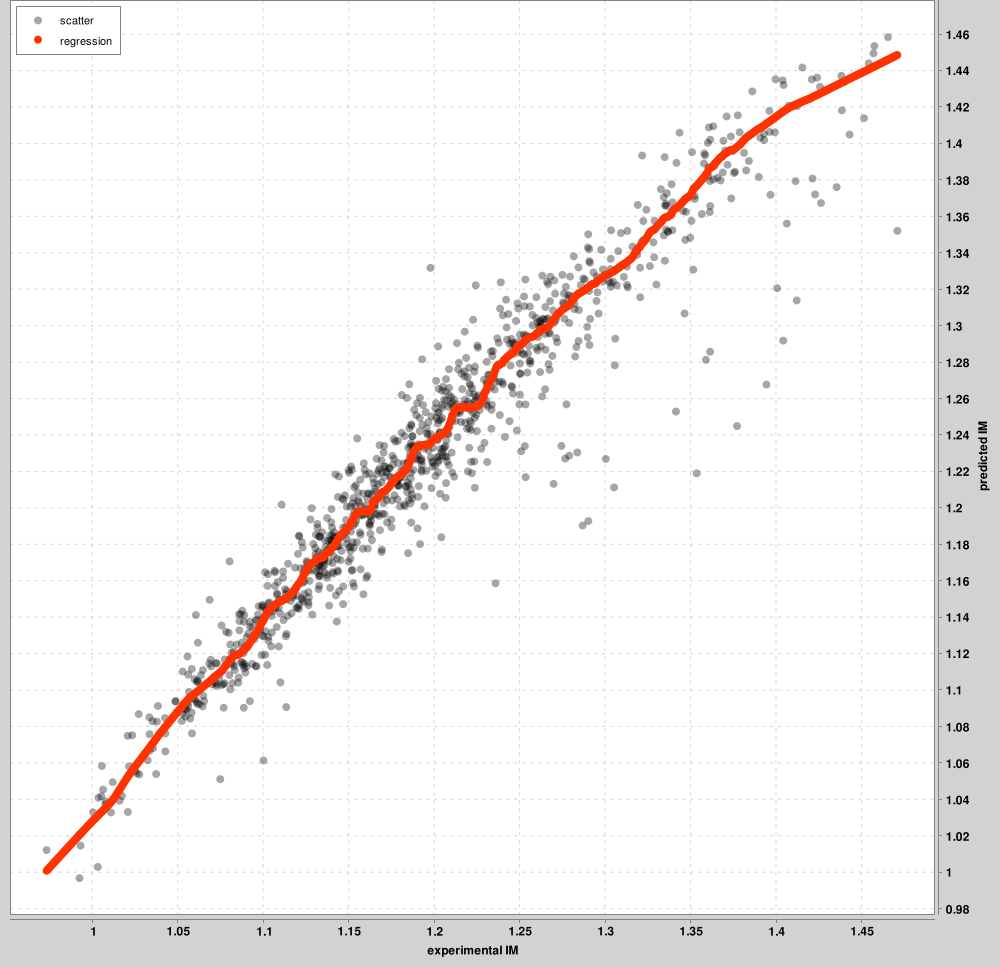
IM_calibration_curves: se utilizarán hasta los 1000 PSM principales para la calibración entre las escalas IM experimentales y predichas. Estos PSM principales se presentan en el gráfico, no todos los PSM. Se aprenderá una curva separada para cada estado de carga. La siguiente figura es un ejemplo de precursores de carga 2.
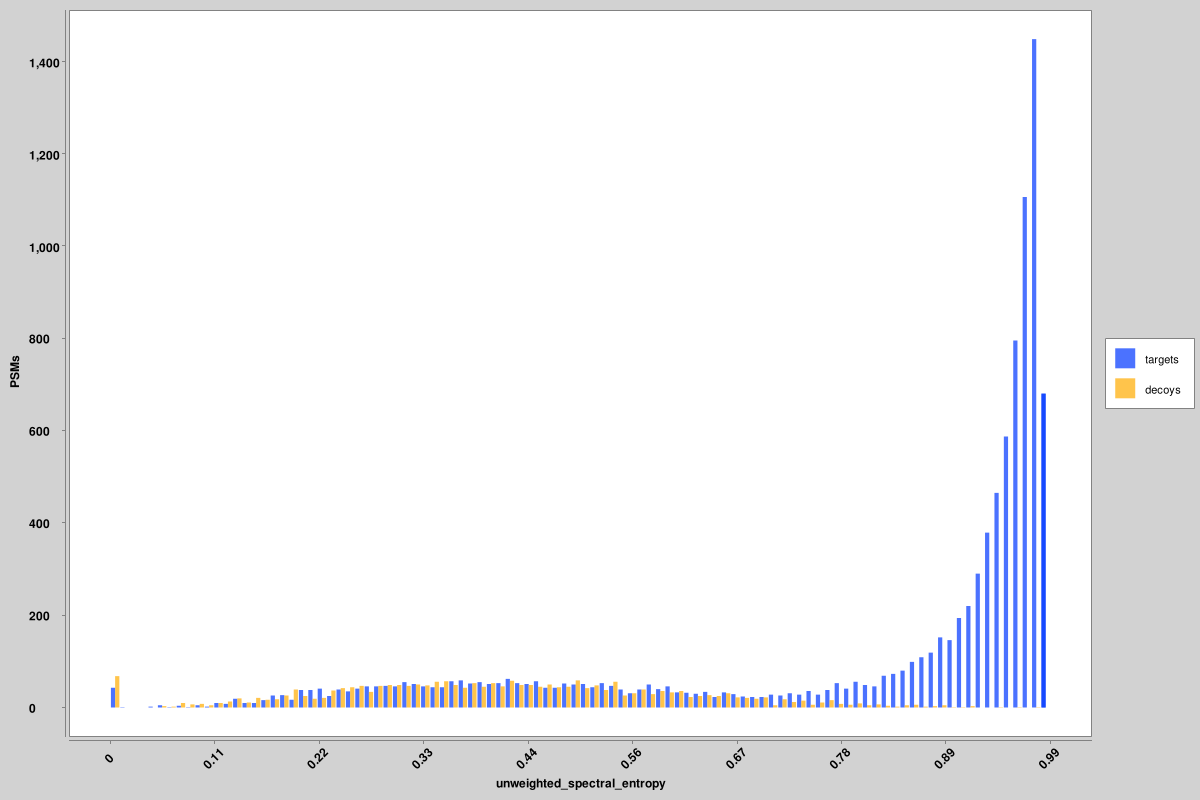
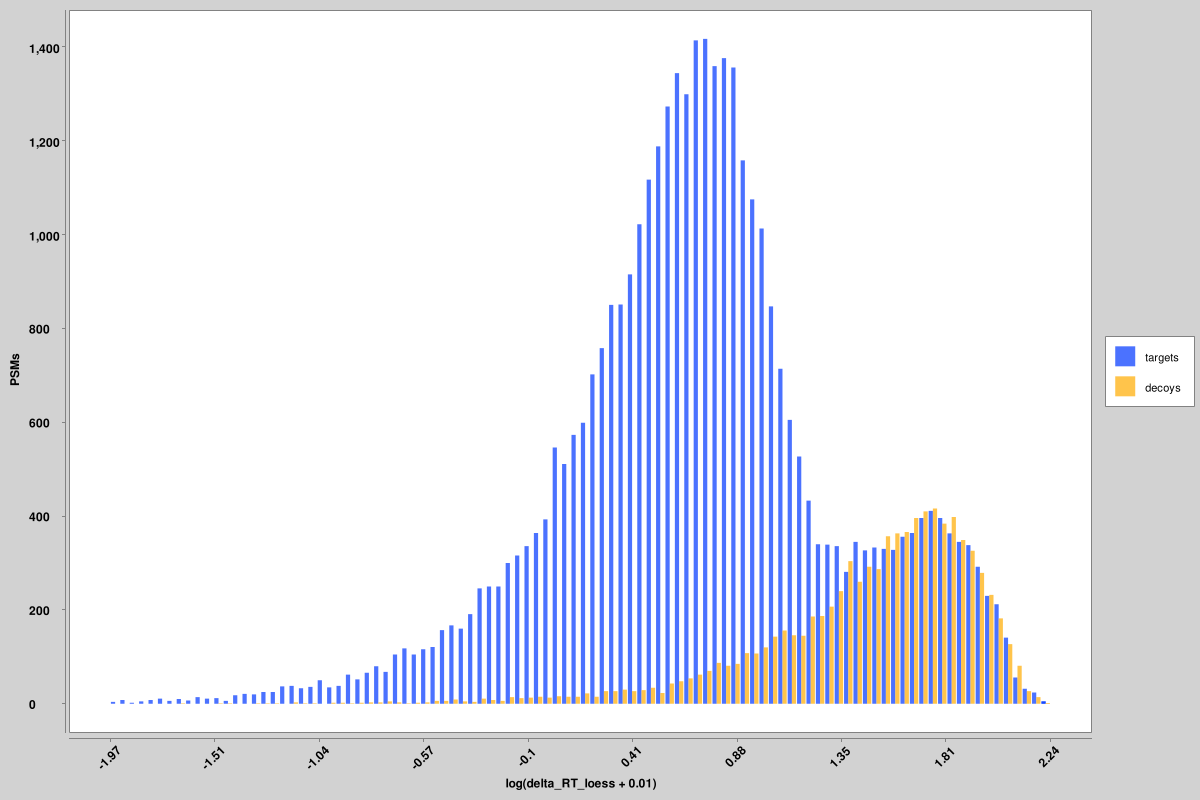
score_histograms: histogramas superpuestos de todos los PSM objetivo y señuelo para cada archivo pin. Algunas características se trazan aquí en una escala logarítmica para una mejor visualización de la distribución bimodal de positivos verdaderos y falsos, pero el valor original es el que se usa en los archivos pin, no en la versión a escala logarítmica. Aquí se muestran histogramas para la entropía espectral no ponderada y las puntuaciones delta RT, pero se producen otros similares para todas las características.
Utilice modelos de predicción de péptidos de Koina para la generación de funciones de MSBooster: https://fragpipe.nesvilab.org/docs/tutorial_koina.html
Lectura de predicciones de cualquier modelo a través de archivos MGF
Documentación sobre todas las funciones permitidas y cómo controlarlas con salida gráfica.
Cite lo siguiente cuando utilice MSBooster: https://www.nature.com/articles/s41467-023-40129-9