Este es el repositorio oficial de "Un modelo para gobernarlos a todos: hacia la segmentación universal de imágenes médicas con indicaciones de texto"
Es un modelo de segmentación universal mejorado por el conocimiento construido sobre una recopilación de datos sin precedentes (72 conjuntos de datos públicos de segmentación médica en 3D), que puede segmentar 497 clases de 3 modalidades diferentes (MR, CT, PET) y 8 regiones del cuerpo humano, solicitadas por texto (anatómico). terminología).
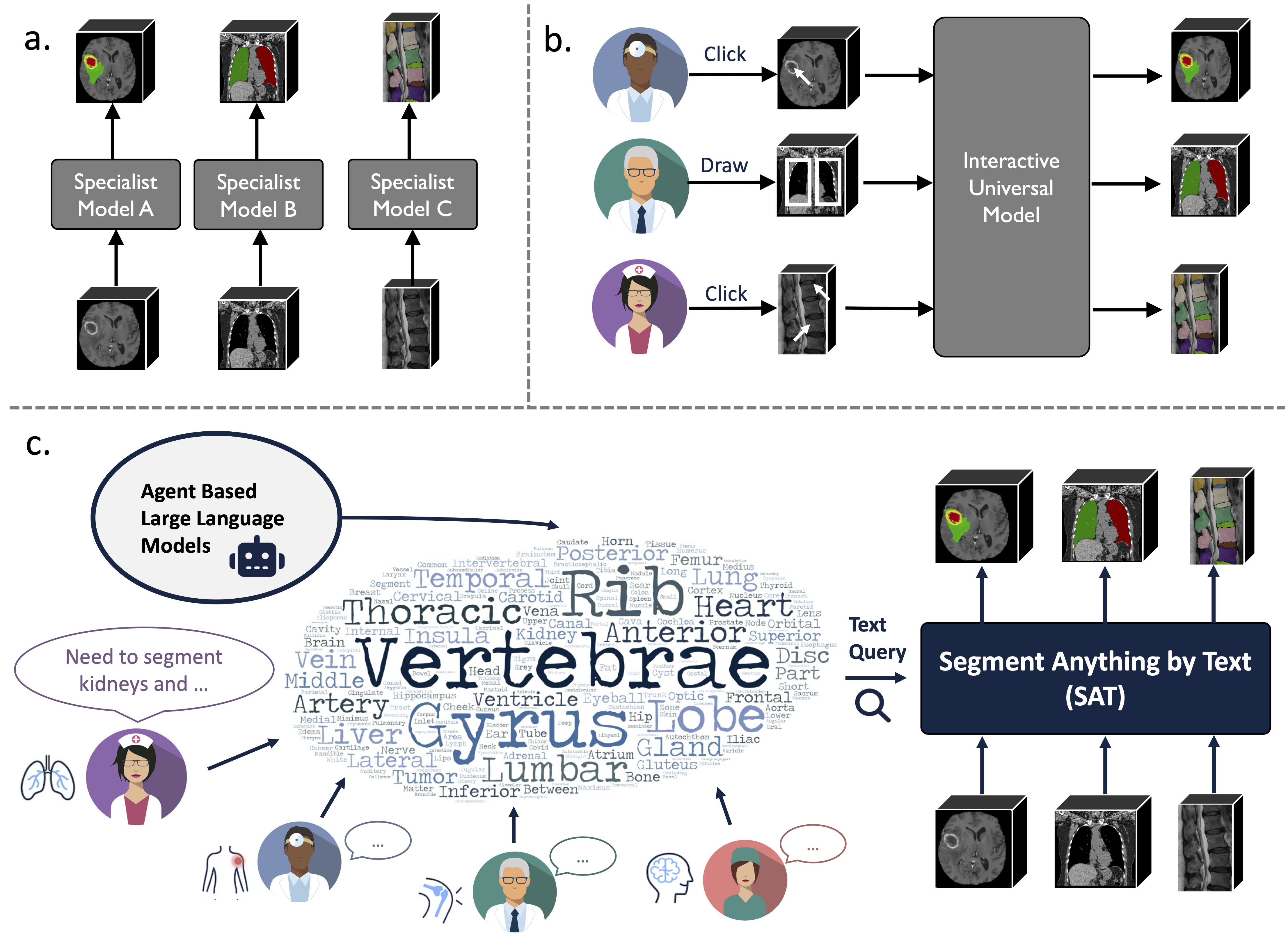
Puede ser poderoso y más eficiente que entrenar e implementar una serie de modelos especializados. Encuentre más en nuestro sitio web o en papel.
2024.08 ? Basándonos en SAT y modelos de lenguaje grande, creamos un conjunto de datos de interpretación de TC de tórax 3D completo, a gran escala y guiado por regiones. Contiene segmentación a nivel de órgano para 196 categorías e informes de granularidad múltiple, donde cada oración se basa en la segmentación correspondiente. Compruébalo en Huggingface.
2024.06 ? Hemos publicado el código para construir SAT-DS , una colección de 72 conjuntos de datos de segmentación públicos, que contiene más de 22.000 imágenes en 3D, 302.000 máscaras de segmentación y 497 clases de 3 modalidades diferentes (MRI, CT, PET) y 8 regiones del cuerpo humano, sobre las cuales construimos SAT. También ofrecemos enlaces de descarga de acceso directo para conjuntos de datos 42/72, que nosotros preprocesamos y empaquetamos para su comodidad, listos para su uso inmediato tras la descarga y extracción. Consulte este repositorio para obtener más detalles.
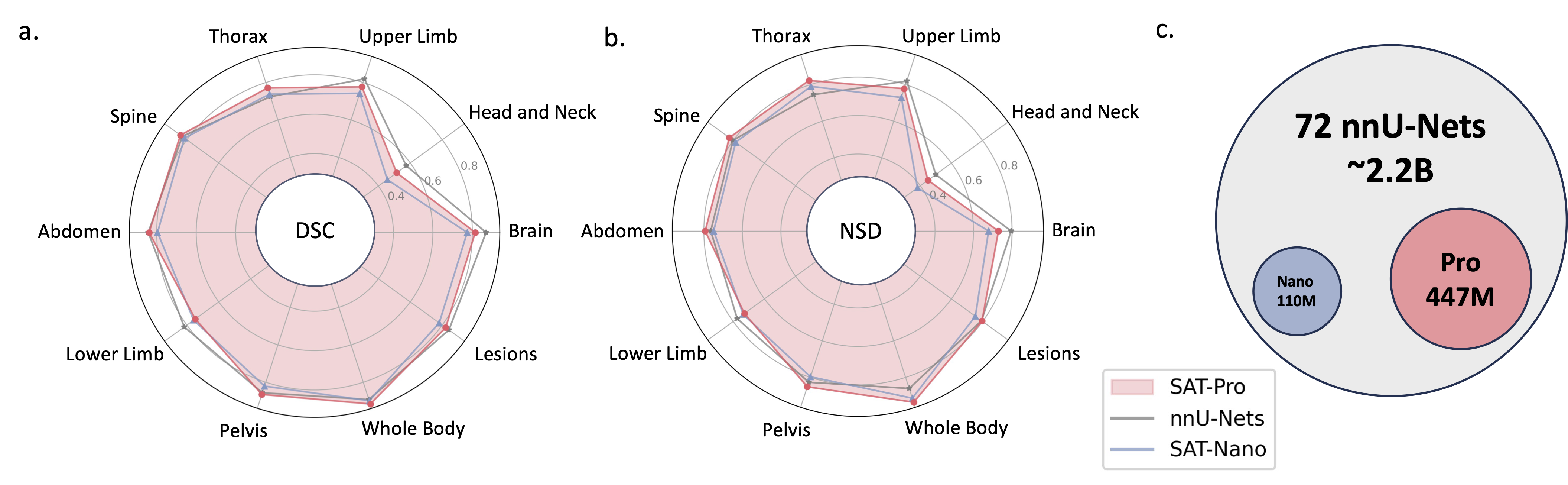
2024.05 ? Entrenamos una nueva versión de SAT con un tamaño de modelo más grande ( SAT-Pro ) y más conjuntos de datos ( 72 ), ¡y ahora admite 497 clases! También renovamos SAT-Nano y lanzamos algunas variantes de SAT-Nano, basadas en diferentes backbones visuales (U-Mamba y SwinUNETR) y codificadores de texto (MedCPT y BERT-Base). Para obtener más detalles sobre esta actualización, consulte nuestro nuevo documento.
La implementación de U-Net se basa en una versión personalizada de arquitecturas de red dinámica, para instalarla:
cd model
pip install -e dynamic-network-architectures-main
Algunos otros requisitos clave:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
También necesitas instalar mamba_ssm si quieres la variante U-Mamba de SAT-Nano
S1. Construya el entorno siguiendo requirements.txt .
S2. Descargue el punto de control de SAT y Text Encoder desde huggingface.
S3. Prepare los datos en un archivo jsonl. Consulte la demostración en data/inference_demo/demo.jsonl .
Se necesitan image (ruta a la imagen), labe (nombre de los objetivos de segmentación), dataset (a qué conjunto de datos pertenece la muestra) y modality (TC, resonancia magnética o mascota) para cada muestra a segmentar. Las modalidades y clases que admite el SAT se pueden encontrar en la Tabla 12 del documento.
orientation_code (orientación) es RAS de forma predeterminada, que se adapta a la mayoría de las imágenes en el plano axial. Para imágenes en el plano sagital (por ejemplo, examen de la columna), configúrelo en ASR . La imagen de entrada debe tener la forma H,W,D Nuestro código de proceso de datos normalizará la imagen de entrada en términos de orientación, intensidad, espaciado, etc. Se pueden encontrar dos imágenes procesadas con éxito en demoprocessed_data . Asegúrese de que la normalización se realice correctamente para garantizar el rendimiento de SAT.
S4. ¿Iniciar la inferencia con SAT-Pro?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d es el tamaño del lote de los parches de imágenes de entrada y debe ajustarse según la memoria de la gpu (consulte la tabla a continuación); --max_queries se recomienda establecer un tamaño mayor que las clases en el conjunto de datos de inferencia, a menos que la memoria de su gpu sea muy limitada;
| Modelo | tamaño de lote_3d | Memoria GPU |
|---|---|---|
| SAT-Pro | 1 | ~ 34GB |
| SAT-Pro | 2 | ~ 62GB |
| SAT-Nano | 1 | ~ 24GB |
| SAT-Nano | 2 | ~ 36GB |
S5. Verifique --rcd_dir para ver las salidas. Los resultados están organizados por conjuntos de datos. Para cada caso, se encontrará la imagen de entrada, el resultado de la segmentación agregado y una carpeta que contiene las segmentaciones de cada clase. Todos los resultados se almacenan como archivos nifiti. Puedes visualizarlos usando el ITK-SNAP.
Si desea utilizar SAT-Nano entrenado en 72 conjuntos de datos, simplemente modifique --vision_backbone a 'UNET' y cambie --checkpoint y --text_encoder_checkpoint en consecuencia.
Para otras variantes de SAT-Nano (entrenadas en 49 conjuntos de datos):
UNET-Nuestro: establezca --vision_backbone 'UNET' y --text_encoder 'ours' ;
UNET-CPT: establezca --vision_backbone 'UNET' y --text_encoder 'medcpt' ;
UNET-BB: establezca --vision_backbone 'UNET' y --text_encoder 'basebert' ;
UMamba-CPT: establezca --vision_backbone 'UMamba' y --text_encoder 'medcpt' ;
SwinUNETR-CPT: establezca --vision_backbone 'SwinUNETR' y --text_encoder 'medcpt' ;
Alguna preparación antes de empezar el entrenamiento:
sh/ para iniciar el proceso de capacitación. Tomemos como ejemplo SAT-Pro: sbatch sh/train_sat_pro.sh
Esto también requiere generar datos de prueba después de este repositorio. Puede consultar el script de slurm sh/evaluate_sat_pro.sh para iniciar el proceso de evaluación:
sbatch sh/evaluate_sat_pro.sh
Si utiliza este código para su investigación o proyecto, cite:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}