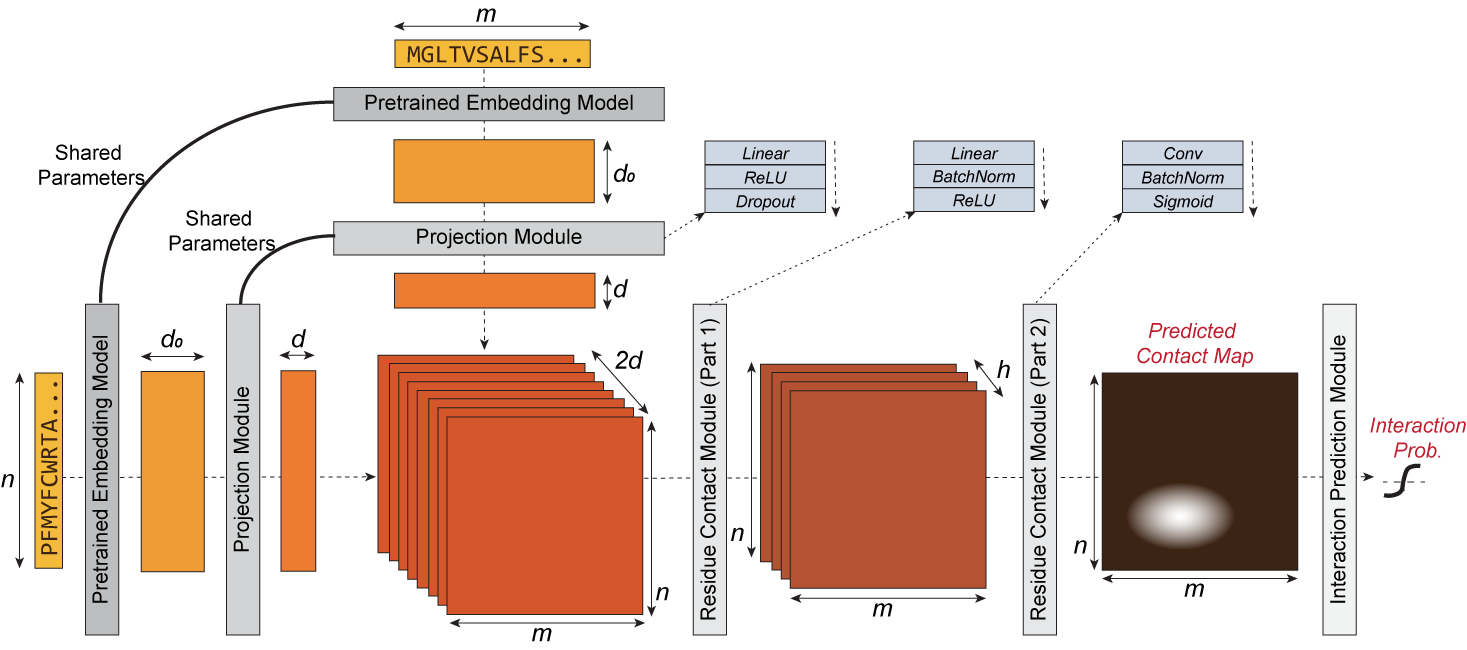
D-SCRIPT es un método de aprendizaje profundo para predecir una interacción física entre dos proteínas dadas solo sus secuencias. Se generaliza bien a nuevas especies y es resistente a las limitaciones en el tamaño de los datos de entrenamiento. Su diseño refleja la intuición de que para que dos proteínas interactúen físicamente, un subconjunto de aminoácidos de cada proteína debe estar en contacto con el otro. Las etapas intermedias de D-SCRIPT implementan directamente esta intuición, siendo la penúltima etapa en D-SCRIPT una estimación aproximada del mapa de contacto entre proteínas del dímero de proteína. Este diseño motivado estructuralmente mejora la interpretabilidad de los resultados y, dado que la estructura se conserva más evolutivamente que la secuencia, mejora la generalización entre especies.
D-SCRIPT se describe en el artículo “Predicción basada en secuencias de interacciones proteína-proteína: un modelo de aprendizaje profundo interpretable y consciente de la estructura” de Sam Sledzieski, Rohit Singh, Lenore Cowen y Bonnie Berger.
Página principal
Documentación