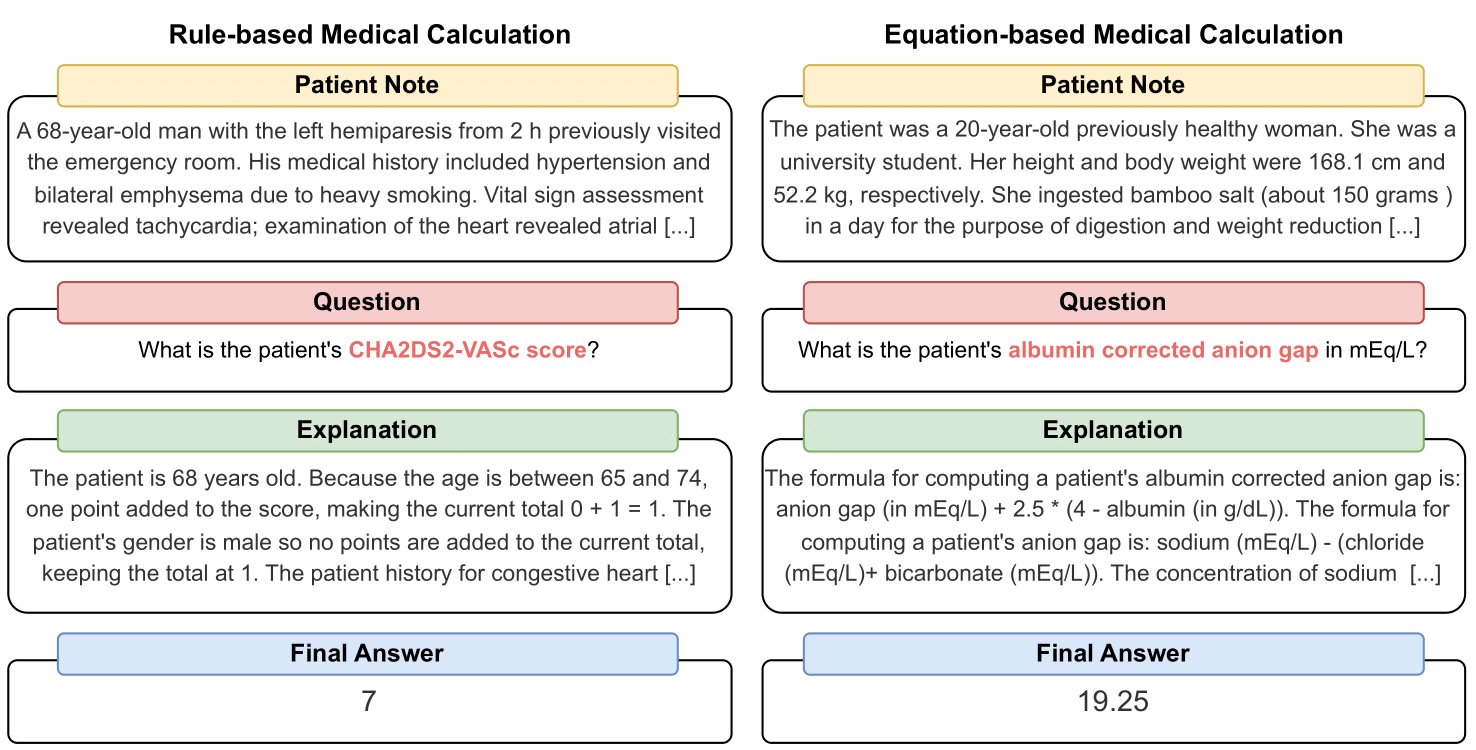
MedCalc-Bench es el primer conjunto de datos de cálculos médicos que se utiliza para comparar la capacidad de los LLM para servir como calculadoras clínicas. Cada instancia del conjunto de datos consta de una nota del paciente, una pregunta que solicita calcular un valor clínico específico, un valor de respuesta final y una solución paso a paso que explica cómo se obtuvo la respuesta final. Nuestro conjunto de datos cubre 55 tareas de cálculo diferentes que son cálculos basados en reglas o cálculos basados en ecuaciones. Este conjunto de datos contiene un conjunto de datos de entrenamiento de 10 053 instancias y un conjunto de datos de prueba de 1047 instancias.
En general, esperamos que nuestro conjunto de datos y nuestro punto de referencia sirvan como un llamado para mejorar las habilidades de razonamiento computacional de los LLM en entornos médicos.
Nuestra preimpresión está disponible en: https://arxiv.org/abs/2406.12036.
Para descargar el CSV para el conjunto de datos de evaluación de MedCalc-Bench, descargue el archivo test_data.csv dentro de la carpeta del dataset de este repositorio. También puede descargar el conjunto de prueba dividido desde HuggingFace en https://huggingface.co/datasets/ncbi/MedCalc-Bench.
Además de las 1047 instancias de evaluación, también proporcionamos un conjunto de datos de capacitación de 10 053 instancias que se pueden utilizar para ajustar los LLM de código abierto (consulte la Sección C del Apéndice). Los datos de entrenamiento se pueden encontrar en el archivo dataset/train_data.csv.zip y se pueden descomprimir para obtener train_data.csv . Este conjunto de datos de entrenamiento también se puede encontrar en la división del tren del enlace HuggingFace.
Cada instancia del conjunto de datos contiene la siguiente información:
Para instalar todos los paquetes necesarios para este proyecto, ejecute el siguiente comando: conda env create -f environment.yml . Este comando creará el entorno conda medcalc-bench . Para ejecutar modelos OpenAI, deberá proporcionar su clave OpenAI en este entorno conda. Puede hacer esto ejecutando el siguiente comando en el entorno medcalc-bench : export OPENAI_API_KEY = YOUR_API_KEY , donde YOUR_API_KEY es su clave API de OpenAI. También deberá proporcionar su token HuggingFace en este entorno ejecutando el siguiente comando: export HUGGINGFACE_TOKEN=your_hugging_face_token , donde your_hugging_face_token es su token huggingface.
Para reproducir la Tabla 2 del artículo, primero cd en la carpeta evaluation . Luego, ejecute el siguiente comando: python run.py --model <model_name> and --prompt <prompt_style> .
Las opciones para --model están a continuación:
Las opciones para --prompt están a continuación:
A partir de esto, obtendrá un archivo jsonl que generará el estado de cada pregunta: al ejecutar run.py , los resultados se guardarán en un archivo llamado <model>_<prompt>.jsonl . Este archivo se puede encontrar en la carpeta de outputs .
Cada instancia en jsonl tendrá los siguientes metadatos asociados:
{
"Row Number": Row index of the item,
"Calculator Name": Name of calculation task,
"Calculator ID": ID of the calculator,
"Category": type of calculation (risk, severity, diagnosis for rule-based calculators and lab, risk, physical, date, dosage for equation-based calculators),
"Note ID": ID of the note taken directly from MedCalc-Bench,
"Patient Note": Paragraph which is the patient note taken directly from MedCalc-Bench,
"Question": Question asking for a specific medical value to be computed,
"LLM Answer": Final Answer Value from LLM,
"LLM Explanation": Step-by-Step explanation by LLM,
"Ground Truth Answer": Ground truth answer value,
"Ground Truth Explanation": Step-by-step ground truth explanation,
"Result": "Correct" or "Incorrect"
}
Además, proporcionamos el porcentaje de precisión media y desviación estándar para cada subcategoría en un json titulado results_<model>_<prompt_style>.json . La precisión acumulada y la desviación estándar entre las 1047 instancias se pueden encontrar en la clave "general" del JSON. Este archivo se puede encontrar en la carpeta de results .
Además de los resultados de la Tabla 2 del artículo principal, también solicitamos a los LLM que escribieran código para realizar aritmética en lugar de que el LLM lo hiciera él mismo. Los resultados de esto se pueden encontrar en el Apéndice D. Debido a la computación limitada, solo ejecutamos los resultados para GPT-3.5 y GPT-4. Para examinar las indicaciones y ejecutar con esta configuración, examine el archivo generate_code_prompt.py en la carpeta evaluation .
Para ejecutar este código, simplemente cd a la carpeta de evaluations y ejecute lo siguiente: python generate_code_prompt.py --gpt <gpt_model> . Las opciones para <gpt_model> son 4 para ejecutar GPT-4 o 35 para ejecutar GPT-3.5-turbo-16k. Luego, los resultados se guardarán en un archivo jsonl llamado: code_exec_{model_name}.jsonl en la carpeta de outputs . Tenga en cuenta que en este caso, model_name será gpt_4 si elige ejecutar usando GPT-4. De lo contrario, model_name será gpt_35_16k si seleccionó ejecutar con GPT-3.5-turbo.
Los metadatos de cada instancia en el archivo jsonl para los resultados del intérprete de código son la misma información de instancia proporcionada en la sección anterior. La única diferencia es que almacenamos el historial de chat de LLM entre el usuario y el asistente y tenemos una clave de "Historial de chat de LLM" en lugar de la clave de "Explicación de LLM". Además, la subcategoría y la precisión general se almacenan en un archivo JSON denominado results_<model_name>_code_augmented.json . Este JSON se encuentra en la carpeta de results .
Esta investigación fue apoyada por el Programa de Investigación Intramuros de los NIH, Biblioteca Nacional de Medicina. Además, las contribuciones realizadas por Soren Dunn se realizaron utilizando el recurso de datos y computación avanzada Delta que cuenta con el apoyo de la Fundación Nacional de Ciencias (premio OAC tel:2005572) y el estado de Illinois. Delta es un esfuerzo conjunto de la Universidad de Illinois Urbana-Champaign (UIUC) y su Centro Nacional de Aplicaciones de Supercomputación (NCSA).
Para seleccionar las notas de los pacientes en MedCalc-Bench, solo utilizamos notas de pacientes disponibles públicamente de artículos de informes de casos publicados en PubMed Central y viñetas de pacientes anónimas generadas por médicos. Como tal, en este estudio no se revela información de salud personal identificable. Si bien MedCalc-Bench está diseñado para evaluar las capacidades de cálculo médico de los LLM, cabe señalar que el conjunto de datos no está destinado al uso de diagnóstico directo ni a la toma de decisiones médicas sin la revisión y supervisión de un profesional clínico. Las personas no deberían cambiar su comportamiento de salud basándose únicamente en nuestro estudio.
Como se describe en la Sección 1, las calculadoras médicas se utilizan comúnmente en el entorno clínico. Con el interés cada vez mayor en el uso de LLM para aplicaciones de dominios específicos, los profesionales de la salud podrían solicitar directamente a chatbots como ChatGPT que realicen tareas de cálculo médico. Sin embargo, actualmente se desconocen las capacidades de los LLM en estas tareas. Dado que la atención médica es un ámbito de alto riesgo y los cálculos médicos incorrectos pueden tener consecuencias graves, incluidos diagnósticos erróneos, planes de tratamiento inadecuados y daños potenciales a los pacientes, es crucial evaluar exhaustivamente el desempeño de los LLM en cálculos médicos. Sorprendentemente, los resultados de la evaluación en nuestro conjunto de datos MedCalc-Bench muestran que todos los LLM estudiados tienen dificultades en las tareas de cálculo médico. El modelo más capaz, GPT-4, logra solo un 50 % de precisión con aprendizaje de una sola vez e indicaciones de cadena de pensamiento. Como tal, nuestro estudio indica que los LLM actuales aún no están listos para usarse en cálculos médicos. Cabe señalar que, si bien las puntuaciones altas en MedCalc-Bench no garantizan la excelencia en las tareas de cálculo médico, fallar en este conjunto de datos indica que los modelos no deben considerarse para tales fines en absoluto. En otras palabras, creemos que aprobar MedCalc-Bench debería ser una condición necesaria (pero no suficiente) para que un modelo se utilice para cálculos médicos.
Para cualquier cambio en este conjunto de datos (es decir, agregar nuevas notas o calculadoras), actualizaremos las instrucciones README, test_set.csv y train_set.csv. Seguiremos manteniendo las versiones anteriores de estos conjuntos de datos en un archive/ carpeta. También actualizaremos los conjuntos de entrenamiento y prueba para HuggingFace.
Esta herramienta muestra los resultados de investigaciones realizadas en la Rama de Biología Computacional, NCBI/NLM. La información producida en este sitio web no está destinada al uso de diagnóstico directo ni a la toma de decisiones médicas sin la revisión y supervisión de un profesional clínico. Las personas no deben cambiar su comportamiento de salud basándose únicamente en la información producida en este sitio web. Los NIH no verifican de forma independiente la validez o utilidad de la información producida por esta herramienta. Si tiene preguntas sobre la información producida en este sitio web, consulte a un profesional de la salud. Más información sobre la política de exención de responsabilidad del NCBI está disponible.
Dependiendo de la calculadora, nuestro conjunto de datos consta de notas que fueron diseñadas a partir de funciones basadas en plantillas implementadas en Python, escritas a mano por médicos o tomadas de nuestro conjunto de datos, Open-Patients.
Open-Patients es un conjunto de datos agregado de 180.000 notas de pacientes procedentes de tres fuentes diferentes. Tenemos autorización para utilizar el conjunto de datos de las tres fuentes. La primera fuente son las preguntas USMLE de MedQA que se publican bajo la licencia MIT. La segunda fuente de nuestro conjunto de datos son Trec Clinical Decision Support y Trec Clinical Trial, que están disponibles para redistribución porque ambos son conjuntos de datos de propiedad gubernamental publicados al público. Por último, PMC-Patients se publica bajo la licencia CC-BY-SA 4.0, por lo que tenemos permiso para incorporar PMC-Patients dentro de Open-Patients y MedCalc-Bench, pero el conjunto de datos debe publicarse bajo la misma licencia. Por lo tanto, nuestra fuente de notas, Open-Patients, y el conjunto de datos elaborado a partir de ella, MedCalc-Bench, se publican bajo la licencia CC-BY-SA 4.0.
Con base en la justificación de las reglas de licencia, tanto Open-Patients como MedCalc-Bench cumplen con la licencia CC-BY-SA 4.0, pero los autores de este artículo asumirán toda la responsabilidad en caso de violación de derechos.
@misc { khandekar2024medcalcbench ,
title = { MedCalc-Bench: Evaluating Large Language Models for Medical Calculations } ,
author = { Nikhil Khandekar and Qiao Jin and Guangzhi Xiong and Soren Dunn and Serina S Applebaum and Zain Anwar and Maame Sarfo-Gyamfi and Conrad W Safranek and Abid A Anwar and Andrew Zhang and Aidan Gilson and Maxwell B Singer and Amisha Dave and Andrew Taylor and Aidong Zhang and Qingyu Chen and Zhiyong Lu } ,
year = { 2024 } ,
eprint = { 2406.12036 } ,
archivePrefix = { arXiv } ,
primaryClass = { id='cs.CL' full_name='Computation and Language' is_active=True alt_name='cmp-lg' in_archive='cs' is_general=False description='Covers natural language processing. Roughly includes material in ACM Subject Class I.2.7. Note that work on artificial languages (programming languages, logics, formal systems) that does not explicitly address natural-language issues broadly construed (natural-language processing, computational linguistics, speech, text retrieval, etc.) is not appropriate for this area.' }
}