Conjunto de flujos de trabajo moleculares y computacionales para secuenciación espacial total de ARN (STRS)
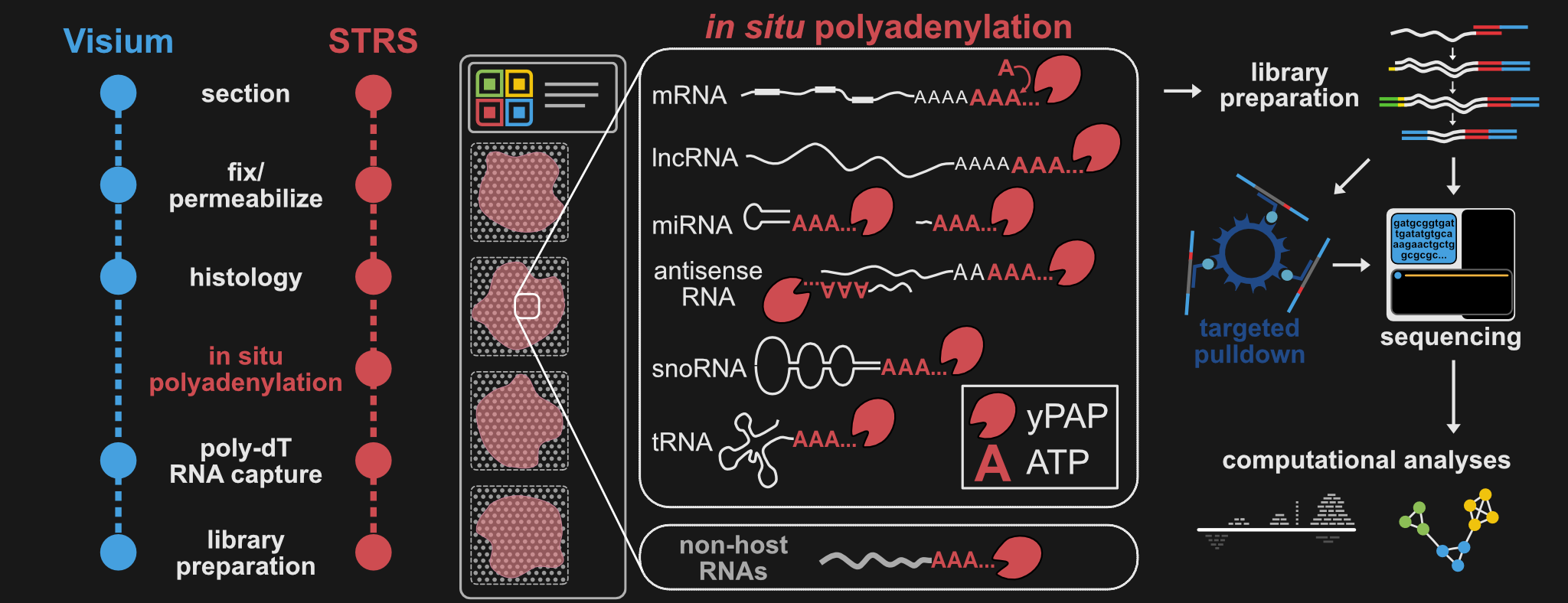
¡Este repositorio tiene todos los protocolos, canalizaciones y scripts que necesitará para ejecutar y analizar STRS en casi cualquier muestra!
txg_snake , un canal de trabajo en progreso para bibliotecas 10x, ¡especialmente conjuntos de datos STRS!McKellar et al, Biotecnología de la naturaleza , 2022
enlace de preimpresión de bioRxiv
manuscripts : archivos .pdf de la preimpresión y el manuscrito posterior (¡con suerte!)
protocols : Documentos en Microsoft Word y .pdf de los protocolos utilizados aquí.
pipelines : flujos de trabajo de Snakemake utilizados en nuestro estudio. Estos incluyen canales de alineación para datos STRS (kallisto, STARsolo y miRge3.0), datos pequeños de RNAseq (STAR y miRge3.0), SmartSeqTotal (kallisto) y VASAdrop (kallisto).
scripts : ¡Todo el resto del código que usamos! Contiene principalmente scripts R y funciones de utilidad utilizadas en nuestros análisis espaciales.
references : información y guiones sobre genomas de referencia y anotaciones utilizadas en este estudio
resources : metadatos variados, listas de genes y otra información que utilizamos para analizar nuestros datos. Consulte los archivos README en cada subdirectorio para obtener más detalles.
Conjuntos de datos STRS: GSE200481
Datos de SkM/corazón pequeños RNAseq: GSE200480
datos de RNAseq total de un solo núcleo (línea celular C2C12): GSE209780
Datos del Atlas de ARN pequeño (Isakova et al, PNAS , 2020): GSE119661
Datos de SmartSeqTotal (Isakova et al, PNAS , 2021): GSE151334
Datos de VASAdrop (Salmen et al, Nature Biotechnology , 2022): GSE176588
Iwijn De Vlaminck ([email protected])
Benjamín D. Cosgrove ([email protected])
David W. McKellar ([email protected])