Este proyecto consta de diferentes métodos para muestrear la población y evaluación de diferentes métodos. Incluimos muchas situaciones que pueden causar sesgos en la estimación del nivel de infección según la muestra, incluidos los que no responden, la tasa de falsos positivos/negativos y la capacidad del perfil de transmisión de los pacientes durante su período de infección. Basado en el modelo EpiABM, este paquete también puede generar el mejor método de muestreo ejecutando simulaciones de transmisión de enfermedades para ver el error de predicción de cada método de muestreo.
EpiOS aún no está disponible en PyPI, pero el módulo se puede instalar localmente. El directorio primero debe descargarse en su máquina local y luego puede instalarse usando el comando:
pip install -e .También le recomendamos instalar el modelo EpiABM para generar los datos de simulación de infección. En primer lugar, puede descargar pyEpiabm en cualquier ubicación de su máquina y luego instalarlo usando el comando:
pip install -e path/to/pyEpiabm Se puede acceder a la documentación a través de la insignia docs anterior.
Aquí hay un diagrama de clases UML para nuestro proyecto: 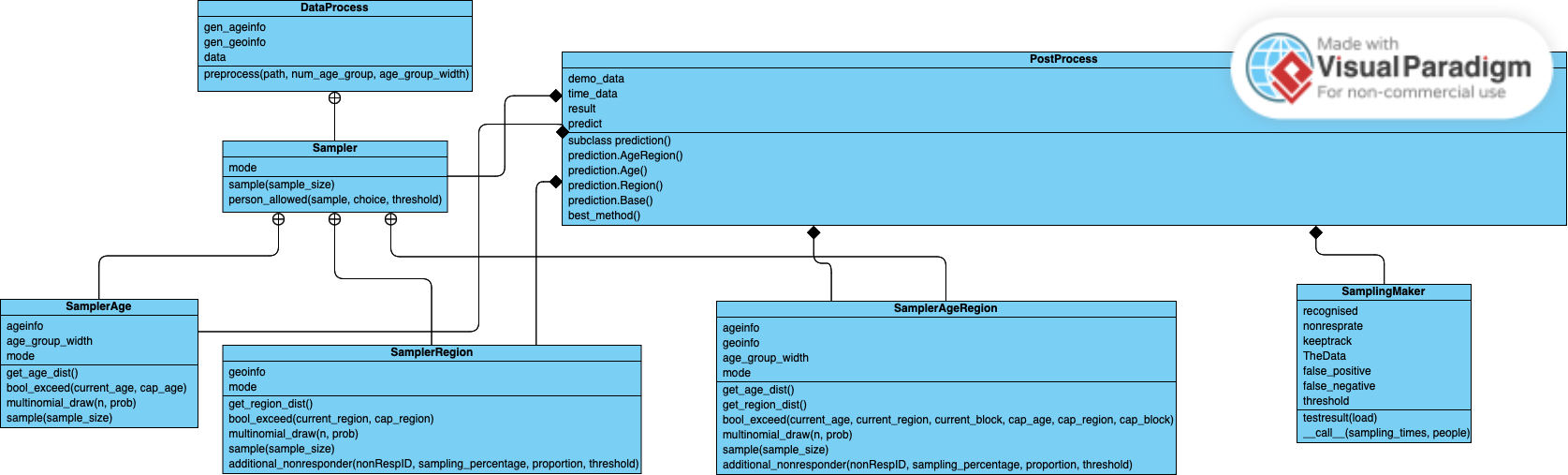
El archivo params.py incluye todos los parámetros necesarios en este modelo. Además, los archivos en la carpeta input son ejemplos de archivos temporales generados durante el preprocesamiento de datos. Será utilizado por las clases de muestra. El parámetro data_store_path en cada clase de muestra es la ruta para almacenar estos archivos.
PostProcess para generar gráficos En primer lugar, debe definir un nuevo objeto PostProcess e ingresar los demodata demográficos y los datos timedata de infección generados a partir de pyEpiabm. En segundo lugar, puede utilizar PostProcess.predict para realizar predicciones basadas en diferentes métodos de muestreo. Puede llamar directamente al método de muestreo que desea utilizar como método; luego especifique los momentos de la muestra y el tamaño de la muestra. Aquí, usaremos AgeRegion como método de muestreo, [0, 1, 2, 3, 4, 5] como puntos de tiempo para muestrear y 3 como tamaño de muestra. Por último, puede especificar si desea considerar a los que no responden y si desea comparar sus resultados con los datos verdaderos especificando el parámetro non_responder y comparison .
Como ejemplo de código, puede ver lo siguiente:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)¡Ahora tendrás tu figura guardada en el camino indicado!
PostProcess para seleccionar el mejor método de muestreo En primer lugar, debe definir un nuevo objeto PostProcess e ingresar los demodata demográficos y los datos timedata de infección generados a partir de pyEpiabm. En segundo lugar, puede utilizar PostProcess.best_method para comparar el rendimiento de diferentes métodos de muestreo. Puede proporcionar los métodos que desee comparar; luego especifique los intervalos de muestreo para la muestra y el tamaño de la muestra. En tercer lugar, puede especificar si desea considerar a los que no responden y si desea comparar sus resultados con los datos reales especificando el parámetro non_responder y comparison . Además, dado que los métodos de muestreo son estocásticos, puede especificar el número de iteraciones ejecutadas para obtener el rendimiento promedio. Además, parallel_computation se puede activar para acelerar. Por último, puede activar hyperparameter_autotune para encontrar automáticamente la mejor combinación de hiperparámetros.
Como ejemplo de código, puede ver lo siguiente:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed