
Este repositorio es la implementación oficial de CELL-E 2: Traducción de proteínas a imágenes y viceversa con un transformador bidireccional de texto a imagen.
Cree un entorno virtual e instale los paquetes necesarios a través de:
pip install -r requirements.txt
A continuación, instale torch = 2.0.0 con la versión CUDA adecuada.
Los modelos están disponibles en Hugging Face.
¡También tenemos dos espacios disponibles donde puedes ejecutar predicciones sobre tus propios datos!
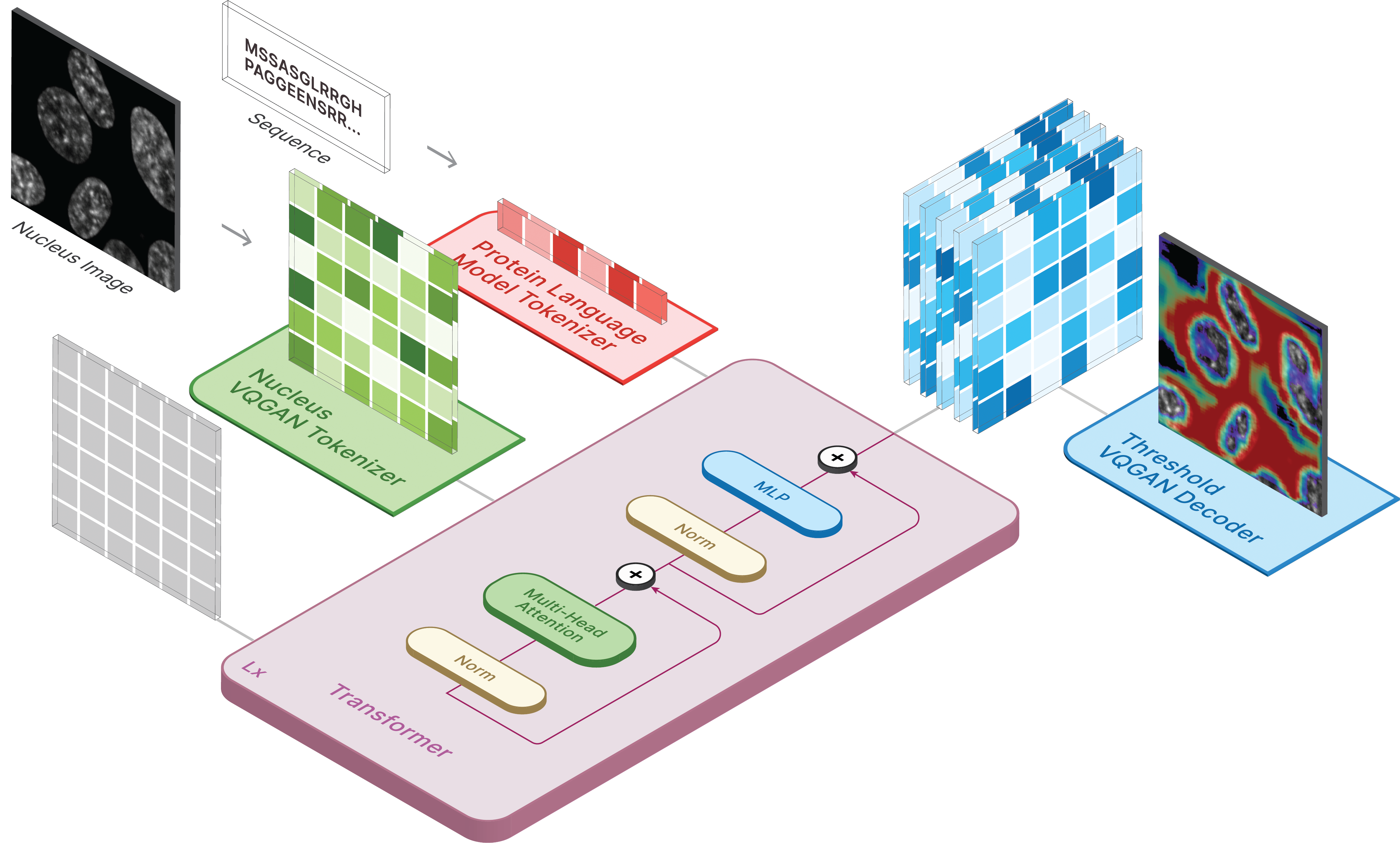
Para generar imágenes, configure el modelo guardado como ckpt_path. Este método puede ser inestable, así que consulte Demo.ipynb para ver otra forma de carga.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
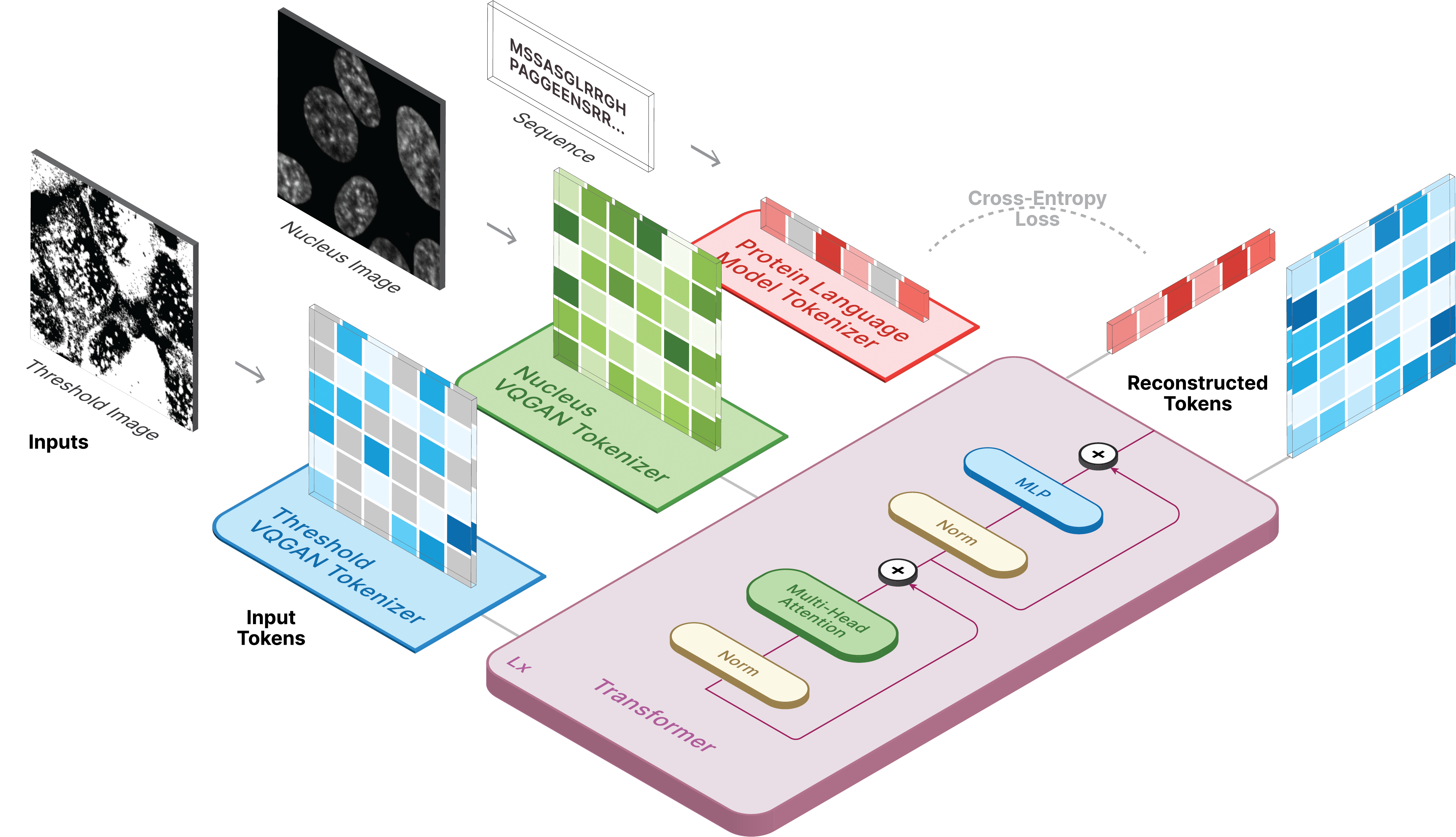
progress = True )El entrenamiento para CELL-E se produce en 3 etapas:
Si utiliza la imagen del umbral de proteínas, establezca threshold: True para el conjunto de datos.
Usamos una versión ligeramente modificada del código de domesticación de transformadores.
Para entrenar, ejecute el siguiente script:
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
Consulte el repositorio original para ver indicadores adicionales, como --gpus .
Proporcionamos secuencias de comandos para descargar imágenes de Human Protein Atlas y OpenCell en la carpeta de secuencias de comandos. Se necesita un data_csv para el cargador de datos. Debe generar un archivo csv que contenga las columnas nucleus_image_path , protein_image_path , metadata_path , split (train o val) y sequence (opcional). Se supone que este archivo existe dentro de la misma carpeta data generales que los archivos de imágenes y metadatos.
Los metadatos son un JSON que deben acompañar a cada secuencia de proteínas. Si una secuencia no aparece en data_csv , debe aparecer en metadata.json con una clave denominada protein_sequence .
Agregar más información aquí puede resultar útil para consultar proteínas individuales. Se pueden recuperar a través de retrieve_metadata , que crea una variable self.metadata dentro del objeto del conjunto de datos.
Para entrenar, ejecute el siguiente script:
python celle_main.py --base configs/celle.yaml -t True
Especifique --gpus en el mismo formato que VQGAN.
CELL-E contiene las siguientes opciones:
ckpt_path : reanuda el entrenamiento anterior de CELL-E 2. Modelo guardado con state_dictvqgan_model_path : modelo de imagen de proteína guardado (con state_dict) para el codificador de imágenes de proteínavqgan_config_path : modelo de imagen de proteína guardada yamlcondition_model_path : modelo de condición (núcleo) guardado (con state_dict) para el codificador de imágenes de proteínascondition_config_path : modelo de condición guardada (núcleo) yamlnum_images : 1 si solo se usa un codificador de imágenes de proteínas, 2 si se incluye un codificador de imágenes de condiciónimage_key : nucleus , target o thresholddim : dimensión de la incrustación del modelo de lenguajenum_text_tokens : número total de tokens en el modelo de lenguaje (33 para ESM-2)text_seq_len : Número total de aminoácidos consideradosdepth : profundidad del modelo de transformador, más profundo suele ser mejor a costa de VRAMheads : número de cabezas utilizadas en atención multicabezaldim_head : tamaño de las cabezas de atenciónattn_dropout : Tasa de abandono de la atención en el entrenamientoff_dropout : Tasa de abandono de feed-forward en el entrenamientoloss_img_weight : Ponderación aplicada a la reconstrucción de imágenes. peso del texto = 1loss_text_weight : ponderación aplicada a la reconstrucción de imágenes condicionadas.stable : Pesos de normas (para cuando se producen gradientes explosivos)learning_rate : tasa de aprendizaje para el optimizador Adammonitor : parámetro utilizado para guardar modelos Cítenos si decide utilizar nuestro código para cualquier parte de su investigación.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}