spfy : plataforma para predecir subtipos a partir de secuencias del genoma completo de E. coli y crea datos gráficos para análisis comparativos de toda la población.
Publicado como: Le,KK, Whiteside,MD, Hopkins,JE, Gannon,VPJ, Laing,CR spfy : una base de datos gráfica integrada para la predicción en tiempo real de fenotipos bacterianos y análisis comparativos posteriores. Base de datos (2018) vol. 2018: artículo ID bay086; doi:10.1093/base de datos/bay086
En vivo: https://lfz.corefacility.ca/superphy/spfy/
git clone --recursive https://github.com/superphy/spfy.gitcd spfy /docker-compose upTipo EC:
PanPredic:
Imagen de Docker para Conda:
Comparando diferentes grupos de población:
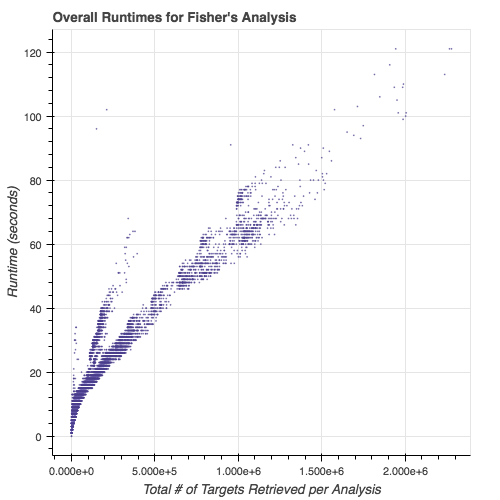
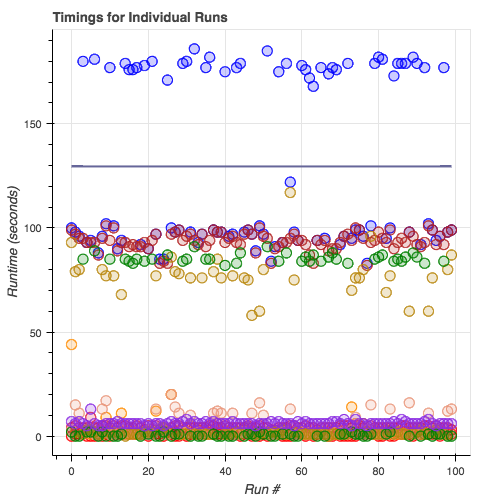
Tiempos de ejecución de módulos de subtipos:
cd app/python -m modules/savvy -i tests/ecoli/GCA_001894495.1_ASM189449v1_genomic.fna donde el argumento después de -i es su archivo genoma (FASTA). 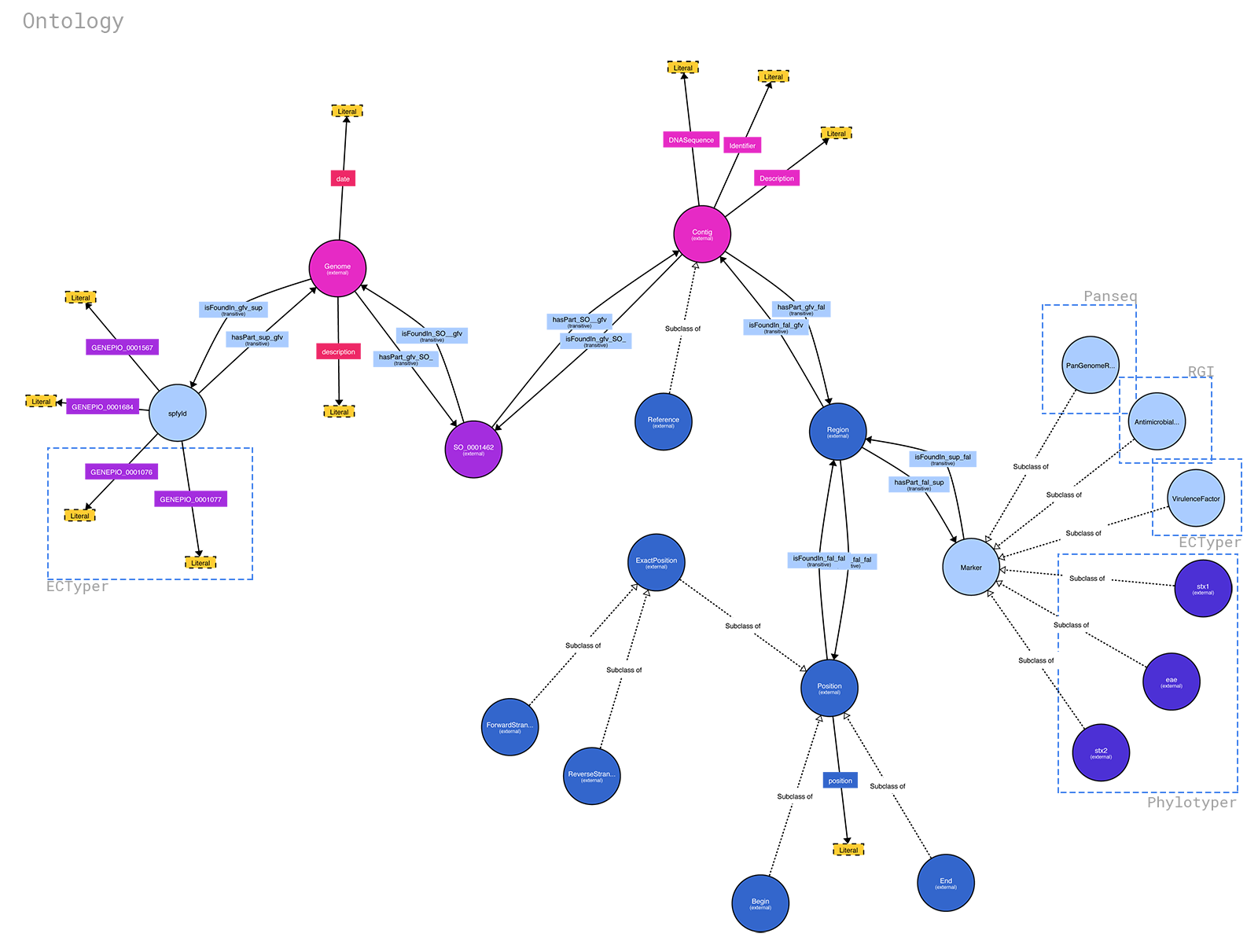
La ontología para spfy está disponible en: https://raw.githubusercontent.com/superphy/backend/master/app/scripts/spfy_ontology.ttl Se generó usando https://raw.githubusercontent.com/superphy/backend/master /app/scripts/generate_ontology.py con funciones compartidas del código backend de spfy . Si desea ejecutarlo, haga: 1. cd app/ 2. python -m scripts/generate_ontology que pondrá la ontología en app/
Puede generar un diagrama bonito a partir del archivo .ttl usando http://www.visualdataweb.de/webvowl/
Nota
actualmente configurado solo para archivos .fna
Puede omitir el sitio web de front-end y aún así poner en cola trabajos de subtipificación de la siguiente manera:
/datastore en los contenedores.Por ejemplo, si guarda sus archivos en
/home/bob/ecoli-genomes/, editará el archivodocker-compose.ymly reemplazará:volumes : - /datastorecon:
volumes : - /home/bob/ecoli-genomes:/datastore
docker-compose down docker-compose up -d
docker exec -it backend_webserver_1 sh python -m scripts/sideload exit
Tenga en cuenta que se pueden crear residuos en la carpeta de su genoma.

| Imagen Docker | Puerto s | Nombres | Descripción |
|---|---|---|---|
| parte trasera- rq | 80/tpc, 443/tcp | back-end_trabajador_1 | los principales trabajadores de la cola de redi s |
| extremo posterior- rq-b laze grap h | 80/tpc, 443/tcp | back end_work kerblaz egra ph-i ds_ 1 | esta mano les spfy relación de genes de identificación para la base de datos de blaz egra ph |
| parte trasera | 0.0. 0,0: 8000 ->80/tcp, 443/tcp | back end_web -ngi nx-u wsgi _1 | la parte trasera del matraz que se encarga de las tareas solicitadas |
| super rphy /bla zegr aph: 2.1. ferenciación de 4 pulgadas | 0.0. 0,0: 8080 ->80 80/t pp | parte trasera_bla zegr aph_1 | Blaz egra ph Base de datos |
| redistribuir:3. 2 | 6379/tcp | back end_red es_ 1 | Base de datos de Redi |
| grifo de reacción | 0.0. 0,0: 8090 ->50 00/t cp | back end_rea ctap p_1 | de frente a spfy |
La imagen superphy/backend-rq:2.0.0 es escalable : puede crear tantas instancias como necesite o tenga potencia de procesamiento. La imagen es responsable de escuchar la cola multiples (12 trabajadores) que maneja la mayoría de las tareas, incluidas las llamadas RGI . También escucha la cola singles (1 trabajador) que ejecuta ECTyper . Esto se hace porque RGI es la parte más lenta de la ecuación. La gestión de trabajadores se maneja en supervisor .
La imagen superphy/backend-rq-blazegraph:2.0.0 no es escalable: es responsable de consultar la base de datos de Blazegraph en busca de entradas duplicadas y de asignar ID spfy en orden secuencial . Sus funciones se mantienen lo más mínimas posible para mejorar el rendimiento (ya que la generación de ID es el único cuello de botella en tuberías que de otro modo serían paralelas); las comparaciones se realizan mediante hash sha1 de los archivos enviados y los no duplicados tienen sus ID reservados vinculando el ID spfy generado al hash del archivo. La gestión de trabajadores se maneja en supervisor .
superphy/backend:2.0.0 que ejecuta los puntos finales de Flask utiliza supervisor para administrar los procesos internos: nginx , uWsgi .
database['blazegraph_url'] en /app/config.py Los pasos necesarios para agregar nuevos módulos están documentados en la Guía para desarrolladores.