Repositorio que contiene datos y código para el genotipado de guar ( Cyamopsis tetragonoloba (L.) Taub.) y el proyecto GWAS. Una breve descripción del contenido del repositorio:
Se utilizó el script make_consensus_vcf.py para generar el conjunto de datos de variantes final utilizando llamadas de variantes GATK-HC, NGSEP y TASSEL 5 sin procesar, y el script make_snp_stats.py se utilizó con las mismas entradas para explorar las estadísticas de calidad de las llamadas de variantes.
El cuaderno callset_refinement_filtering.ipynb se utilizó para ejecutar el control de calidad de las variantes y construir el conjunto de datos filtrado final de las variantes comunes.
El cuaderno genetic_analysis.ipynb se utilizó para realizar la mayoría de los análisis genéticos, incluido PCA, análisis de desequilibrio de ligamiento y análisis de asociación basado en modelos lineales generalizados.
El script prepare_farmcpu.py se utilizó para formatear los datos de entrada para la herramienta FarmCPU (el gráfico QQ de los valores p resultantes se muestra en el artículo y a continuación).
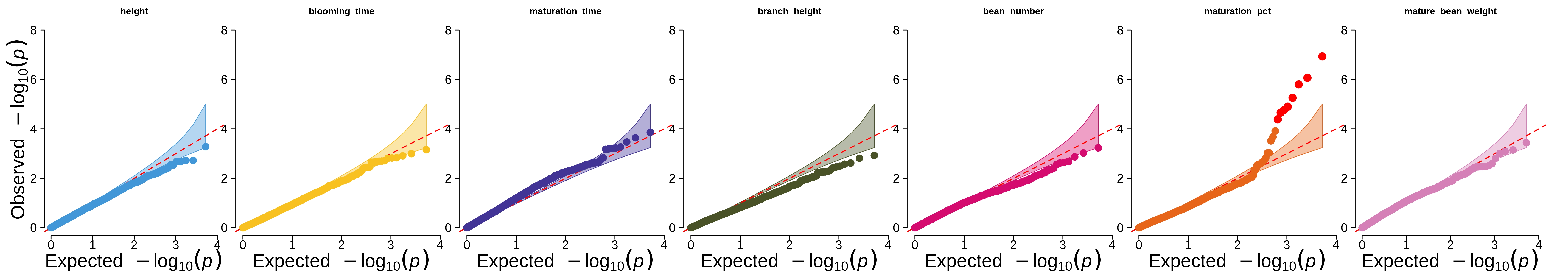
El archivo admix_file_create.py se usó para preprocesar genotipos para el análisis ADMIXTURE (parte de esto se hizo usando Hail en genetic_analysis.ipynb ).
El script guar_stat.R se utilizó para ejecutar análisis estadístico de los datos finales, realizar análisis de asociación de todo el genoma utilizando la herramienta FarmCPU y trazar las cifras principales.
Se utilizó el script parse_vcf_fcpu.py para formatear la tabla final de genotipo SNP disponible como archivo genotypes.xlsx en este repositorio.