MedSegDiff un marco basado en el modelo probabilístico de difusión (DPM) para la segmentación de imágenes médicas. El algoritmo se elabora en nuestro artículo MedSegDiff: Segmentación de imágenes médicas con modelo probabilístico de difusión y MedSegDiff-V2: Segmentación de imágenes médicas basada en difusión con transformador.
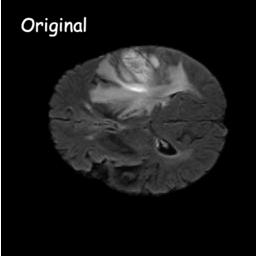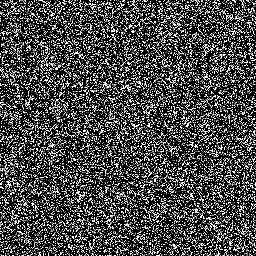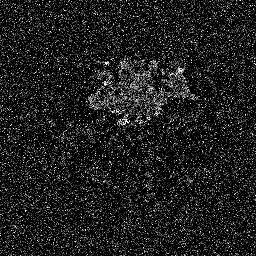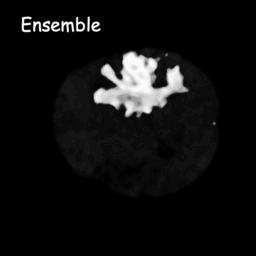 Los modelos de difusión funcionan destruyendo los datos de entrenamiento mediante la adición sucesiva de ruido gaussiano y luego aprendiendo a recuperar los datos invirtiendo este proceso de generación de ruido. Después del entrenamiento, podemos usar el modelo de difusión para generar datos simplemente pasando ruido muestreado aleatoriamente a través del proceso de eliminación de ruido aprendido. En este proyecto, ampliamos esta idea a la segmentación de imágenes médicas. Utilizamos la imagen original como condición y generamos múltiples mapas de segmentación a partir de ruidos aleatorios, luego los ensamblamos para obtener el resultado final. Este enfoque captura la incertidumbre en las imágenes médicas y supera a los métodos anteriores en varios puntos de referencia.
Los modelos de difusión funcionan destruyendo los datos de entrenamiento mediante la adición sucesiva de ruido gaussiano y luego aprendiendo a recuperar los datos invirtiendo este proceso de generación de ruido. Después del entrenamiento, podemos usar el modelo de difusión para generar datos simplemente pasando ruido muestreado aleatoriamente a través del proceso de eliminación de ruido aprendido. En este proyecto, ampliamos esta idea a la segmentación de imágenes médicas. Utilizamos la imagen original como condición y generamos múltiples mapas de segmentación a partir de ruidos aleatorios, luego los ensamblamos para obtener el resultado final. Este enfoque captura la incertidumbre en las imágenes médicas y supera a los métodos anteriores en varios puntos de referencia.
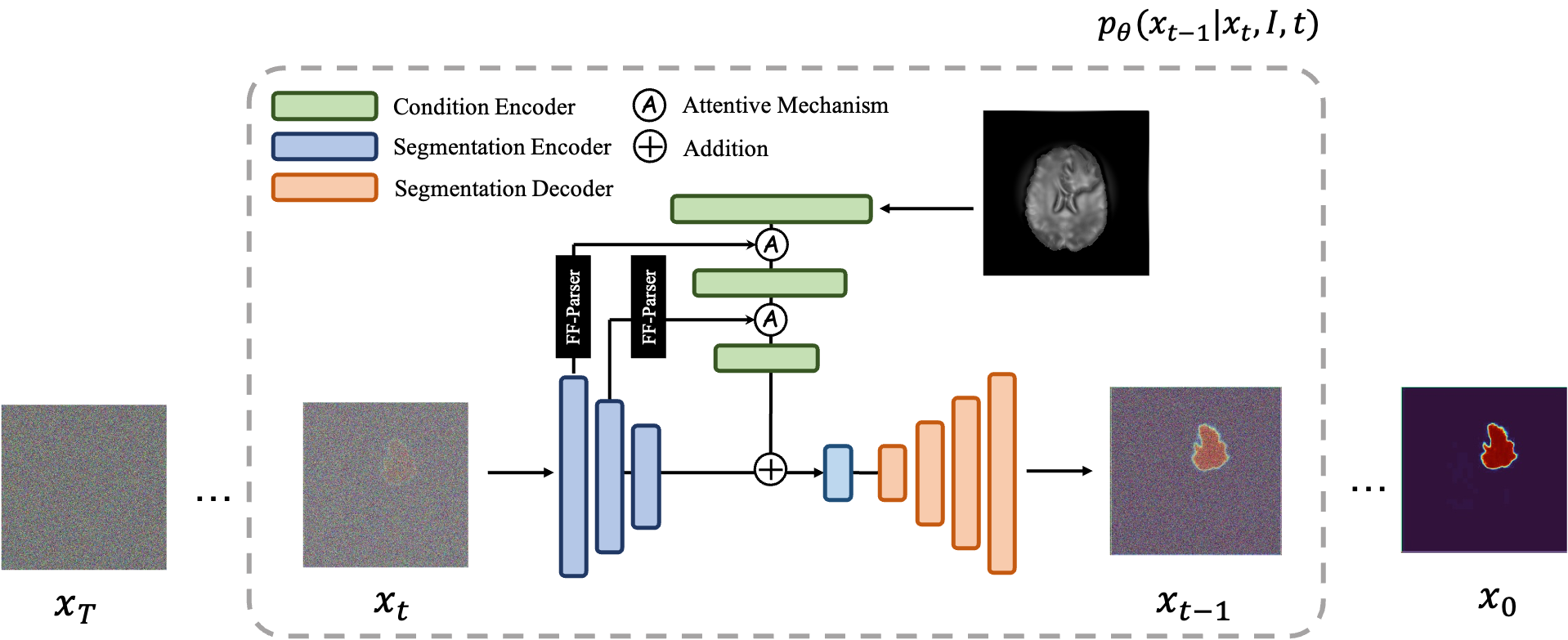 | 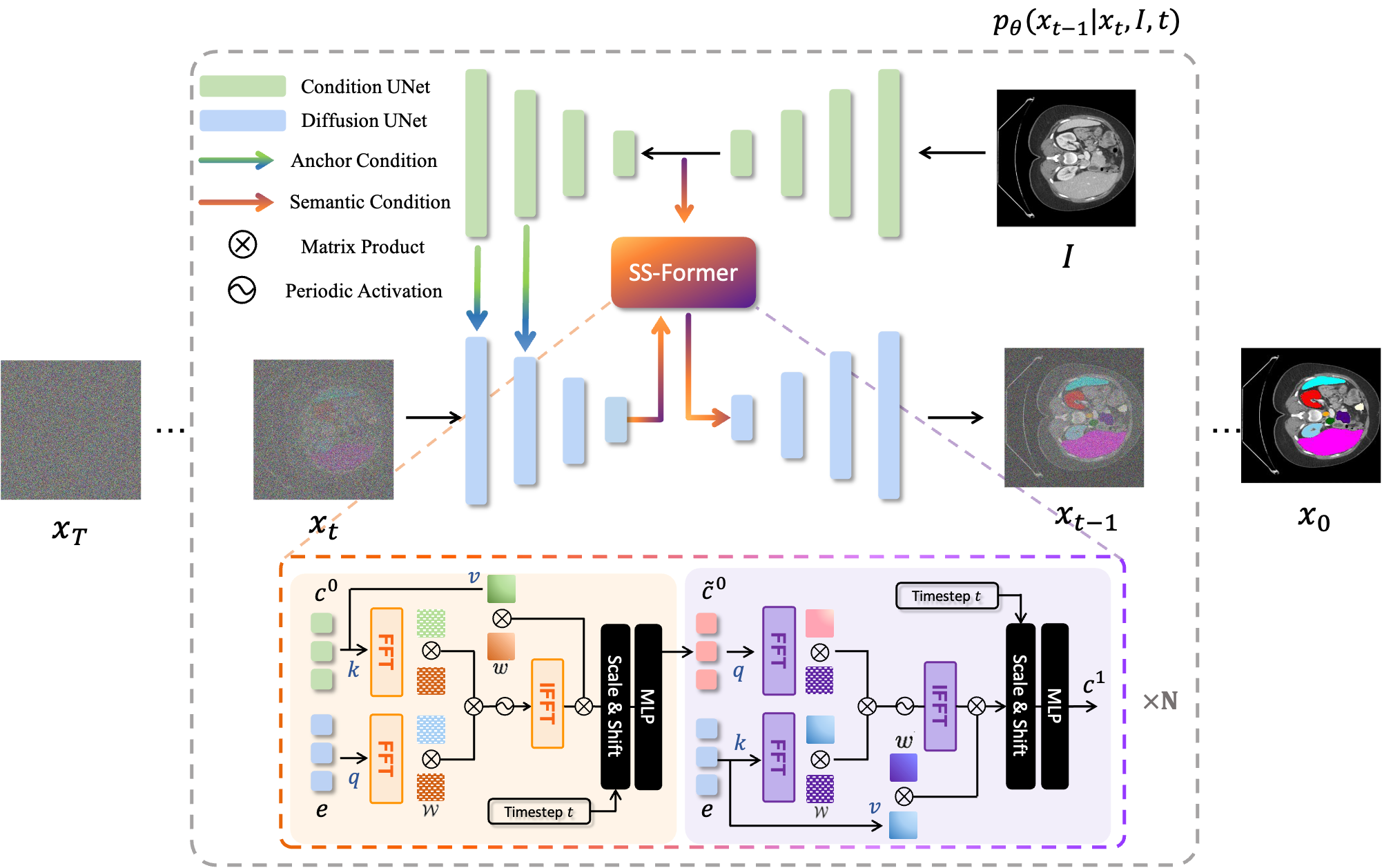 |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True .python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
Para el entrenamiento, ejecute: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Para el muestreo, ejecute: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
Para la evaluación, ejecute python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
De forma predeterminada, las muestras se guardarán en ./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
Para entrenar, ejecute: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Para el muestreo, ejecute: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
Es sencillo ejecutar MedSegDiff en los otros conjuntos de datos. Simplemente escriba otro archivo de carga de datos después de ./guided_diffusion/isicloader.py o ./guided_diffusion/bratsloader.py . Bienvenido a temas abiertos si encuentra algún problema. Le agradeceríamos que pudiera contribuir con las extensiones de su conjunto de datos. A diferencia de las imágenes naturales, las imágenes médicas varían mucho según las distintas tareas. Ampliar la generalización de un método requiere el esfuerzo de todos.
Para entrenar un modelo fino, es decir, MedSegDiff-B en el documento, establezca los hiperparámetros del modelo como:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
hiperparámetros de difusión como:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
Para acelerar el muestreo:
--diffusion_steps 50 --dpm_solver True
ejecutar en múltiples GPU:
--multi-gpu 0,1,2 (for example)
hiperparámetros de entrenamiento como:
--lr 5e-5 --batch_size 8
y establezca --num_ensemble 5 en el muestreo.
La ejecución de alrededor de 100.000 pasos en el entrenamiento convergerá en la mayoría de los conjuntos de datos. Tenga en cuenta que, aunque la pérdida no disminuirá en la mayoría de los pasos posteriores, la calidad de los resultados sigue mejorando. Este proceso también se observa en otras aplicaciones DPM, como la generación de imágenes. Espero que alguien inteligente pueda decirme por qué.
Pronto publicaré su rendimiento en un tamaño de lote más pequeño (adecuado para ejecutarse en una GPU de 24 GB) por si es necesario realizar una comparación.
Una configuración para liberar todo su potencial es (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
Luego entrénelo con el tamaño de lote --batch_size 64 y pruébelo con el número de conjunto --num_ensemble 25 .
Bienvenido a contribuir a MedSegDiff. Se agradece cualquier técnica que pueda mejorar el rendimiento o acelerar el algoritmo. Estoy escribiendo MedSegDiff V2, con el objetivo de publicar en revistas Nature/CVPR. ¿Me alegra incluir a los contribuyentes como mis coautores?
Código copiado mucho de openai/improved-diffusion, WuJunde/ MrPrism, WuJunde/ DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Diffusion-based-Segmentation, hojonathanho/diffusion, guiado-difusión, bigmb/Unet-Segmentation-Pytorch-Nest -de-Unets, nnUnet, lucidrains/vit-pytorch
Por favor cita
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu