med seg diff pytorch
0.3.3
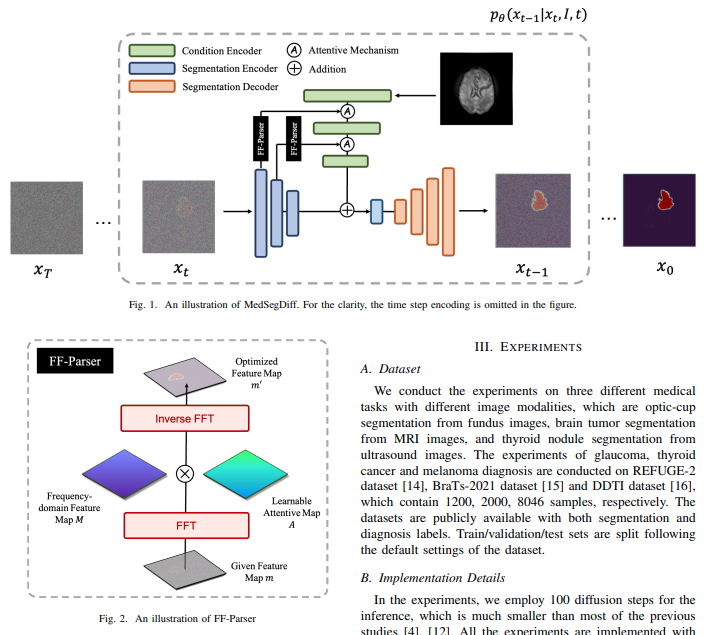
Implementación de MedSegDiff en Pytorch: segmentación médica SOTA fuera de Baidu utilizando DDPM y acondicionamiento mejorado a nivel de funciones, con filtrado de funciones en el espacio de Fourier.
StabilityAI por el generoso patrocinio, así como a mis otros patrocinadores.
¡Isamu y Daniel por agregar un guión de capacitación para un conjunto de datos de lesiones cutáneas!
$ pip install med-seg-diff-pytorch import torch
from med_seg_diff_pytorch import Unet , MedSegDiff
model = Unet (
dim = 64 ,
image_size = 128 ,
mask_channels = 1 , # segmentation has 1 channel
input_img_channels = 3 , # input images have 3 channels
dim_mults = ( 1 , 2 , 4 , 8 )
)
diffusion = MedSegDiff (
model ,
timesteps = 1000
). cuda ()
segmented_imgs = torch . rand ( 8 , 1 , 128 , 128 ) # inputs are normalized from 0 to 1
input_imgs = torch . rand ( 8 , 3 , 128 , 128 )
loss = diffusion ( segmented_imgs , input_imgs )
loss . backward ()
# after a lot of training
pred = diffusion . sample ( input_imgs ) # pass in your unsegmented images
pred . shape # predicted segmented images - (8, 3, 128, 128) Comando para ejecutar
accelerate launch driver.py --mask_channels=1 --input_img_channels=3 --image_size=64 --data_path= ' ./data ' --dim=64 --epochs=100 --batch_size=1 --scale_lr --gradient_accumulation_steps=4Si desea agregar autocondición donde condicionamos con la máscara que tenemos hasta ahora, haga --self_condition
@article { Wu2022MedSegDiffMI ,
title = { MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model } ,
author = { Junde Wu and Huihui Fang and Yu Zhang and Yehui Yang and Yanwu Xu } ,
journal = { ArXiv } ,
year = { 2022 } ,
volume = { abs/2211.00611 }
} @inproceedings { Hoogeboom2023simpleDE ,
title = { simple diffusion: End-to-end diffusion for high resolution images } ,
author = { Emiel Hoogeboom and Jonathan Heek and Tim Salimans } ,
year = { 2023 }
}