
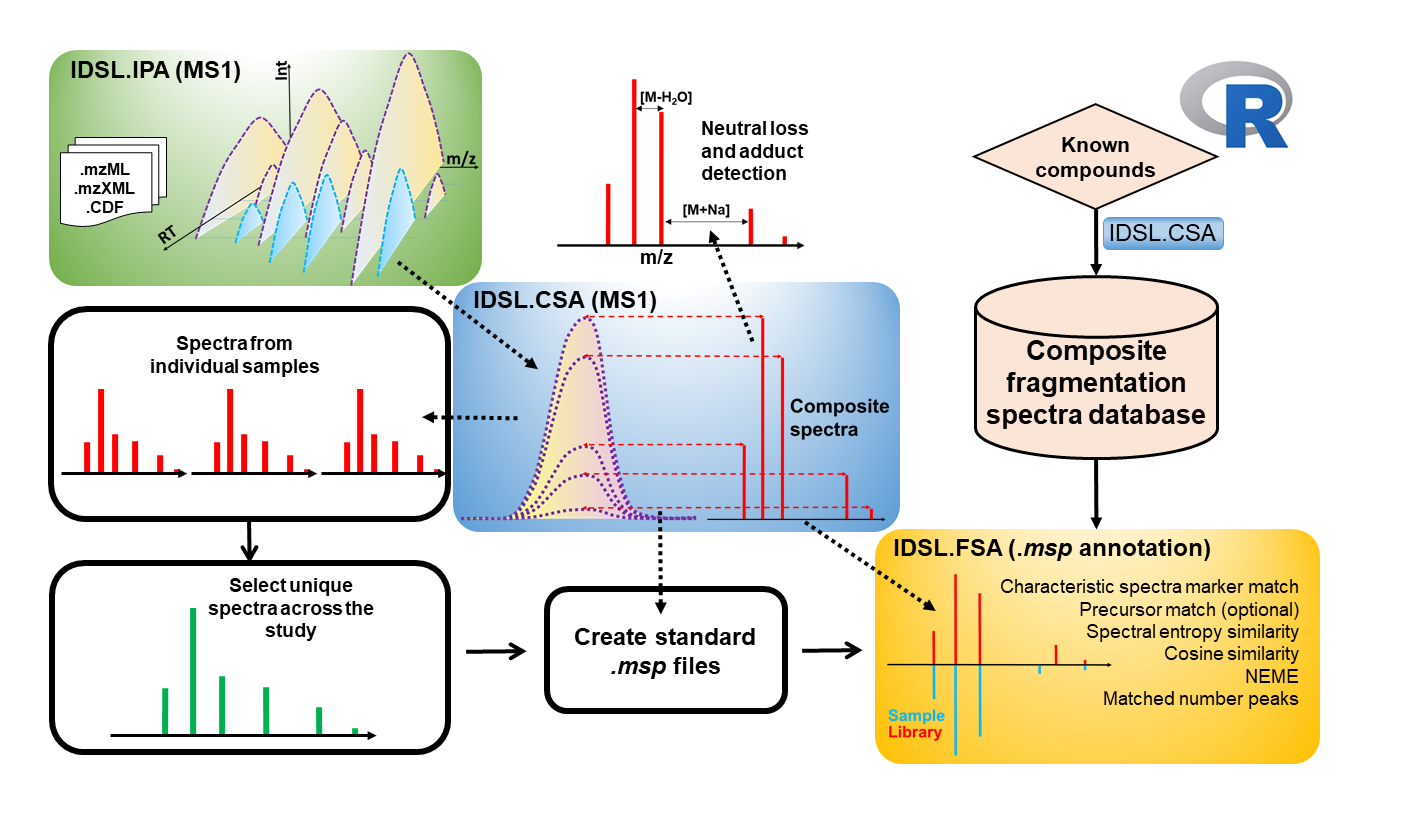
El Paquete R Composite Spectra (IDSL.CSA) R para el análisis de datos de espectrometría de masas ha sido desarrollado por el Laboratorio Integrado de Ciencia de Datos para Metabolómica y Exposómica (IDSL.ME) . Este paquete se puede utilizar para la deconvolución de los espectros de fragmentación obtenidos a través de varios métodos analíticos, como el análisis de deconvolución de espectros compuestos ( CSA ) de solo MS1, la adquisición dependiente de datos ( DDA ) y una adquisición de adquisición ( DIA ) independiente de datos que incluyen MS E MS E , Fragmentación de todas las iones (AIF) y análisis SWATH-MS. El objetivo del paquete IDSL.CSA es ayudar a optimizar el proceso de análisis de datos y mejorar la anotación general de la estructura química en los campos de metabolómica y exposómica.
Start en la hoja de cálculo de parámetros IDSL.CSA. install.packages("IDSL.CSA")
Antes de procesar sus datos de espectrometría de masas ( MZXML , MZML , NETCDF ) utilizando el flujo de trabajo IDSL.CSA, los datos de espectrometría de masas deben procesarse utilizando el flujo de trabajo IDSL.IPA para adquirir información cromatográfica de los picos ( M/Z-RT ). Cuando la información cromatográfica de las listas pico alineadas individuales y agregadas se generó utilizando el flujo de trabajo IDSL.IPA, descargue la hoja de cálculo de parámetros IDSL.CSA y seleccione los parámetros en consecuencia y luego use esta hoja de cálculo como la entrada para el flujo de trabajo IDSL.CSA:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Siga estos pasos para un estudio de caso rápido (n = 33) ST002263 que tiene datos de orbitrap híbrido HF Thermo Q Exactives recopilados en los modos HILIC-ESI-POS/NEG.
Procesar datos de espectrometría de masas sin procesar e información cromatográfica utilizando el método descrito para IDSL.IPA
El análisis de espectros compuestos requiere 39 parámetros distribuidos en 5 secciones separadas para un análisis de escala completa. Para este estudio, use los valores de parámetros predeterminados presentados en la hoja de cálculo de parámetros IDSL.CSA. A continuación, proporcione información para
2.1. Seleccione Sí para Param0001 en la pestaña Start para procesar solo el flujo de trabajo CSA .
2.2. CSA0005 para la dirección de ubicación de datos HRMS (datos de HRMS de nivel MS1)
2.3. CSA0008 para la dirección del directorio peaklists generado por el flujo de trabajo IDSL.IPA
2.4. CSA0009 para la dirección del directorio peak_alignment generado por el flujo de trabajo IDSL.IPA
2.5. CSA0011 para la ubicación de salida (archivos .msp y EICS)
2.6. También puede aumentar el número de subprocesos de procesamiento utilizando CSA0004 de acuerdo con su energía computacional
Ejecute este comando en la consola o terminal R/RStudio:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Puede analizar los resultados en la dirección que proporcionó para CSA0011 .
4.1. CSA_MSP incluye el archivo .msp
4.2. CSA_ADDUCT_ANNOTATION incluye listas pico con información de aducto potencial
4.3. Peak_alignment_subset incluye subconjuntos de tablas máximas alineadas para los iones principales en cada clúster CSA
4.4. Aligned_spectra_table incluye información para la agregación CSA en la tabla alineada
[1] Fakouri Baygi, S., Kumar, Y. Barupal, DK IDSL.CSA: Análisis de espectros compuestos para anotación química de conjuntos de datos de metabolómica no dirigidos. Analítica Química , 2023 , 95 (25) , 9480–9487.
[2] Fakouri Baygi, S., Kumar, Y. Barupal, DK Idsl. IPA caracteriza el espacio químico orgánico en conjuntos de datos LC/HRMS no dirigidos. Journal of Proteome Research , 2022 , 21 (6) , 1485-1494.