
Tenga en cuenta que el código de Poseidón se acaba de transferir a NextFlow para que todavía haya algunos errores. ¡No dude en informar problemas!
Aquí presentamos Poseidón, una tubería para detectar sitios seleccionados significativamente positivos y posibles eventos de recombinación en una alineación de múltiples secuencias de codificación de proteínas. Los sitios que experimentan una selección positiva proporcionan ideas en la historia evolutiva de sus secuencias, por ejemplo, mostrando importantes puntos calientes de mutación, acumulados como resultados de las carreras de armas de huésped de virus durante la evolución.
Poseidon se basa en una variedad de diferentes herramientas de terceros (ver más abajo). Pero no se preocupe, encapsulamos cada herramienta en su propio contenedor Docker y los conectamos en el sistema de administración de flujo de trabajo NextFlow.
Vaya directamente a un pequeño ejemplo de salida de Poseidón para la proteína SARS-CoV-2 Spike en comparación con un estudio reciente de Zhou et al . 2020.
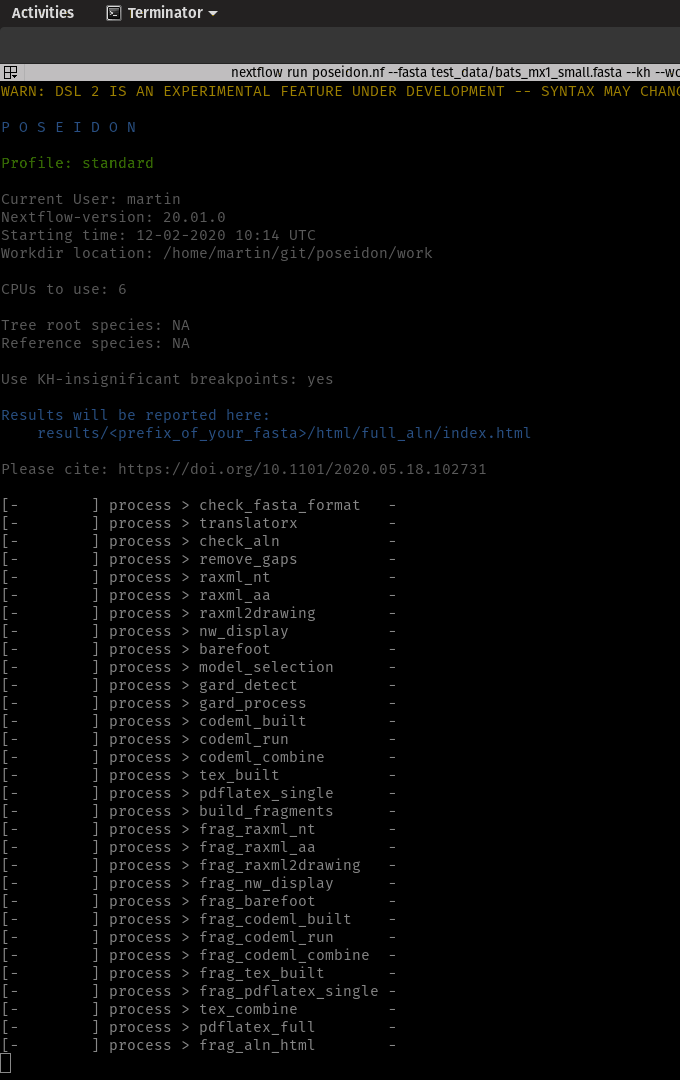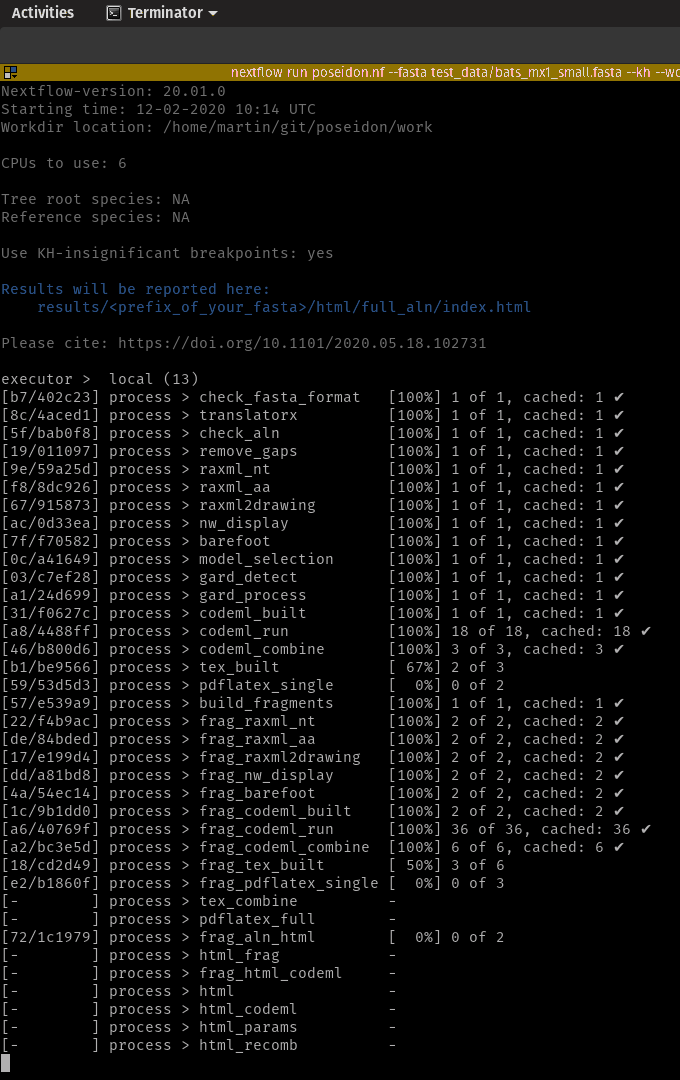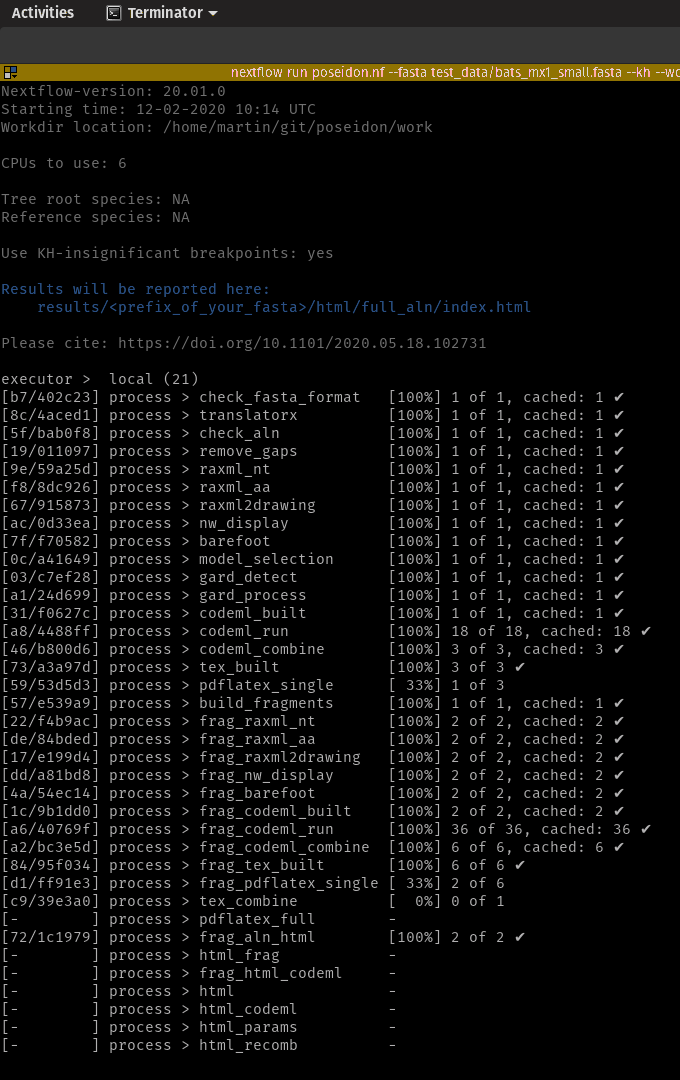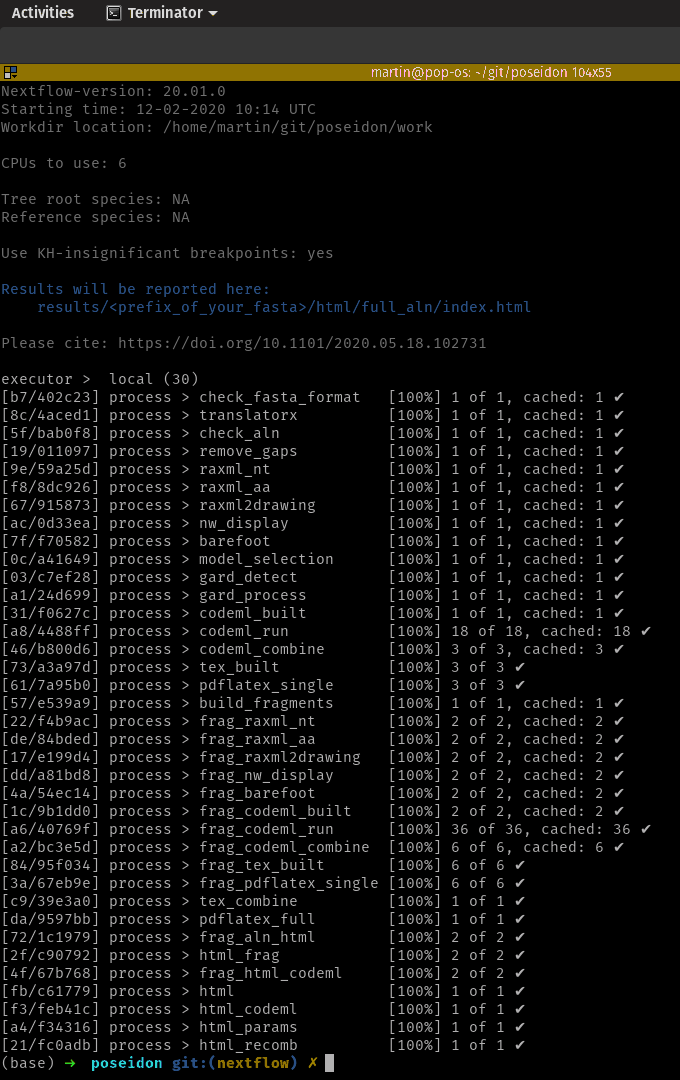
Solo necesita NextFlow (versión 20.+) y Docker instalados para ejecutar la tubería. Todas las dependencias se extraerán automáticamente.
O ejecute Poseidon clonando este repositorio:
clon git https://github.com/hoelzer/posidon.gitcd Poseidon NextFlow Run Poseidon.nf --help
O deja que Nextflow haga el tirón
NextFlow Pull Hoelzer/Poseidón
Recomendamos usar una versión específica de Poseidón a través de
#PullNextFlow Pull Hoelzer/Poseidon -R V1.0.1#RunNextflow Run Hoelzer/Poseidon -R V1.0.1 -Help
Dependiendo de su procedimiento de instalación, actualice la tubería a través de git pull o nextflow pull hoelzer/poseidon .
Importante: Poseidon necesita secuencias de nucleótidos con un marco de lectura abierto correcto como entrada. Además, los resultados dependen en gran medida de su selección de secuencias, por lo tanto, puede considerar ejecutar la tubería varias veces con diferentes muestras de sus secuencias de entrada. Además, la tubería no puede funcionar con demasiadas secuencias porque en su núcleo Poseidón usa CodeMl de la suite PAML que no está destinada a> 100 secuencias. Encuentre una descripción detallada de los parámetros y configuraciones de entrada a continuación.
NextFlow se puede ejecutar fácilmente en diferentes entornos como su máquina local, un clúster de alto rendimiento o la nube. Se utilizan diferentes -profile para decirle a NextFlow qué sistema debe usarse. Para la ejecución local -profile local,docker debe usarse (y también es el valor predeterminado). También puede ejecutar Poseidon en un HPC usando singularidad a través de -profile lsf,singularity , -profile slurm,singularity o -profile sge,singularity . En tales casos, también considere ajustar --cachedir para apuntar dónde almacenar imágenes de singularidad en su clúster. El parámetro --workdir también podría ser útil para ajustar dónde almacenar directorios de trabajo temporales (por ejemplo, uso /scratch en lugar de /tmp dependiendo de su configuración de HPC).
Ahora supongamos que usó NextFlow para extraer el código Poseidon y ejecuta la tubería en una máquina local utilizando el perfil predeterminado -profile local,docker .
# Show Help Ayuda Nextflow Run Hoelzer/Poseidon -Help # Ejecutar un pequeño ejemplo en una máquina local con # (la primera vez que esto necesitará más tiempo porque los contenedores Docker se descargan) Nextflow Run Hoelzer/Poseidon -R V1.0.1 -Fasta ~/.nextflow/assets/hoelzer/Poseidon/test_data/bats_mx1_small.fastA - -CORE 4# Reanudar un runnextflow roto run Hoelzer/Poseidon -r v1.0.1 --fasta ~/.nextflow/assets/hoelzer/Poseidon/test_data/bats_mx1_small.fasta - -CORES 4 -RESUME# en lugar de usar todos los núcleos disponibles solo use una cantidad máxima en el MachinExtflow local Run Hoelzer/Poseidon -R V1.0.1 -Fasta ~/.Nextflow/Assets/Hoelzer/Poseidon/Test_Data/BATS_MX1_SMALL.FASTA --max_cores 8 -Corres 4
Reproducir los resultados de selección positivos informados en Fuchs et al . (2017), Journal of Virology Run:
NextFlow Run Hoelzer/Poseidon -R V1.0.0 --fasta ~/.nextflow/assets/hoelzer/Poseidon/test_data/bats_mx1.fasta --cores 4 --kh --outgroup "PTEROPUS_ALECTO, EIDOLON_HELVUM, Rousettus_Aegyptiacus, Hypsignatus_Monstrosus"-Reference "Myotis_daubentonii"
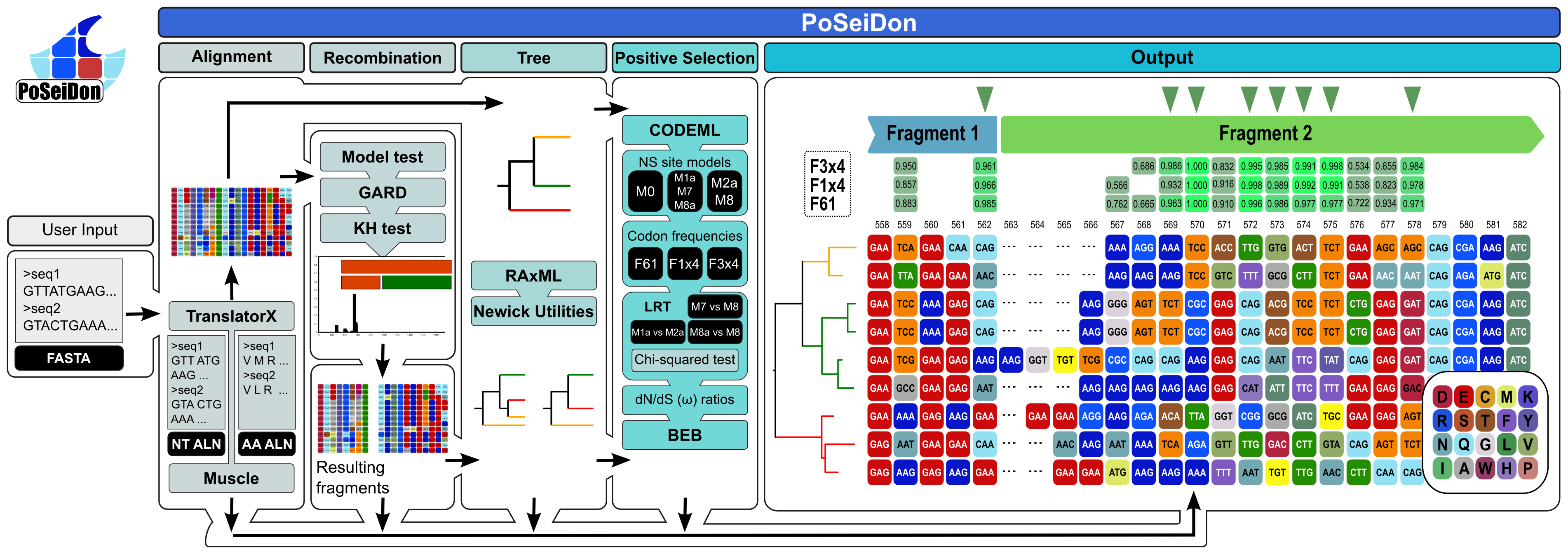
La tubería de Poseidon comprende la alineación en el marco de secuencias de codificación de proteínas homólogas, la detección de eventos de recombinación supuestos y puntos de interrupción evolutivos, reconstrucciones filogenéticas y detección de sitios seleccionados positivamente en la alineación completa y todos los fragmentos posibles. Finalmente, todos los resultados se combinan y visualizan en una página web HTML fácil y fácil de usar. Los fragmentos de alineación resultantes se indican con barras de color en la salida HTML.
TranslatorX (V1.1), Abascal et al . (2010); 20435676
Músculo (V3.8.31), Edgar (2004); 15034147
RAXML (V8.0.25), Stamatakis (2014); 24451623
Newick Utilities (V1.6), Junier y Zdobnov (2010); 20472542
Modeltest, Posada y Crandall (1998); 9918953
Hyphy (V2.2), Pond et al . (2005); 15509596
Gard, Pond et al. (2006); 17110367
Paml/Codeml (V4.8), Yang (2007); 17483113
Ruby (v2.3.1)
Inkscape (V1.0)
PDFTEX (v3.14)
La mayoría de los parámetros de Poseidon son opcionales y se explican a continuación en detalle.
--fasta
Obligatorio. Su archivo de entrada FASTA debe seguir el formato:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA... >Myotis_davidii Mx1 Gene ATGGCGGTCGAGATAAGATACGTT...
Todas las secuencias deben tener un marco de lectura abierto correcto, solo se les permite contener caracteres de nucleótidos [A | C | G | T] y sin codón de parada interna.
Las ID de secuencia deben ser únicas hasta la primera ocurrencia de un espacio.
--reference
Opcional. Valor predeterminado: use la primera ID de secuencia como referencia. Puede definir una ID de especie de su archivo FASTA múltiple como especie de referencia. Los sitios seleccionados positivamente y los aminoácidos correspondientes se dibujarán con respecto a esta especie. La identificación debe coincidir con el encabezado FASTA hasta la ocurrencia del primer espacio. Por ejemplo, si desea Myotis Lucifugus como su especie de referencia y su archivo FASTA contiene:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA...
usar
--reference "Myotis_lucifugus"
como parámetro para establecer la especie de referencia. Por defecto, se utilizará la primera ID en el archivo FASTA múltiple.
--outgroup
Opcional. Valor predeterminado: los árboles están sin rooted. Puede definir una o múltiples identificaciones de especies (separadas por comas) como grupo externo. Todos los árboles filogenéticos estarán enraizados de acuerdo con esta especie. Por ejemplo, si su archivo FASTA múltiple contiene
ATGGCGATCGAGATACGATACGTA...
>Myotis_davidii Mx1 Gene
ATGGCGGTCGAGATAAGATACGTT...
>Pteropus_vampyrus Mx1 Gene
ATGGCCGTAGAGATTAGATACTTT...
>Eidolon_helvum Mx1 Gene
ATGCCCGTAGAGAATAGATACTTT...Puedes definir:
--outgroup "Pteropus_vampyrus,Eidolon_helvum"
Para rootear todos los árboles en relación con estas dos especies.
--kh
Opcional. Predeterminado: falso. Con este parámetro, puede decidir si se deben tener en cuenta los puntos de interrupción insignificantes. Todos los puntos de interrupción se prueban para una incongruencia topológica significativa utilizando una prueba de Kashino Hasegawa (KH) Kishino, H. y Hasegawa, M. (1989). Los puntos de interrupción insignificantes de KH surgen con mayor frecuencia de la variación en las longitudes de la rama entre segmentos. Sin embargo, tener en cuenta los puntos de ruptura insignificantes de KH podría ser interesante, porque ya observamos sitios putativos seleccionados positivamente en fragmentos sin ninguna incongruencia topológica significativa. Los fragmentos insignificantes de KH están marcados en la salida final, ya que podrían no ocurrir a partir de eventos reales de recombinación.
Por defecto, solo se usan puntos de interrupción significativos para cálculos adicionales.
También tenga en cuenta que usar puntos de ruptura también insignificantes puede extender el tiempo de ejecución de Poseidón de minutos a horas, dependiendo de la cantidad de puntos de interrupción detectados.
¡Consulte el --help para otros parámetros (Gard, RAXML, ...) y háganos saber si necesita más personalización!
Si Poseidon te ayuda, por favor citar:
Martin Hölzer y Manja Marz, "Poseidon: una tubería NextFlow para la detección de eventos evolutivos de recombinación y selección positiva", OUP Bioinformatics (2020)