Nos complace anunciar que el tamaño del índice en el disco está inactivo por 8 veces y en la memoria 4 veces debido a pasar a un solo tipo de índice FM (2bit.64 en lugar de 2 bits.64 y 8bit.32) y 8x compresión de matriz de sufijo. Por ejemplo, para el genoma humano, el tamaño del índice en el disco se redujo a ~ 10 GB de ~ 80GB y la huella de memoria se redujo a ~ 10 GB de ~ 40GB. Existe una reducción sustancial en el tiempo de índice IO debido a la reducción y apenas cualquier impacto de rendimiento en el mapeo de lectura. Debido a este cambio en la estructura del índice (en Commit #4B59796, 10 de octubre de 2020), deberá reconstruir el índice.
Se agregó el indicador MC en el archivo SAM de salida en Commit A591E22. La salida debe coincidir con la versión original de BWA-MEM 0.7.17.
A partir de Commit E0AC59E, tenemos un submódulo Git SafeStringlib. Para obtenerlo, use -Recursivo al clonar o usar "Git Submodule Init" y "Git Submodule Update" en un repositorio ya clonado (ver más abajo para obtener más detalles).
# Use binarios precompilados (recomendado) curl -l https://github.com/bwa-mem2/bwa-mem2/releases/download/v2.2.1/bwa-mem2-2.2.1_x64-linux.tar.bz2 | tar JXF - BWA-MEM2-2.2.1_X64-Linux/BWA-MEM2 ÍNDICE REF.FA BWA-MEM2-2.2.1_X64-Linux/BWA-MEM2 MEM Ref.FA Read1.fq Read2.FQ> OUT.SAM# Compilar desde la fuente (no recomendado para usuarios generales)# Obtenga el clon de SourceGit-https recursivo: // github.com/bwa-mem2/bwa-mem2cd bwa-mem2# orgit clon https://github.com/bwa-mem2/bwa-mem2cd bwa-mem2 git submodule init Git Submodule Update# compilar y runmake ./BWA-MEM2
La herramienta BWA-MEM2 es la próxima versión del algoritmo BWA-MEM en BWA. Produce una alineación idéntica a BWA y es ~ 1.3-3.1x más rápido dependiendo del caso de uso, el conjunto de datos y la máquina en ejecución.
El BWA original fue desarrollado por Heng Li (@LH3). La mejora del rendimiento en BWA-MEM2 fue realizada principalmente por Vasimuddin MD (@YUK12) y Sanchit Misra (@Sanchit-Misra) del laboratorio de computación paralelo, Intel. BWA-MEM2 se distribuye bajo la licencia MIT.
Para los usuarios generales, se recomienda usar los binarios precompilados desde la página de lanzamiento. Estos binarios se compilaron con el compilador Intel y funcionan más rápido que los binarios compilados por GCC. Los binarios precompilados también admiten indirectamente el envío de CPU. El binario bwa-mem2 puede elegir automáticamente la implementación más eficiente basada en el conjunto de instrucciones SIMD disponible en la máquina en ejecución. Los binarios precompilados se generaron en una máquina CentOS7 utilizando la siguiente línea de comando:
hacer cxx = ICPC multi
El uso es exactamente el mismo que la herramienta BWA MEM original. Aquí hay una breve sinopsis. Ejecutar ./bwa-mem2 para comandos disponibles.
# Indexación de la secuencia de referencia (requiere una memoria de 28n Gb donde n es el tamaño de la secuencia de referencia) ../ BWA-MEM2 ÍNDICE [-P prefijo] <in.fast> donde <in.fasta> es la ruta para la secuencia de referencia FISTA FILE y <Prefix> es el prefijo de los nombres de los archivos que almacenan el índice resultante. Predeterminado es in.fasta. # Mapeo # ejecutar "./bwa-mem2 mem" para obtener todas las opciones ./bwa-mem2 mem -t <num_threads> <prefix> <reads.fq/fa >> out.sam Donde <prefijo> es el prefijo especificado cuando se crea el índice o la ruta al archivo FASTA de referencia en caso de que no se proporcione ningún prefijo.
Conjuntos de datos:
Genoma de referencia: Human_G1K_V37.FASTA
| Alias | Fuente del conjunto de datos | No. de lecturas | Longitud de lectura |
|---|---|---|---|
| D1 | Instituto amplio | 2 x 2.5m BP | 151bp |
| D2 | SRA: SRR7733443 | 2 x 2.5m BP | 151bp |
| D3 | SRA: SRR9932168 | 2 x 2.5m BP | 151bp |
| D4 | SRA: SRX6999918 | 2 x 2.5m BP | 151bp |
Detalles de la máquina:
Procesador: Intel (R) Xeon (R) 8280 CPU @ 2.70GHz
OS: Centro Linux Release 7.6.1810
Memoria: 100 GB
Seguimos los pasos a continuación para recopilar los resultados de rendimiento:
A. Pasos de descarga de datos:
Descargue SRA Toolkit de https://trace.ncbi.nlm.nih.gov/traces/sra/sra.cgi?view=software#header-global
tar xfzv sratoolkit.2.10.5-centos_linux64.tar.gz
Descargar D2: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump --split-files srr7733443
Descargar d3: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump --split-files srr9932168
Descargar d4: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump --split-files srx699918
B. Pasos de alineación:
clon git https://github.com/bwa-mem2/bwa-mem2.git
CD BWA-MEM2
make CXX=icpc (usando el compilador Intel C/C ++)
o make (usando el compilador GCC)
./BWA-MEM2 ÍNDICE <Ref.FA>
./BWA-MEM2 MEM [-T <#Threads>] <Ref.fa> <in_1.fastq> [<in_2.Fastq>]> <Output.sam>
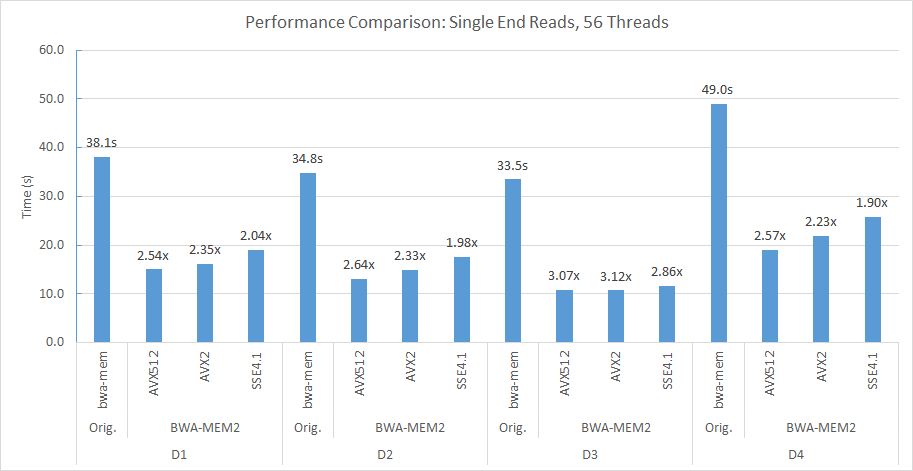
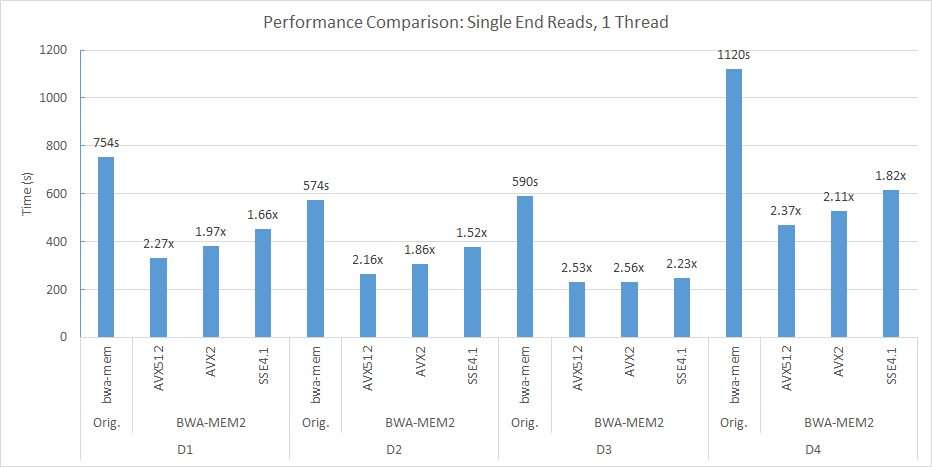
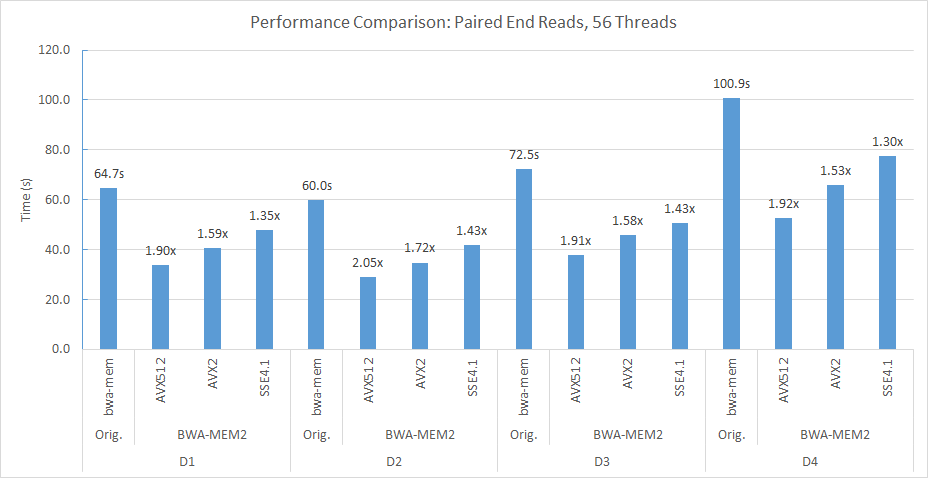
Por ejemplo, en nuestro socket doble (56 subprocesos cada uno) y el nodo de cómputo de doble NUMA, utilizamos la siguiente línea de comandos para alinear D2 a Human_G1K_V37.FASTA DE REFERENCIA DE REFERENCIA.
numactl -m 0 -C 0-27,56-83 ./bwa-mem2 index human_g1k_v37.fasta numactl -m 0 -C 0-27,56-83 ./bwa-mem2 mem -t 56 human_g1k_v37.fasta SRR7733443_1.fastq SRR7733443_2.fastq > d2_align.sam

BWA-MEM2-Lisa es una versión acelerada de BWA-MEM2, donde aplicamos los índices aprendidos a la fase de siembra. La rama BWA-MEM2-LISA contiene el código fuente de la implementación. Las siguientes son las características de BWA-MEM2-Lisa:
Exactamente la misma salida que BWA-MEM2.
Todas las líneas de comandos para crear un índice y la asignación de lectura son exactamente los mismos que BWA-MEM2.
BWA-MEM2-LISA acelera la fase de siembra (uno de los principales cuellos de botella en BWA-MEM2) por hasta 4.5x en comparación con BWA-MEM2.
La huella de memoria del índice BWA-MEM2-LISA es ~ 120 GB para el genoma humano.
El código está presente en BWA-MEM2-Lisa Branch: https://github.com/bwa-mem2/bwa-mem2/tree/bwa-mem2-lisa
La rama ERT del repositorio BWA-MEM2 contiene la base de código de la aceleración basada en el árbol de radix enuerado de BWA-MEM2. El código ERT está construido en la parte superior de BWA-MEM2 (gracias al arduo trabajo de @Arun-Sub). Los siguientes son los aspectos más destacados de la herramienta BWA-MEM2 basada en ERT:
Exactamente la misma salida que BWA-MEM (2)
La herramienta tiene dos indicadores adicionales para habilitar el uso de la solución ERT (para la creación y mapeo de índices), de lo contrario se ejecuta en el modo de vainilla BWA-MEM2
Utiliza 1 indicador adicional para crear ERT Índice (diferente del índice BWA-MEM2) y 1 indicador adicional para usar ese índice ERT (consulte el ReadMe de ERT Branch)
La solución ERT es 10% -30% más rápida (probada en la configuración de la máquina anterior) en comparación con Vanilla BWA -MEM2 -Se recomienda a los usuarios que usen la opción -K 1000000 para ver las aceleraciones
La impresión de la memoria de la memoria del índice ERT es de ~ 60 GB
El código está presente en Ert Branch: https://github.com/bwa-mem2/bwa-mem2/tree/ert
Vasimuddin MD, Sanchit Misra, Heng Li, Srinivas Aluru. Aceleración eficiente de arquitectura de BWA-MEM para sistemas multinúcleo. Simposio de procesamiento paralelo y distribuido de IEEE (IPDPS), 2019. 10.1109/IPDPS.2019.00041