
Isling es una herramienta para detectar la integración viral o vectorial en lecturas pareadas. Lea nuestro artículo para obtener detalles completos.
Si tiene instalados conda y snakemake , para ejecutar con los datos de prueba (incluidos) localmente:
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
Si tiene instalado snakemake y singularity , puede usar en su lugar:
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
Alternativamente, si tiene Docker instalado, en MacOS puede ejecutar:
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
Esto utilizará el archivo de configuración y los datos dentro del contenedor, y los resultados aparecerán en una carpeta out en su directorio de trabajo actual. En Linux, deberá ejecutar este comando como root, y en Windows deberá ajustar la sintaxis de montaje de enlace ( -v argumento).
Los datos de entrada (lecturas y referencias de host y virales) se especifican ni un archivo de configuración: para sus propios datos, deberá modificar el archivo de configuración de ejemplo ( test/config/test.yml ) para señalar sus propios datos. Consulte configfile.md para obtener más información sobre el formato del archivo de configuración.
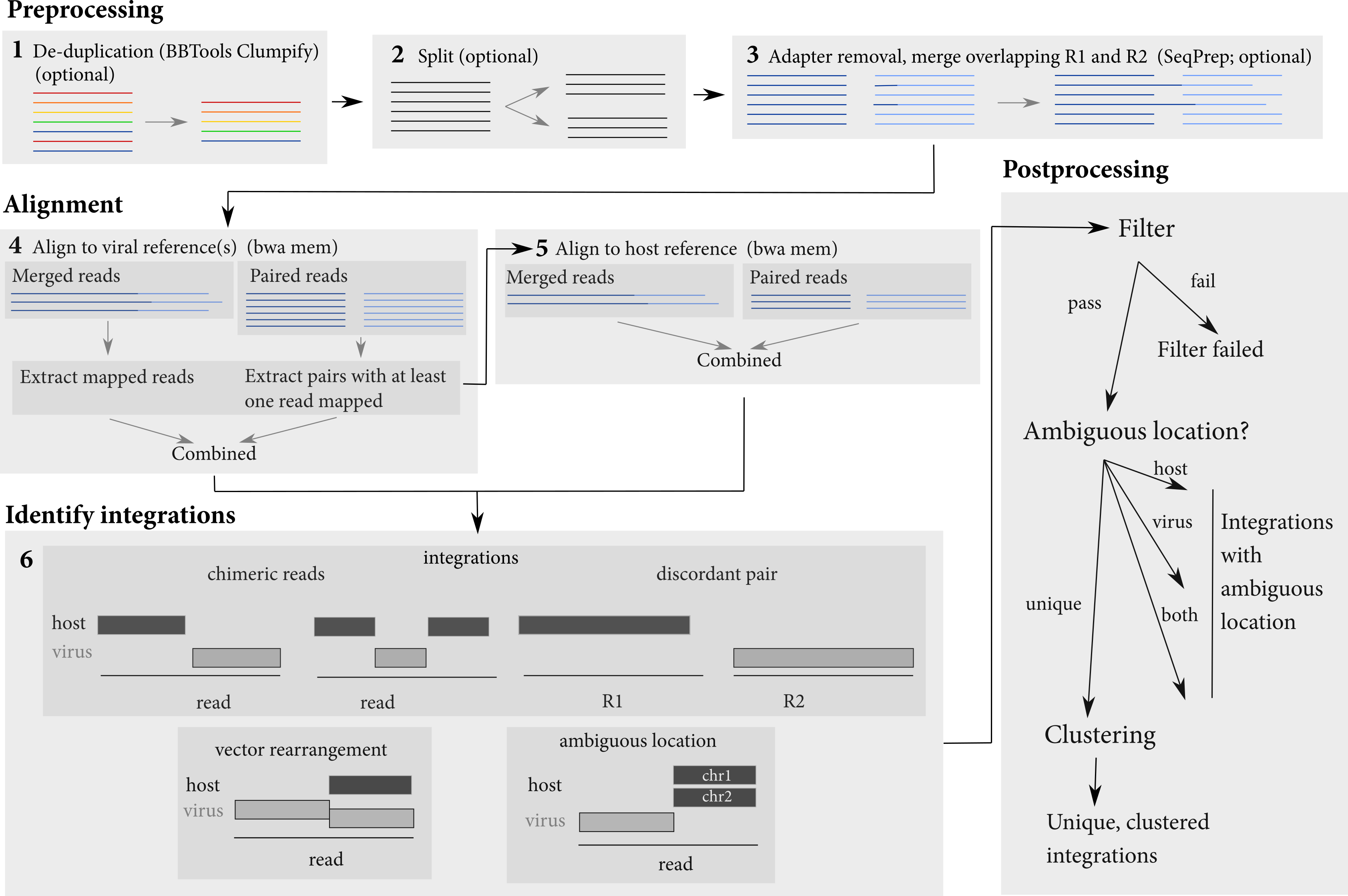
La tubería realiza varios pasos para identificar sitios de integración. Se necesitan como conjuntos de datos de entrada que consisten en archivos FASTQ o archivos BAM. Realiza un preprocesamiento de las lecturas (fusionando lecturas superpuestas, opcionales) y luego las alinea tanto con un huésped como a una secuencia viral. Las lecturas se alinean primero con las secuencias virales, y luego las lecturas alineadas se extraen y se alinean con el huésped. Estas alineaciones se utilizan para identificar integraciones virales.
Isling requiere snakemake y singularity (recomendado) o conda para suministrar dependencias. Además, se requieren python versión 3.5 o superior y pandas (estos deben instalarse automáticamente si se instala snakemake con conda .
Alternativley, use la versión Docker que contiene isling y todas las dependencias.
Las entradas requeridas son el archivo de configuración, que especifica las referencias host y virales/vectoriales, y se requieren lecturas. Especifique todas las entradas en un archivo de configuración. Actualmente, Isling solo funciona para lecturas de extremo emparejado.
Consulte el archivo configfile.md para obtener una descripción del formato de este archivo de configuración.
Sitios de integración de salidas isling en un formato separado por pestañas en el directorio de salida especificado en el archivo de configuración.
Dentro de la carpeta de salida, se crea una carpeta para cada conjunto de datos en el archivo de configuración, y para cada conjunto de datos, las integraciones se pueden encontrar en el directorio ints . Habrá un conjunto de archivos de salida para cada muestra.
Para cada muestra, hay una serie de archivos de salida, que pueden ser de interés para casos de uso particulares.
<sample>.<host>.<virus>.integrations.txt : información sobre todas las uniones detectadas, independientemente de si pasaron los filtros o no<sample>.<host>.<virus>.integrations.post.txt : información sobre las uniones detectadas que pasaron todos los filtros<sample>.<host>.<virus>.integrations.post.unique.txt : información sobre las uniones detectadas que pasaron todos los filtros y tienen una ubicación inequívoca tanto en huésped como en vector/virus<sample>.<host>.<virus>.integrations.post.unique.merged.txt : el resultado de fusionar uniones o uniones superpuestas con las mismas coordenadas tanto en huésped como en virus/vector. La fusión se realiza solo con las uniones de integración que tienen una ubicación inequívoca tanto en huésped como en virus/vector.<sample>.<host>.<virus>.integrations.post.host_ambig.txt : información sobre las uniones detectadas que pasaron todos los filtros y tienen una ubicación inequívoca vector/virus pero ubicación ambigua en el anfitrión<sample>.<host>.<virus>.integrations.post.virus_ambig.txt : información sobre las uniones detectadas que pasaron todos los filtros y tienen un host de ubicación inequívoca pero ubicación ambigua en vector/virus<sample>.<host>.<virus>.integrations.post.both_ambig.txt : información sobre las uniones detectadas que pasaron todos los filtros y tienen una ubicación ambigua tanto en host como en vector/virus Los archivos de salida dan la ubicación de las integraciones identificadas y sus propiedades. Las coordenadas para las uniones de integración se especifican en términos de sus bases ambiguas. Es decir, a menudo hay una brecha o superposición entre el huésped y las porciones virales de una lectura: 
Dado que la ubicación de la integración no se puede determinar de manera única en este caso, Isling genera las coordenadas de estas bases en el genoma del huésped y el vector/virus como la ubicación de la integración.
Con la excepción del archivo de salida del clúster fusionado, todos los archivos contienen estas columnas:
gap o overlap para una lectura quimérica (partes virales y huéspedes identificadas en la misma lectura), una unión está clean si no tiene bases ambiguas. Alternativamente, si la jucntion se identificó en un par discordante, el OverlapType es discordanthv ) o virus/huésped ( vh )+ o -bwa ) de la alineación del hostbwa ) de la alineación viralOverlapType es gapchimeric , un par discordant o es una integración short en la que se observaron ambas uniones en la misma lecturabwa )bwa )'xxx' Para reproducir las figuras en el manuscrito isling, vea el Readme en el directorio benchmarking .