Investigadores del Instituto Tecnológico de Massachusetts (MIT) han lanzado un potente modelo de inteligencia artificial de código abierto llamado Boltz-1 que logra la misma precisión que AlphaFold3 de Google DeepMind en la predicción de estructuras de biomoléculas. Se espera que este avance acelere significativamente el proceso de investigación biomédica y desarrollo de fármacos, proporcionando una herramienta más conveniente y eficiente para los investigadores científicos globales. La naturaleza totalmente de código abierto de Boltz-1 también fomenta una colaboración y participación comunitaria más amplias, lo que hace avanzar aún más el ritmo de los descubrimientos científicos.
Los científicos del Instituto Tecnológico de Massachusetts (MIT) lanzaron recientemente un potente modelo de inteligencia artificial de código abierto llamado Boltz-1. Esta innovación promete acelerar significativamente la investigación biomédica y el desarrollo de fármacos.
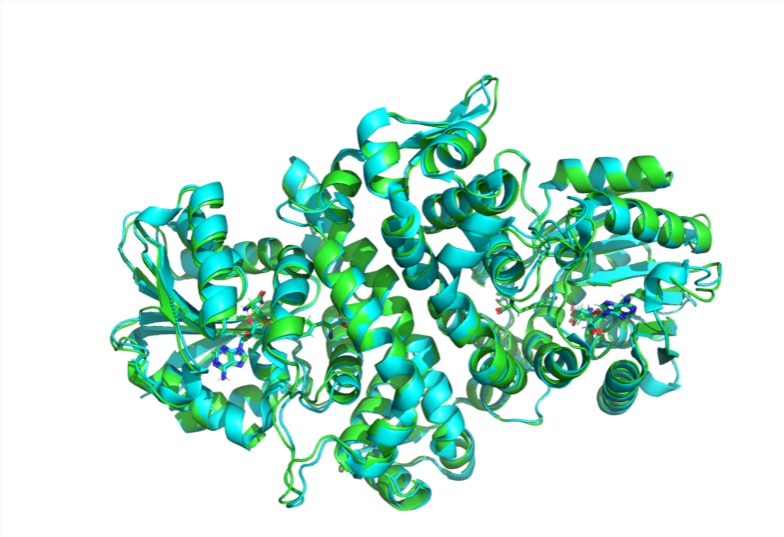
Boltz-1 es el primer modelo de código abierto capaz de alcanzar el mismo nivel avanzado que AlphaFold3 de Google DeepMind en predicción de estructuras de biomoléculas. El modelo fue desarrollado por un equipo de la Clínica de Salud de Aprendizaje Automático Jameel del MIT, dirigido por los estudiantes graduados Jeremy Wolvind y Gabriel Corso, y que también incluye al investigador del MIT Salo Passaro y al ingeniero eléctrico con los profesores de informática Regina Bardsley y Tommy Akara.
En el lanzamiento del 5 de diciembre, Wohlwind y Corso dijeron que su objetivo final es fomentar la colaboración global, acelerar el descubrimiento científico y proporcionar una plataforma sólida para avanzar en el modelado biomolecular. "Esperamos que esto pueda ser un punto de partida para la comunidad", dijo Corso, destacando que el nombre de "Boltz-1" en lugar de "Boltz" tiene como objetivo fomentar la participación de la comunidad.
Las proteínas desempeñan un papel clave en casi todos los procesos biológicos y la forma de una proteína está estrechamente relacionada con su función, por lo que comprender la estructura de una proteína es crucial para diseñar nuevos fármacos o diseñar nuevas proteínas con funciones específicas. Debido a que el proceso de plegar largas cadenas de aminoácidos proteicos en estructuras tridimensionales es extremadamente complejo, predecir con precisión sus estructuras siempre ha sido un gran desafío para la comunidad científica.
AlphaFold2 de DeepMind utiliza el aprendizaje automático para predecir rápidamente estructuras de proteínas 3D con una precisión tan alta que a los científicos experimentales les resulta difícil distinguirlas. AlphaFold3 mejoró sobre esta base y adoptó un modelo de IA generativa. Sin embargo, debido a que no era completamente de código abierto, fue criticado por la comunidad científica. Por lo tanto, el equipo de investigación del MIT se propuso desarrollar Boltz-1, siguiendo las ideas básicas de AlphaFold3 y realizando mejoras sobre esta base para mejorar la precisión y la eficiencia de predicción del modelo.
El equipo de investigación pasó cuatro meses y realizó múltiples experimentos para superar los problemas de ambigüedad y heterogeneidad encontrados en el banco de datos de proteínas. En última instancia, sus experimentos demostraron que Boltz-1 logró la misma precisión que AlphaFold3 en la predicción de la estructura de biomoléculas complejas.
Los investigadores planean seguir mejorando el rendimiento de Boltz-1 y acortando el tiempo de predicción. También invitan a los investigadores a probar Boltz-1 en GitHub y comunicarse con otros usuarios a través del canal Slack. El equipo de investigación espera que Boltz-1 promueva una colaboración más amplia e inspire aplicaciones creativas en la comunidad.
Proyecto: https://jclinic.mit.edu/democratizing-science-boltz-1/
Reflejos:
Boltz-1 es el primer modelo de predicción de estructuras de biomoléculas de código abierto que logra el mismo rendimiento que AlphaFold3.
El modelo fue desarrollado para promover la colaboración global y avanzar en la investigación biomédica y el desarrollo de fármacos.
El equipo del MIT espera utilizar Boltz-1 para simplificar la predicción de la estructura de las proteínas para que más investigadores puedan utilizar esta poderosa herramienta.
La naturaleza de código abierto de Boltz-1 reduce el umbral para la investigación biomédica, proporciona a los científicos de todo el mundo herramientas poderosas y acelera el proceso de desarrollo de fármacos. Esta contribución del equipo del MIT tendrá sin duda un profundo impacto en la investigación biomédica futura. Esperamos que Boltz-1 logre más avances en aplicaciones futuras.