Dernière mise à jour : 30/09/2024
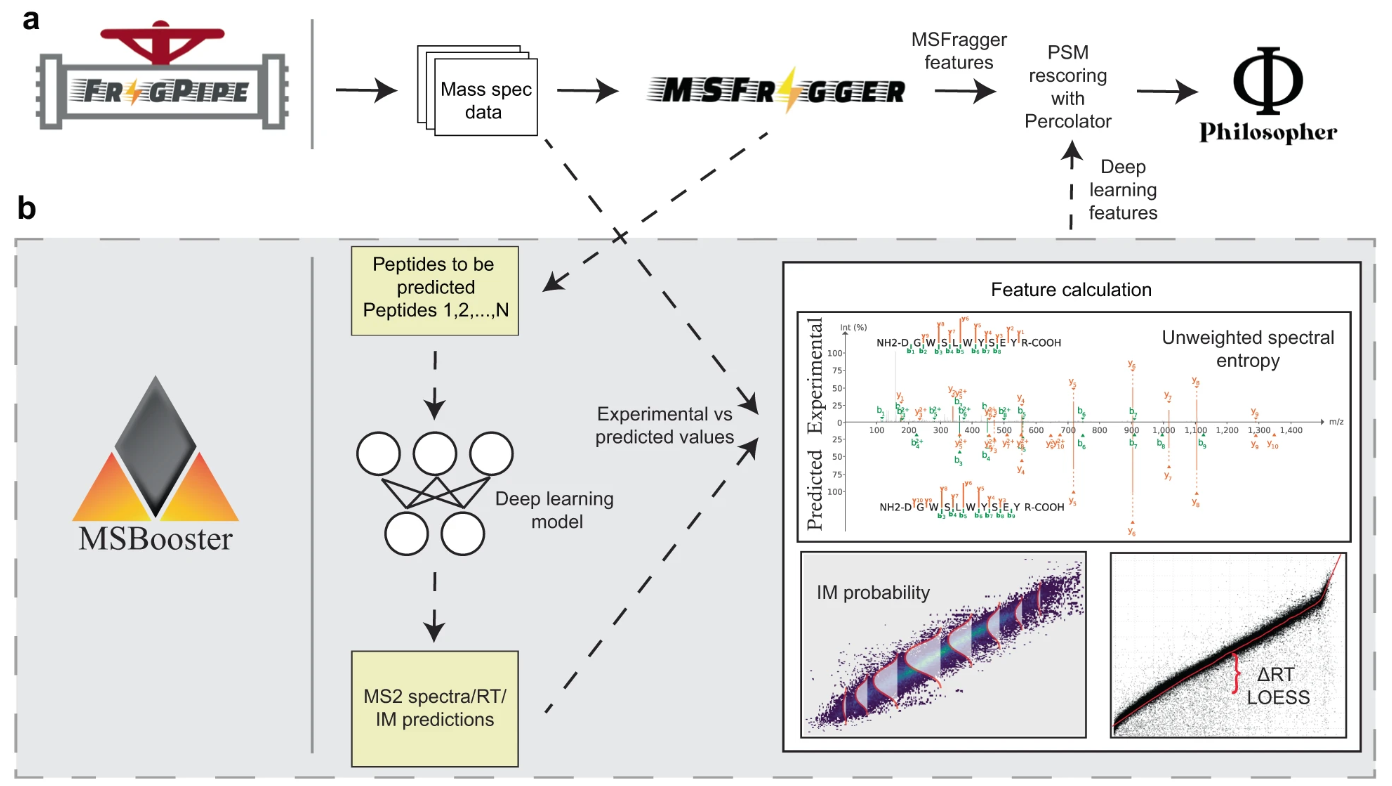
MSBooster est un outil permettant d'incorporer des prédictions de bibliothèques spectrales dans la réévaluation de la correspondance peptide-spectre (PSM) dans les données protéomiques de spectrométrie de masse par chromatographie liquide tandem ascendante. Elle se décompose grosso modo en 4 étapes :
Extraction de peptides à partir de PSM dans les résultats de recherche et formatage pour les fichiers d'entrée des prédicteurs d'apprentissage automatique/deep learning (ML/DL)
Appel du ou des modèles de prédiction et enregistrement de la sortie
Calcul des fonctionnalités
Ajout de nouvelles fonctionnalités au fichier des résultats de recherche
MSBooster est compatible avec de nombreux types de recherches dans des bases de données, notamment l'immunopeptidomique HLA, DDA et DIA, ainsi que la protéomique unicellulaire. Il est intégré à FragPipe et est inclus dans plusieurs de ses flux de travail. MSBooster a été développé en pensant à d'autres outils FragPipe, tels que FragPipe-PDV.
MSBooster est équipé pour gérer plusieurs formats et modèles de fichiers d'entrée :
| Sortie du spectromètre de masse |
|---|
| .mzML |
| .mgf |
| Fichier PSM |
|---|
| .épingle |
| .pepXML (en cours) |
| Modèle de prédiction |
|---|
| DIA-NN |
| Modèles Koina |
MSBooster peut être exécuté sur les systèmes Windows et Linux. Si vous utilisez FragPipe, aucune autre étape d'installation n'est nécessaire en dehors de l'installation de FragPipe. MSBooster se trouve dans l'onglet "Validation". Choisissez d'activer les fonctionnalités de temps de rétention avec « Predict RT » et les fonctionnalités spectrales MS/MS avec « Predict spectra ». Veuillez vous référer à la documentation FragPipe pour savoir comment exécuter une analyse.
Si vous utilisez MSBooster autonome pour l'exécuter dans la ligne de commande, veuillez télécharger le dernier fichier jar à partir des versions. MSBooster nécessite également DIA-NN pour la prédiction MS/MS et RT. Veuillez installer DIA-NN et noter le chemin d'accès à l'exécutable DIA-NN (ex. DiaNN.exe pour Windows, diann-1.8.1.8 pour Linux).
Vous pouvez exécuter MSBooster à l'aide d'une commande similaire à la suivante :
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
Les paramètres minimum à transmettre sont :
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
Bien que vous puissiez transmettre ces paramètres individuellement, il est plus facile d’en placer un sur chaque ligne du fichier paramsList. Veuillez vous référer à msbooster_params.txt pour un modèle.
Les paramètres ci-dessous sont destinés à un usage général. Les paramètres spécifiques à Koina se trouvent dans la documentation de Koina
paramsList (String) : emplacement dans le fichier texte contenant les paramètres pour cette exécution
fragger (String) : chemin du fichier fragger.params de l'exécution MSFragger. MSBooster lira plusieurs paramètres et ajustera les paramètres internes en fonction de ceux-ci, tels que la tolérance d'erreur de masse des fragments et les décalages de masse.
outputDirectory (String) : où sortir les nouveaux fichiers
editedPin (String) : MSBooster nommera le nouveau fichier en fonction de ceux fournis. Par exemple, A.pin aura un homologue appelé A_edited.pin. Pour modifier la valeur par défaut "modifié", fournissez une nouvelle chaîne ici
renamePin (int) : s'il faut générer un nouveau fichier pin ou réécrire l'ancien. La valeur par défaut ici est 1, qui ne sera pas écrasée. Le définir sur 0 écrasera l'ancien fichier PIN
deletePreds (boolean) : s'il faut supprimer les fichiers stockant les prédictions du modèle après avoir terminé une exécution réussie. Par défaut, défini sur false. Définissez sur true si vous souhaitez les supprimer
loadingPercent (int) : à quelle fréquence signaler la progression des tâches à l'aide d'un rapporteur de progression. Par défaut, défini sur 10, ce qui signifie qu'une mise à jour sera imprimée tous les 10 %.
numThreads (int) : nombre de threads à utiliser. Par défaut, défini sur 0, qui utilise tous les threads disponibles moins 1
splitPredInputFile (int) : utilisé uniquement lorsque les prédictions DIA-NN échouent en raison d'une erreur de mémoire insuffisante (137). Par défaut, défini sur 1, mais vous pouvez l'augmenter pour spécifier en combien de fichiers plus petits le fichier d'entrée DIA-NN doit être divisé. Chaque fichier sera alors prédit séquentiellement, allégeant la charge de mémoire
plotExtension (String) : quel format de fichier les tracés doivent être. png par défaut, et pdf est également autorisé
features (String) : liste des fonctionnalités à calculer. Sensible à la casse, séparé par des communications sans espaces entre les deux. La valeur par défaut est "predRTrealUnits, unweightedSpectralEntropy, deltaRTLOESS"
spectraPredFile (String) : si vous réutilisez d'anciennes prédictions spectrales (par exemple de DIA-NN ou Koina), vous pouvez spécifier l'emplacement du fichier ici
RTPredFile (String) : identique à spectraPredFile, mais pour les prédictions RT
IMPredFile (String) : identique à spectraPredFile, mais pour les prédictions IM
spectraModel (String) : quel modèle de prédiction spectrale utiliser
rtModel (String) : identique à spectraModel, mais pour RT
imModel (String) : identique à spectraModel, mais pour IM
useSpectra (boolean) : s'il faut utiliser des fonctionnalités basées sur la prédiction spectrale. Définir sur vrai par défaut
useRT (boolean) : s'il faut utiliser les fonctionnalités basées sur la prédiction RT. Définir sur vrai par défaut
useIM (boolean) : s'il faut utiliser les fonctionnalités basées sur la prédiction de messagerie instantanée. Défini sur false par défaut
ppmTolerance (float) : tolérance ppm d'erreur de fragment (20 ppm par défaut)
matchWithDaltons (boolean) : s'il faut faire correspondre les fragments prédits et observés dans les Daltons (faux par défaut)
DaTolerance (float) : combien de daltons autour du pic prédit pour rechercher le pic expérimental (par défaut 0,05)
useTopFragments (boolean) : s'il faut filtrer la prédiction spectrale sur les N pics d'intensité les plus élevés (true par défaut)
topFragments (int) : jusqu'à combien de fragments prédits doivent être utilisés pour le calcul des caractéristiques (20 par défaut). Appliqué uniquement si useTopFragments est vrai
removeRankPeaks (boolean) : défini sur true par défaut, qui filtre les fragments des spectres expérimentaux une fois mis en correspondance. Si faux, les fragments expérimentaux peuvent être mis en correspondance par plusieurs PSM issus de la même analyse
useBasePeak (boolean) : indique si une limite inférieure doit être appliquée aux prédictions MS2 pour utiliser uniquement des fragments d'intensité plus élevée (true par défaut)
percentBasePeak (float) : pourcentage auquel le fragment avec une intensité de quelques pour cent de l'intensité maximale de base est inclus dans le calcul de similarité. Appliqué uniquement si useBasePeak est vrai (par défaut 1)
loessEscoreCutoff (float) : seuil de valeur attendue utilisé pour le premier passage lors de la collecte des PSM pour l'étalonnage RT/IM. La valeur par défaut est 10^-3,5, soit environ 0,000316
rtLoessRegressionSize (int) : nombre maximum de PSM utilisés pour l'étalonnage RT LOESS (par défaut 5000)
imLoessRegressionSize (int) : identique à rtLoessRegressionSize mais pour IM (par défaut 1000)
minLoessRegressionSize (int) : nombre minimum de PSM nécessaires pour tenter l'étalonnage LOESS RT/IM (par défaut 100). Si moins de ce nombre de PSM sont disponibles, la régression linéaire est utilisée à la place
minLinearRegressionSize (int) : nombre minimum de PSM nécessaires pour tenter l'étalonnage RT/IM de régression linéaire (par défaut 10). Si moins de ce nombre de PSM sont disponibles, aucun étalonnage n'est tenté
loessBandwidth (String) : liste des bandes passantes à essayer pour l'étalonnage RT/IM LOESS (par défaut 0.01,0.05,0.1,0.2). Celui-ci doit être séparé par des virgules, sans espaces entre les deux.
regressionSplits (int) : nombre de validations croisées utilisées pour l'étalonnage RT/IM LOESS (par défaut 5)
massesForLoessCalibration (String) : masses pour les déplacements de masse qui doivent être adaptées à leurs propres courbes d'étalonnage. La liste est séparée par des virgules sans espaces entre les deux. Les masses doivent être écrites avec le même nombre de chiffres que dans le fichier PIN
loessScatterOpacity (float) : opacité des nuages de points dans les figures de calibration LOESS, de 0 à 1 (par défaut 0.35)
Fichier .pin avec de nouvelles fonctionnalités. Par défaut, de nouveaux fichiers de broches seront produits et se termineront par "_edited.pin". Les fonctionnalités par défaut utilisées sont "unweighted_spectral_entropy", "delta_RT_loess" et "pred_RT_real_units". Si les fonctionnalités de mobilité ionique sont activées, "delta_IM_loess" et "ion_mobility" seront également inclus
spectraRT.tsv et spectraRT_full.tsv : fichiers d'entrée pour le modèle de prédiction DIA-NN
spectraRT.predicted.bin : un fichier binaire avec les prédictions de DIA-NN à utiliser par MSBooster pour le calcul des caractéristiques. Si vous utilisez FragPipe-PDV, ces fichiers sont utilisés pour générer des tracés miroir de spectres expérimentaux et prédits.
MSBooster produit plusieurs graphiques qui peuvent être utilisés pour examiner plus en détail la façon dont vos données se comparent aux prédictions du modèle.
Dossier MSBooster_plots :
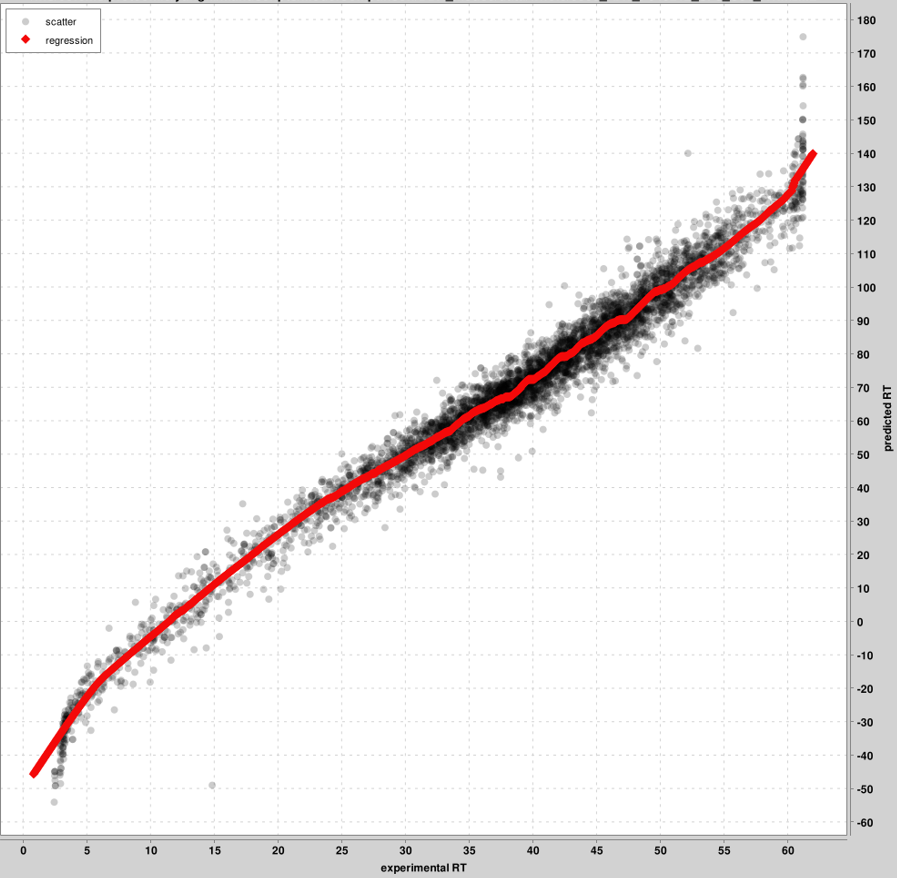
RT_calibration_curves : jusqu'aux 5 000 premiers PSM seront utilisés pour l'étalonnage entre les échelles RT expérimentales et prédites. Ces principaux MSP sont présentés dans le graphique, et non tous les MSP. Un graphique sera produit par fichier pin
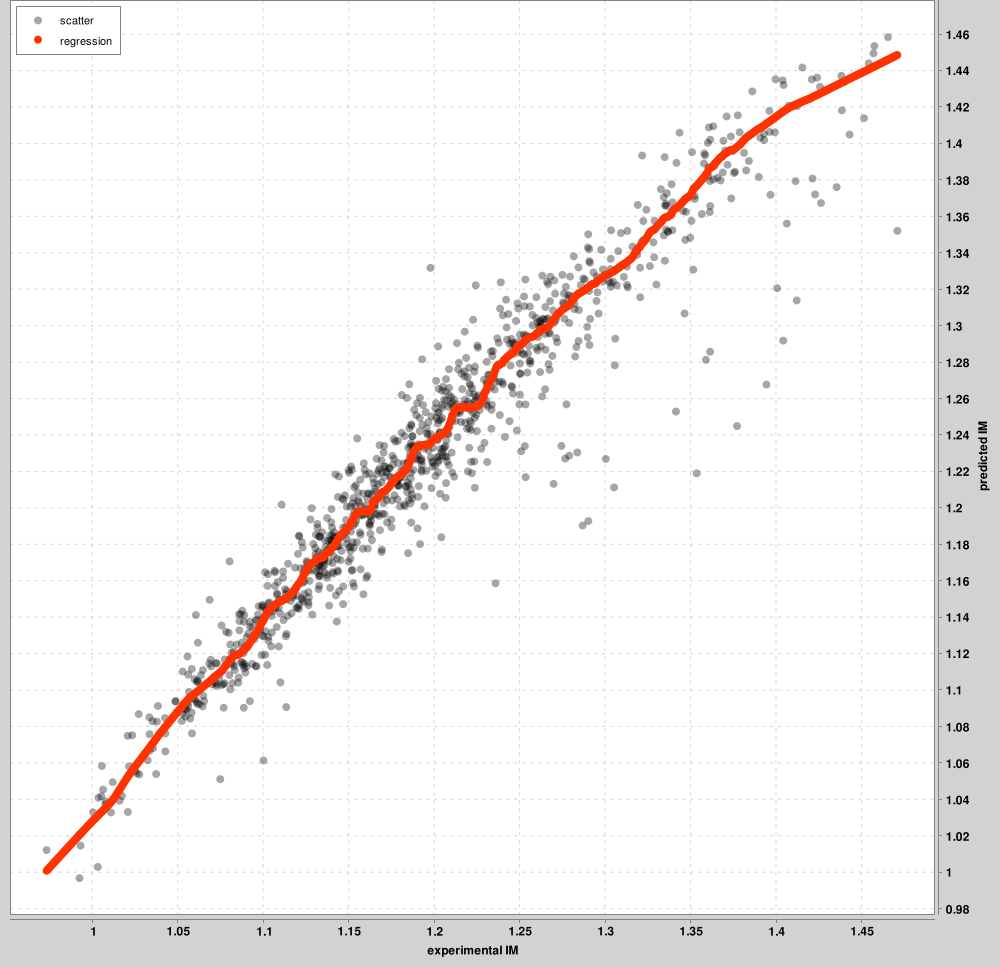
IM_calibration_curves : jusqu'aux 1 000 premiers PSM seront utilisés pour l'étalonnage entre les échelles IM expérimentales et prédites. Ces principaux MSP sont présentés dans le graphique, et non tous les MSP. Une courbe distincte sera apprise pour chaque état de charge. La figure ci-dessous est un exemple pour les précurseurs de charge 2
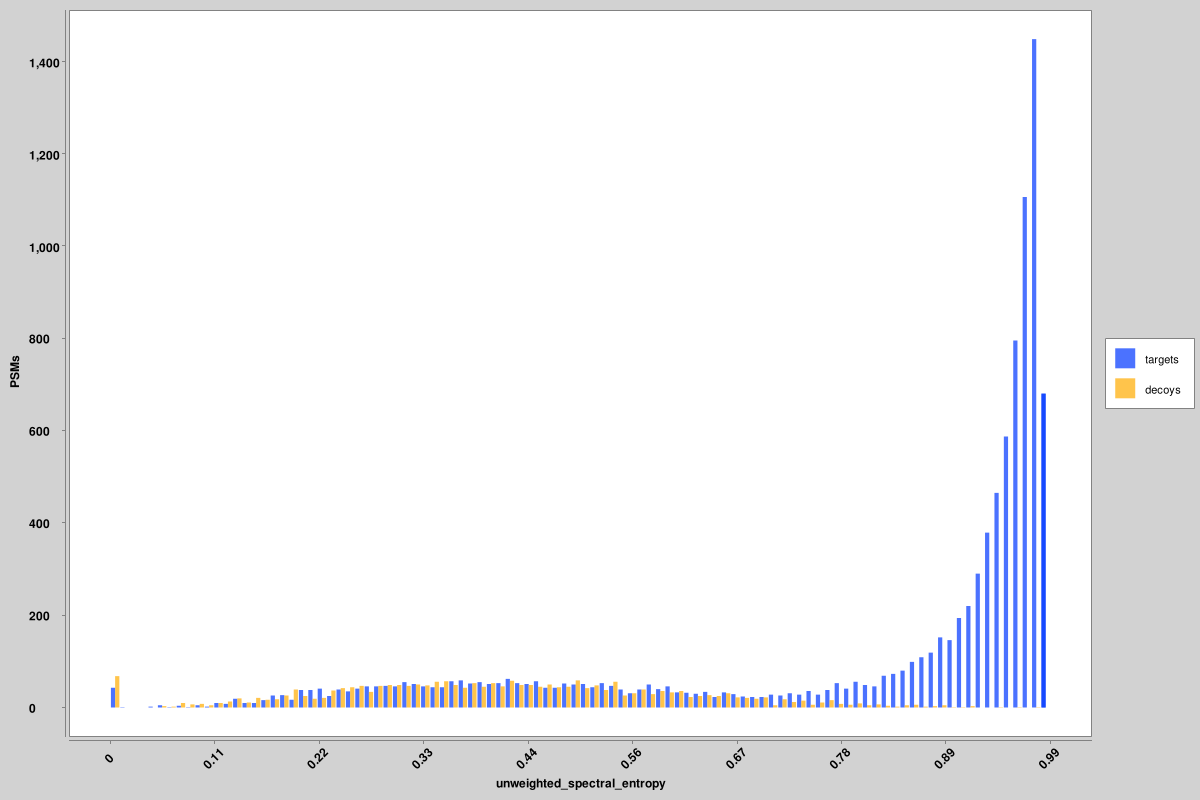
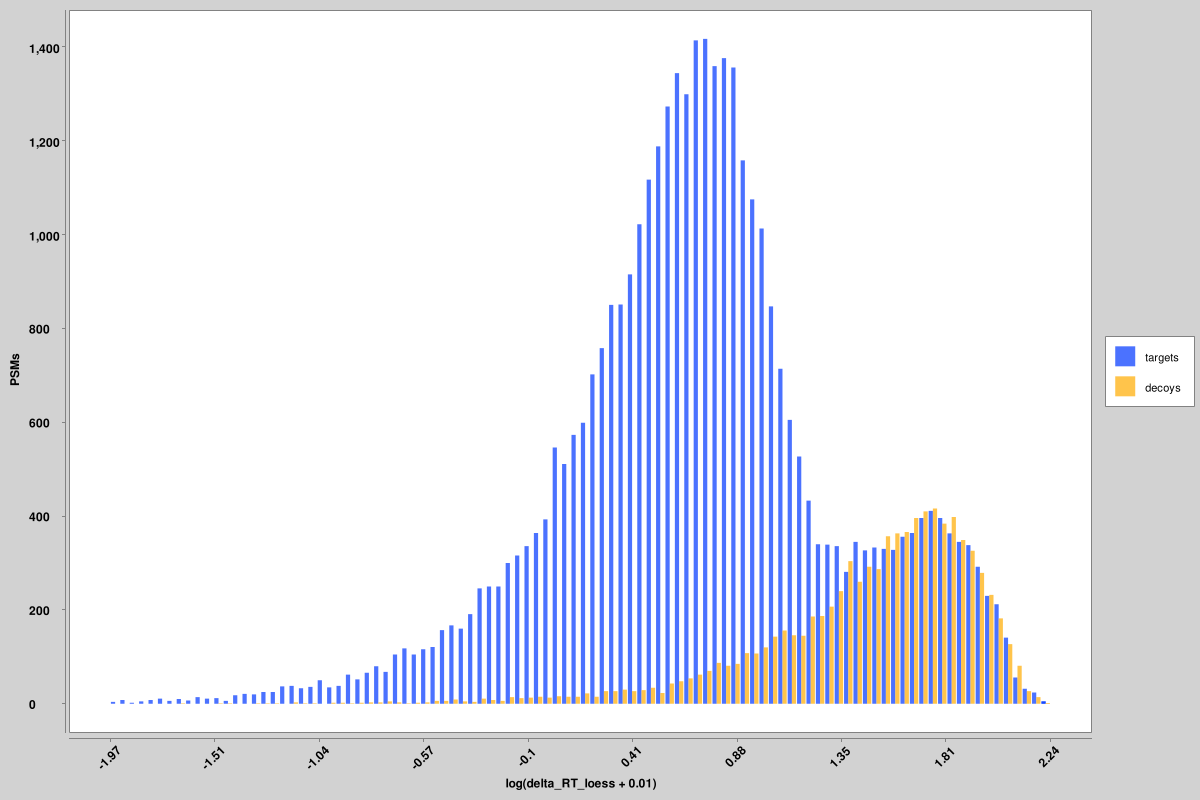
score_histograms : histogrammes superposés de tous les PSM cibles et leurres pour chaque fichier de broches. Certaines caractéristiques sont tracées ici sur une échelle logarithmique pour une meilleure visualisation de la distribution bimodale des vrais et des faux positifs, mais la valeur d'origine est celle utilisée dans les fichiers PIN, et non la version à l'échelle logarithmique. Voici des histogrammes pour l'entropie spectrale non pondérée et les scores delta RT, mais des histogrammes similaires sont produits pour toutes les fonctionnalités.
Utilisez les modèles de prédiction peptidique de Koina pour la génération de fonctionnalités MSBooster : https://fragpipe.nesvilab.org/docs/tutorial_koina.html
Lecture des prédictions de n'importe quel modèle via des fichiers MGF
Documentation sur toutes les fonctionnalités autorisées et comment les contrôler avec une sortie graphique
Veuillez citer ce qui suit lorsque vous utilisez MSBooster : https://www.nature.com/articles/s41467-023-40129-9