Ceci est le référentiel officiel de « Un modèle pour les gouverner tous : vers une segmentation universelle des images médicales avec des invites de texte »
Il s'agit d'un modèle de segmentation universel amélioré par les connaissances, construit sur une collecte de données sans précédent (72 ensembles de données de segmentation médicale 3D publiques), capable de segmenter 497 classes de 3 modalités différentes (IRM, CT, TEP) et 8 régions du corps humain, à l'aide d'un texte (analyse anatomique). terminologie).
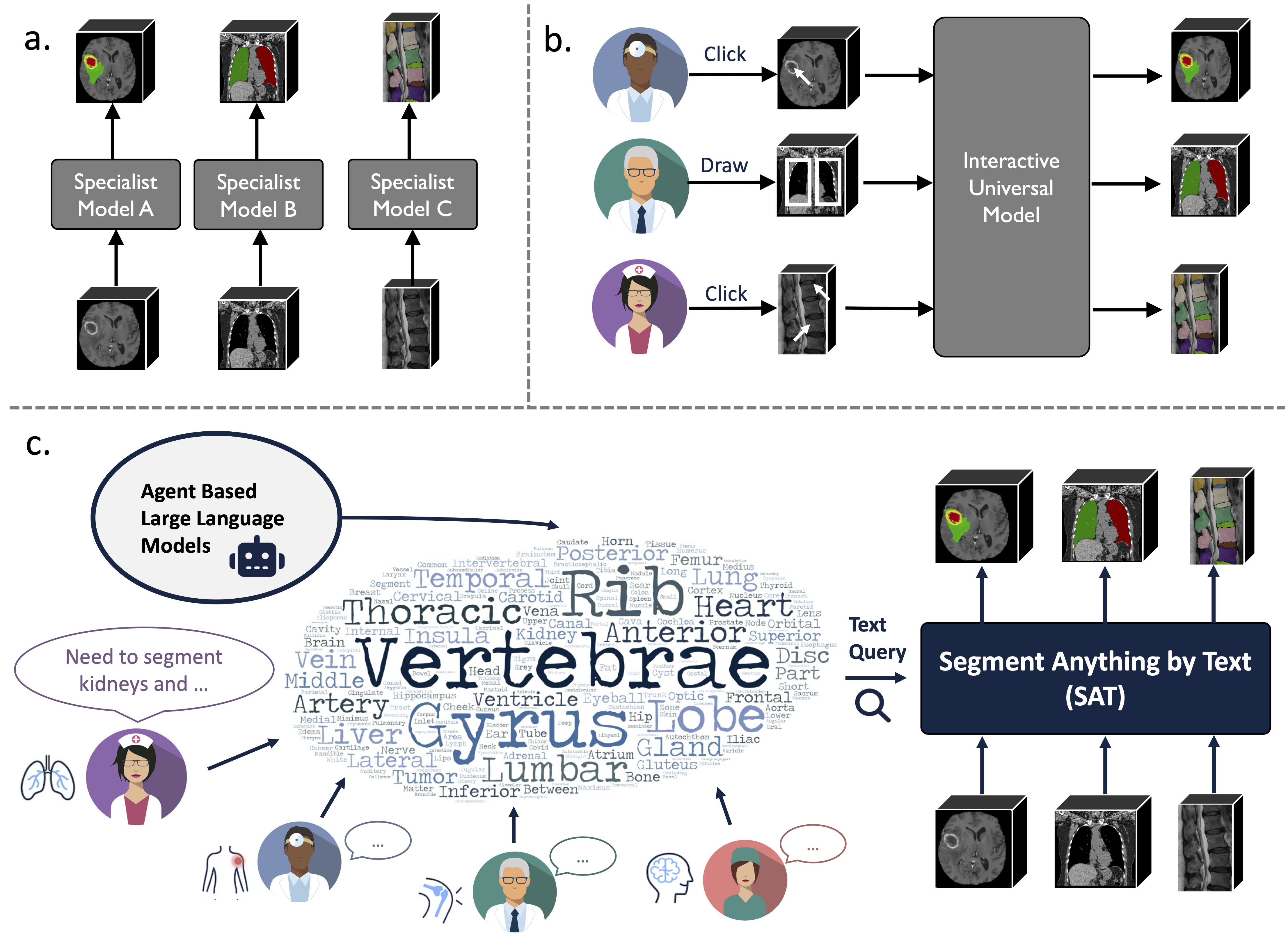
Cela peut être puissant et plus efficace que la formation et le déploiement d’une série de modèles spécialisés. Apprenez-en davantage sur notre site Web ou sur papier.
2024.08 ? Sur la base de SAT et de grands modèles linguistiques, nous construisons un ensemble de données d'interprétation de tomodensitométrie thoracique 3D complète, à grande échelle et guidée par région. Il contient une segmentation au niveau de l'organe pour 196 catégories et des rapports multi-granularités, où chaque phrase est basée sur la segmentation correspondante. Vérifiez-le sur Huggingface.
2024.06 ? Nous avons publié le code pour créer SAT-DS , une collection de 72 ensembles de données de segmentation publics, contenant plus de 22 000 images 3D, 302 000 masques de segmentation et 497 classes de 3 modalités différentes (IRM, CT, TEP) et 8 régions du corps humain, sur lesquelles nous construisons SAT. Nous proposons également des liens de téléchargement raccourcis pour les ensembles de données 42/72, qui sont prétraités et emballés par nos soins pour votre commodité, prêts à être utilisés immédiatement après le téléchargement et l'extraction. Consultez ce dépôt pour plus de détails.
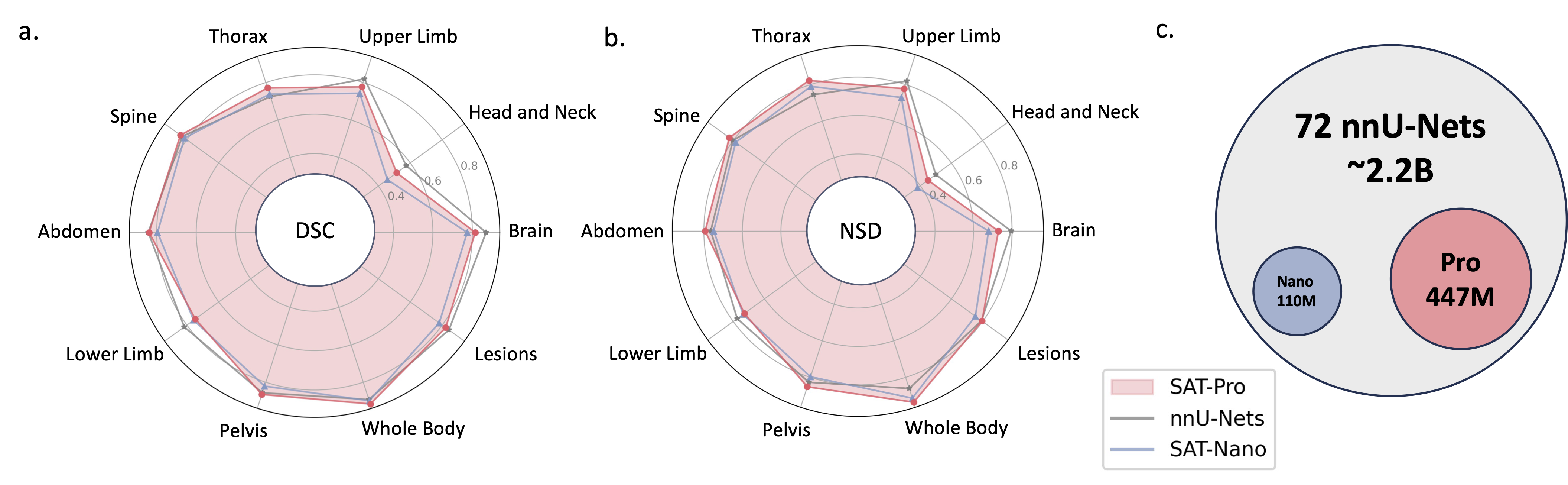
2024.05 ? Nous formons une nouvelle version de SAT avec une taille de modèle plus grande ( SAT-Pro ) et plus d'ensembles de données ( 72 ), et elle prend désormais en charge 497 classes ! Nous renouvelons également SAT-Nano et publions quelques variantes de SAT-Nano, basées sur différents piliers visuels (U-Mamba et SwinUNETR) et encodeurs de texte (MedCPT et BERT-Base). Pour plus de détails sur cette mise à jour, reportez-vous à notre nouveau document.
L'implémentation d'U-Net s'appuie sur une version personnalisée d'architectures de réseau dynamique, pour l'installer :
cd model
pip install -e dynamic-network-architectures-main
Quelques autres exigences clés :
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
Vous devez également installer mamba_ssm si vous souhaitez la variante U-Mamba de SAT-Nano
S1. Créez l'environnement en suivant requirements.txt .
S2. Téléchargez le point de contrôle de SAT et Text Encoder depuis Huggingface.
S3. Préparez les données dans un fichier jsonl. Consultez la démo dans data/inference_demo/demo.jsonl .
image (chemin d'accès à l'image), labe (nom des cibles de segmentation), dataset (à quel ensemble de données appartient l'échantillon) et modality (ct, IRM ou animal de compagnie) sont nécessaires pour chaque échantillon à segmenter. Les modalités et les classes prises en charge par SAT peuvent être trouvées dans le tableau 12 du document.
orientation_code (orientation) est RAS par défaut, ce qui convient à la plupart des images dans le plan axial. Pour les images dans le plan sagittal (par exemple, examen de la colonne vertébrale), réglez ceci sur ASR . L'image d'entrée doit avoir la forme H,W,D Notre code de traitement de données normalisera l'image d'entrée en termes d'orientation, d'intensité, d'espacement, etc. Deux images traitées avec succès peuvent être trouvées dans demoprocessed_data , assurez-vous que la normalisation est effectuée correctement pour garantir les performances de SAT.
S4. Démarrer l'inférence avec SAT-Pro ? :
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d est la taille du lot des correctifs d'image d'entrée et doit être ajusté en fonction de la mémoire GPU (consultez le tableau ci-dessous) ; Il est recommandé de définir --max_queries une valeur supérieure aux classes de l'ensemble de données d'inférence, sauf si la mémoire de votre GPU est très limitée ;
| Modèle | taille du lot_3d | Mémoire GPU |
|---|---|---|
| SAT-Pro | 1 | ~ 34 Go |
| SAT-Pro | 2 | ~ 62 Go |
| SAT-Nano | 1 | ~ 24 Go |
| SAT-Nano | 2 | ~ 36 Go |
S5. Vérifiez --rcd_dir pour les sorties. Les résultats sont organisés par ensembles de données. Pour chaque cas, l'image d'entrée, le résultat de segmentation agrégé et un dossier contenant les segmentations de chaque classe seront trouvés. Toutes les sorties sont stockées sous forme de fichiers nifiti. Vous pouvez les visualiser à l'aide de l'ITK-SNAP.
Si vous souhaitez utiliser SAT-Nano formé sur 72 ensembles de données, modifiez simplement --vision_backbone en 'UNET' et modifiez le --checkpoint et --text_encoder_checkpoint en conséquence.
Pour les autres variantes de SAT-Nano (entraînées sur 49 ensembles de données) :
UNET-Ours : définissez --vision_backbone 'UNET' et --text_encoder 'ours' ;
UNET-CPT : définissez --vision_backbone 'UNET' et --text_encoder 'medcpt' ;
UNET-BB : définissez --vision_backbone 'UNET' et --text_encoder 'basebert' ;
UMamba-CPT : définissez --vision_backbone 'UMamba' et --text_encoder 'medcpt' ;
SwinUNETR-CPT : définissez --vision_backbone 'SwinUNETR' et --text_encoder 'medcpt' ;
Un peu de préparation avant de commencer la formation :
sh/ pour démarrer le processus de formation. Prenez SAT-Pro par exemple : sbatch sh/train_sat_pro.sh
Cela nécessite également de construire des données de test suite à ce dépôt. Vous pouvez vous référer au script slurm sh/evaluate_sat_pro.sh pour démarrer le processus d'évaluation :
sbatch sh/evaluate_sat_pro.sh
Si vous utilisez ce code pour votre recherche ou projet, veuillez citer :
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}