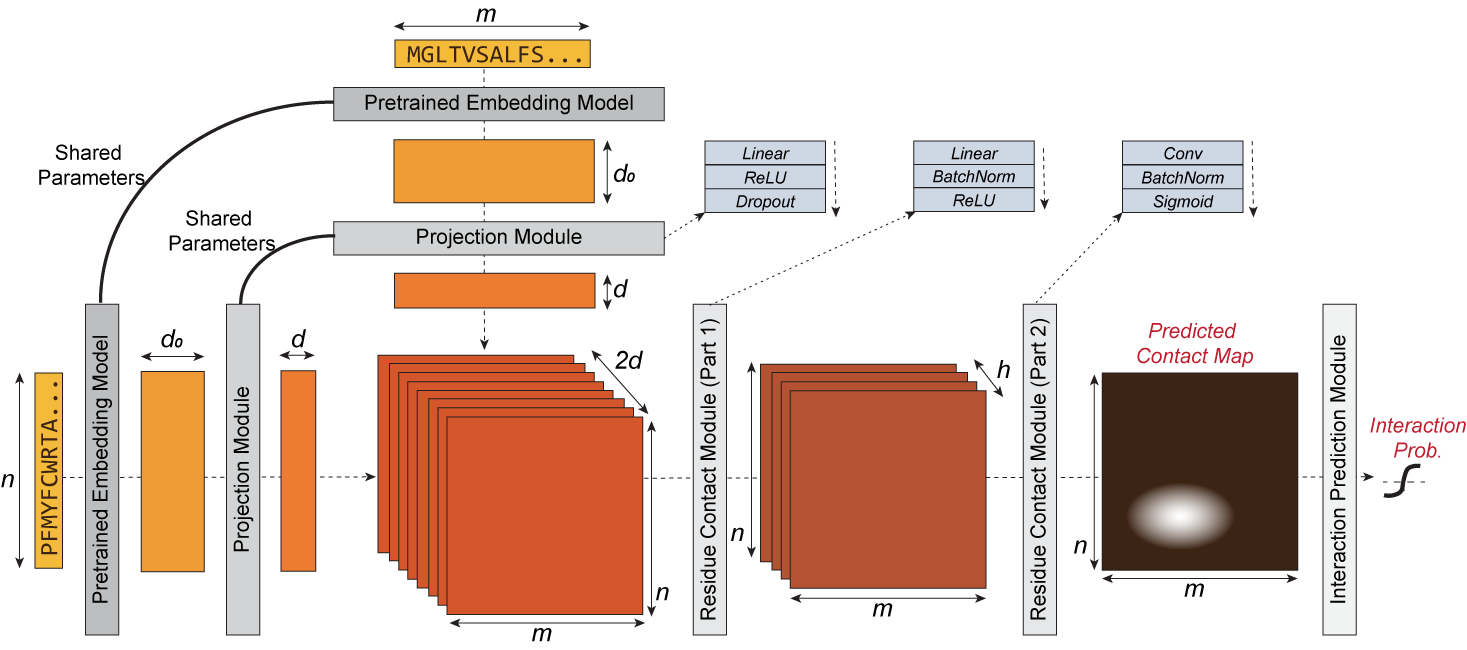
D-SCRIPT est une méthode d'apprentissage profond permettant de prédire une interaction physique entre deux protéines en fonction de leurs séquences uniquement. Il se généralise bien aux nouvelles espèces et résiste aux limitations de la taille des données d'entraînement. Sa conception reflète l'intuition selon laquelle pour que deux protéines interagissent physiquement, un sous-ensemble d'acides aminés de chaque protéine doit être en contact avec l'autre. Les étapes intermédiaires de D-SCRIPT mettent directement en œuvre cette intuition, l'avant-dernière étape de D-SCRIPT étant une estimation approximative de la carte de contact interprotéique du dimère protéique. Cette conception structurellement motivée améliore l'interprétabilité des résultats et, puisque la structure est plus conservée au cours de l'évolution que la séquence, améliore la généralisabilité entre les espèces.
D-SCRIPT est décrit dans l’article « Prédiction basée sur la séquence des interactions protéine-protéine : un modèle d’apprentissage profond interprétable sensible à la structure » par Sam Sledzieski, Rohit Singh, Lenore Cowen et Bonnie Berger.
Page d'accueil
Documentation