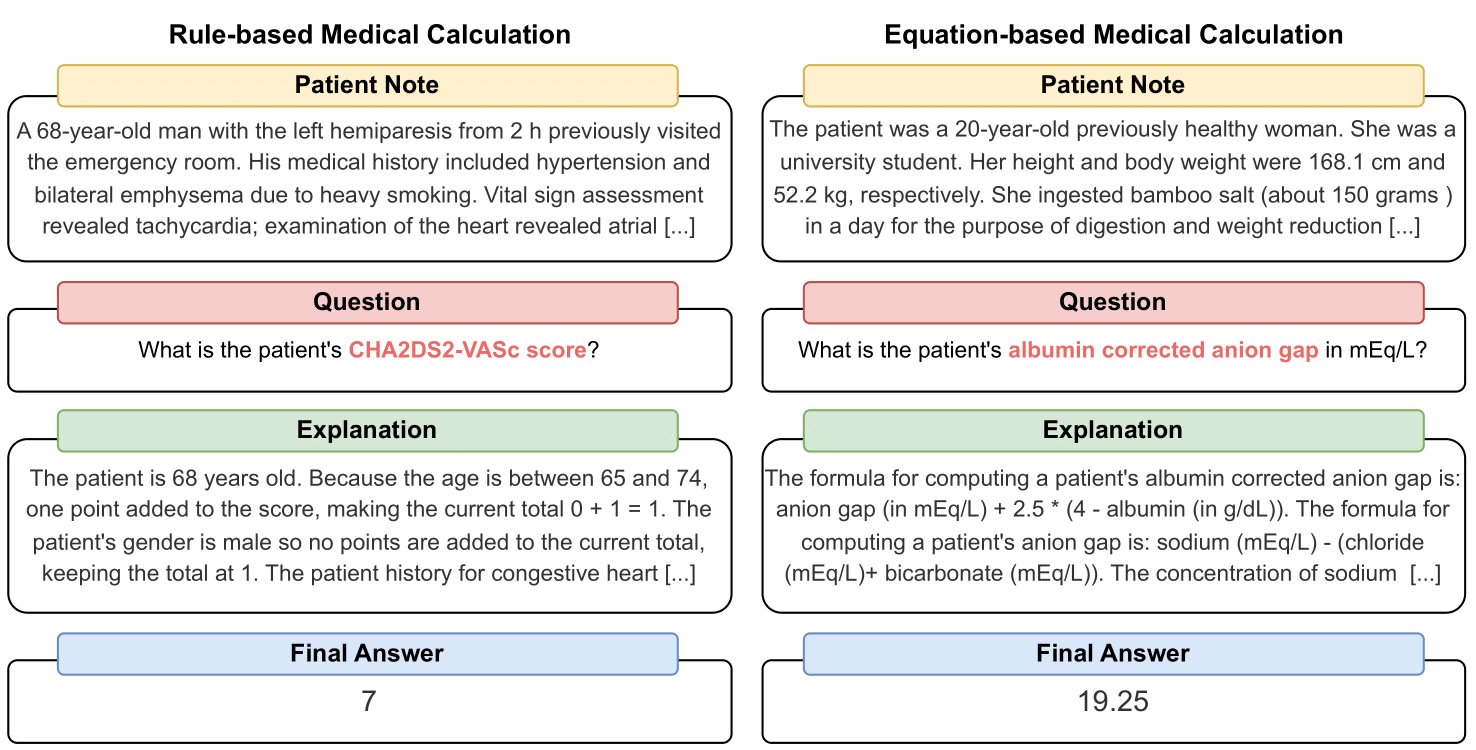
MedCalc-Bench est le premier ensemble de données de calcul médical utilisé pour évaluer la capacité des LLM à servir de calculateurs cliniques. Chaque instance de l'ensemble de données comprend une note du patient, une question demandant de calculer une valeur clinique spécifique, une valeur de réponse finale et une solution étape par étape expliquant comment la réponse finale a été obtenue. Notre ensemble de données couvre 55 tâches de calcul différentes qui sont soit des calculs basés sur des règles, soit des calculs basés sur des équations. Cet ensemble de données contient un ensemble de données de formation de 10 053 instances et un ensemble de données de test de 1 047 instances.
Dans l’ensemble, nous espérons que notre ensemble de données et notre référence serviront d’appel à améliorer les compétences de raisonnement informatique des LLM en milieu médical.
Notre prépublication est disponible sur : https://arxiv.org/abs/2406.12036.
Pour télécharger le CSV de l'ensemble de données d'évaluation MedCalc-Bench, veuillez télécharger le fichier test_data.csv dans le dossier de dataset de ce référentiel. Vous pouvez également télécharger l'ensemble de tests divisé à partir de HuggingFace à l'adresse https://huggingface.co/datasets/ncbi/MedCalc-Bench.
En plus des 1 047 instances d'évaluation, nous fournissons également un ensemble de données de formation de 10 053 instances qui peuvent être utilisées pour affiner les LLM open source (voir la section C de l'annexe). Les données d'entraînement se trouvent dans le fichier dataset/train_data.csv.zip et peuvent être décompressées pour obtenir train_data.csv . Cet ensemble de données de formation peut également être trouvé dans la division train du lien HuggingFace.
Chaque instance de l'ensemble de données contient les informations suivantes :
Pour installer tous les packages nécessaires à ce projet, veuillez exécuter la commande suivante : conda env create -f environment.yml . Cette commande créera l'environnement conda medcalc-bench . Pour exécuter des modèles OpenAI, vous devrez fournir votre clé OpenAI dans cet environnement conda. Vous pouvez le faire en exécutant la commande suivante dans l'environnement medcalc-bench : export OPENAI_API_KEY = YOUR_API_KEY , où YOUR_API_KEY est votre clé API OpenAI. Vous devrez également fournir votre jeton HuggingFace dans cet environnement en exécutant la commande suivante : export HUGGINGFACE_TOKEN=your_hugging_face_token , où your_hugging_face_token est votre jeton huggingface.
Pour reproduire le tableau 2 à partir du document, placez cd d'abord dans le dossier evaluation . Ensuite, veuillez exécuter la commande suivante : python run.py --model <model_name> and --prompt <prompt_style> .
Les options pour --model sont ci-dessous :
Les options pour --prompt sont ci-dessous :
À partir de là, vous obtiendrez un fichier jsonl affichant l'état de chaque question : lors de l'exécution run.py , les résultats seront enregistrés dans un fichier appelé <model>_<prompt>.jsonl . Ce fichier se trouve dans le dossier outputs .
Chaque instance du jsonl sera associée aux métadonnées suivantes :
{
"Row Number": Row index of the item,
"Calculator Name": Name of calculation task,
"Calculator ID": ID of the calculator,
"Category": type of calculation (risk, severity, diagnosis for rule-based calculators and lab, risk, physical, date, dosage for equation-based calculators),
"Note ID": ID of the note taken directly from MedCalc-Bench,
"Patient Note": Paragraph which is the patient note taken directly from MedCalc-Bench,
"Question": Question asking for a specific medical value to be computed,
"LLM Answer": Final Answer Value from LLM,
"LLM Explanation": Step-by-Step explanation by LLM,
"Ground Truth Answer": Ground truth answer value,
"Ground Truth Explanation": Step-by-step ground truth explanation,
"Result": "Correct" or "Incorrect"
}
De plus, nous fournissons la précision moyenne et le pourcentage d'écart type pour chaque sous-catégorie dans un json intitulé results_<model>_<prompt_style>.json . La précision cumulée et l'écart type parmi les 1 047 instances peuvent être trouvés sous la clé « globale » du JSON. Ce fichier se trouve dans le dossier results .
En plus des résultats du tableau 2 de l'article principal, nous avons également invité les LLM à écrire du code pour effectuer des opérations arithmétiques au lieu de laisser le LLM le faire lui-même. Les résultats se trouvent à l'annexe D. En raison de calculs limités, nous avons uniquement analysé les résultats pour GPT-3.5 et GPT-4. Pour examiner les invites et exécuter avec ce paramètre, veuillez examiner le fichier generate_code_prompt.py dans le dossier evaluation .
Pour exécuter ce code, cd -le simplement dans le dossier evaluations et exécutez ce qui suit : python generate_code_prompt.py --gpt <gpt_model> . Les options pour <gpt_model> sont soit 4 pour exécuter GPT-4, soit 35 pour exécuter GPT-3.5-turbo-16k. Les résultats seront ensuite enregistrés dans un fichier jsonl nommé : code_exec_{model_name}.jsonl dans le dossier outputs . Notez que dans ce cas, model_name sera gpt_4 si vous choisissez d'exécuter en utilisant GPT-4. Sinon, model_name sera gpt_35_16k si vous avez choisi d'exécuter avec GPT-3.5-turbo.
Les métadonnées de chaque instance dans le fichier jsonl pour les résultats de l'interpréteur de code sont les mêmes informations sur l'instance fournies dans la section ci-dessus. La seule différence est que nous stockons l'historique des discussions LLM entre l'utilisateur et l'assistant et que nous disposons d'une clé "Historique des discussions LLM" au lieu de la clé "Explication LLM". De plus, la sous-catégorie et la précision globale sont stockées dans un fichier JSON nommé results_<model_name>_code_augmented.json . Ce JSON se trouve dans le dossier results .
Cette recherche a été soutenue par le programme de recherche intra-muros des NIH, National Library of Medicine. De plus, les contributions apportées par Soren Dunn ont été réalisées en utilisant la ressource informatique et de données avancée Delta qui est soutenue par la National Science Foundation (prix OAC tel: 2005572) et l'État de l'Illinois. Delta est le fruit d'un effort conjoint de l'Université de l'Illinois à Urbana-Champaign (UIUC) et de son Centre national pour les applications du calcul intensif (NCSA).
Pour organiser les notes des patients dans MedCalc-Bench, nous utilisons uniquement des notes de patients accessibles au public provenant d'articles de rapports de cas publiés dans PubMed Central et de vignettes de patients anonymes générées par des cliniciens. À ce titre, aucune information personnelle identifiable sur la santé n’est révélée dans cette étude. Bien que MedCalc-Bench soit conçu pour évaluer les capacités de calcul médical des LLM, il convient de noter que l'ensemble de données n'est pas destiné à un usage diagnostique direct ou à une prise de décision médicale sans examen et surveillance par un professionnel clinique. Les individus ne devraient pas modifier leur comportement en matière de santé uniquement sur la base de notre étude.
Comme décrit à la section 1, les calculatrices médicales sont couramment utilisées en milieu clinique. Avec l'intérêt croissant pour l'utilisation des LLM pour des applications spécifiques à un domaine, les professionnels de la santé pourraient directement inciter des chatbots comme ChatGPT à effectuer des tâches de calcul médical. Cependant, les capacités des LLM dans ces tâches sont actuellement inconnues. Étant donné que les soins de santé sont un domaine à enjeux élevés et que des calculs médicaux erronés peuvent entraîner de graves conséquences, notamment des erreurs de diagnostic, des plans de traitement inappropriés et des dommages potentiels aux patients, il est crucial d'évaluer minutieusement les performances des LLM dans les calculs médicaux. Étonnamment, les résultats de l'évaluation de notre ensemble de données MedCalc-Bench montrent que tous les LLM étudiés ont des difficultés dans les tâches de calcul médical. Le modèle GPT-4 le plus performant n'atteint qu'une précision de 50 % avec un apprentissage ponctuel et une chaîne de réflexion. En tant que telle, notre étude indique que les LLM actuels ne sont pas encore prêts à être utilisés pour les calculs médicaux. Il convient de noter que même si des scores élevés sur MedCalc-Bench ne garantissent pas l'excellence dans les tâches de calcul médical, l'échec dans cet ensemble de données indique que les modèles ne doivent pas du tout être pris en compte à de telles fins. En d’autres termes, nous pensons que la réussite de MedCalc-Bench devrait être une condition nécessaire (mais pas suffisante) pour qu’un modèle puisse être utilisé à des fins de calcul médical.
Pour toute modification apportée à cet ensemble de données (c'est-à-dire l'ajout de nouvelles notes ou calculatrices), nous mettrons à jour les instructions README, test_set.csv et train_set.csv. Nous conserverons toujours les anciennes versions de ces ensembles de données dans un dossier archive/ . Nous mettrons également à jour les ensembles d’entraînement et de test pour HuggingFace.
Cet outil montre les résultats des recherches menées dans la Direction de la biologie computationnelle, NCBI/NLM. Les informations produites sur ce site Web ne sont pas destinées à un usage diagnostique direct ou à une prise de décision médicale sans examen et surveillance par un professionnel clinique. Les individus ne doivent pas modifier leur comportement en matière de santé uniquement sur la base des informations produites sur ce site Web. Le NIH ne vérifie pas de manière indépendante la validité ou l'utilité des informations produites par cet outil. Si vous avez des questions sur les informations produites sur ce site Web, veuillez consulter un professionnel de la santé. Plus d’informations sur la politique de non-responsabilité de NCBI sont disponibles.
En fonction du calculateur, notre ensemble de données se compose de notes conçues à partir de fonctions basées sur des modèles implémentées en Python, écrites à la main par des cliniciens ou extraites de notre ensemble de données, Open-Patients.
Open-Patients est un ensemble de données agrégées de 180 000 notes de patients provenant de trois sources différentes. Nous avons l'autorisation d'utiliser l'ensemble de données des trois sources. La première source est constituée des questions USMLE de MedQA, publiées sous licence MIT. La deuxième source de notre ensemble de données est l'aide à la décision clinique Trec et l'essai clinique Trec, qui peuvent être redistribués car il s'agit tous deux d'ensembles de données appartenant au gouvernement et rendus publics. Enfin, PMC-Patients est publié sous la licence CC-BY-SA 4.0 et nous avons donc la permission d'incorporer PMC-Patients dans Open-Patients et MedCalc-Bench, mais l'ensemble de données doit être publié sous la même licence. Par conséquent, notre source de notes, Open-Patients, et l'ensemble de données qui en est tiré, MedCalc-Bench, sont tous deux publiés sous la licence CC-BY-SA 4.0.
Sur la base de la justification des règles de licence, Open-Patients et MedCalc-Bench se conforment à la licence CC-BY-SA 4.0, mais les auteurs de cet article assumeront l'entière responsabilité en cas de violation des droits.
@misc { khandekar2024medcalcbench ,
title = { MedCalc-Bench: Evaluating Large Language Models for Medical Calculations } ,
author = { Nikhil Khandekar and Qiao Jin and Guangzhi Xiong and Soren Dunn and Serina S Applebaum and Zain Anwar and Maame Sarfo-Gyamfi and Conrad W Safranek and Abid A Anwar and Andrew Zhang and Aidan Gilson and Maxwell B Singer and Amisha Dave and Andrew Taylor and Aidong Zhang and Qingyu Chen and Zhiyong Lu } ,
year = { 2024 } ,
eprint = { 2406.12036 } ,
archivePrefix = { arXiv } ,
primaryClass = { id='cs.CL' full_name='Computation and Language' is_active=True alt_name='cmp-lg' in_archive='cs' is_general=False description='Covers natural language processing. Roughly includes material in ACM Subject Class I.2.7. Note that work on artificial languages (programming languages, logics, formal systems) that does not explicitly address natural-language issues broadly construed (natural-language processing, computational linguistics, speech, text retrieval, etc.) is not appropriate for this area.' }
}