Suite de flux de travail moléculaires et informatiques pour le séquençage spatial total d'ARN (STRS)
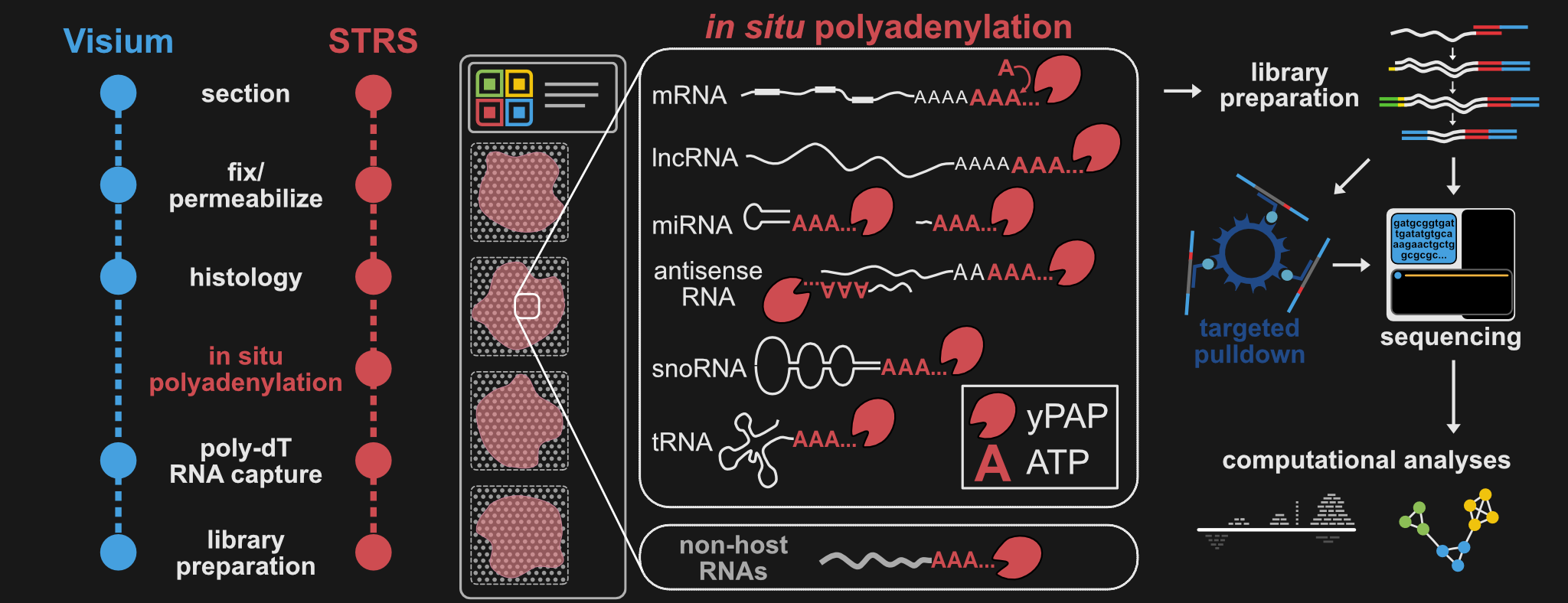
Ce référentiel contient tous les protocoles, pipelines et scripts dont vous aurez besoin pour exécuter et analyser STRS sur presque tous les échantillons !
txg_snake , un pipeline en cours de travail pour les bibliothèques 10x, en particulier les ensembles de données STRS !McKellar et al, Nature Biotechnologie , 2022
lien de préimpression bioRxiv
manuscripts : fichiers .pdf de la prépublication et du manuscrit ultérieur (espérons-le !)
protocols : documents Microsoft word et .pdf des protocoles utilisés ici
pipelines : workflows Snakemake utilisés dans notre étude. Ceux-ci incluent des pipelines d'alignement pour les données STRS (kallisto, STARsolo et miRge3.0), les petites données RNAseq (STAR et miRge3.0), SmartSeqTotal (kallisto) et VASAdrop (kallisto).
scripts : Tous les autres codes que nous avons utilisés ! Contient principalement des scripts R et des fonctions utilitaires utilisés dans nos analyses spatiales.
references : informations & scripts sur les génomes de référence & annotations utilisés dans cette étude
resources : métadonnées variées, listes de gènes et autres informations que nous avons utilisées pour analyser nos données, voir les fichiers README dans chaque sous-répertoire pour plus de détails
Ensembles de données STRS : GSE200481
Données SkM/coeur sur les petites RNAseq : GSE200480
Données RNAseq totales sur un seul noyau (lignée cellulaire C2C12) : GSE209780
Données de l'Atlas des petits ARN (Isakova et al, PNAS , 2020) : GSE119661
Données SmartSeqTotal (Isakova et al, PNAS , 2021) : GSE151334
Données VASAdrop (Salmen et al, Nature Biotechnology , 2022) : GSE176588
Iwijn De Vlaminck ([email protected])
Benjamin D. Cosgrove ([email protected])
David W. McKellar ([email protected])