Ce projet comprend différentes méthodes pour échantillonner la population et évaluer différentes méthodes. Nous incluons de nombreuses situations susceptibles de biaiser l'estimation du niveau d'infection sur la base de l'échantillon, notamment les non-répondeurs, le taux de faux positifs/négatifs, la capacité de profil de transmission des patients pendant leur période d'infection. Basé sur le modèle EpiABM, ce package peut également générer la meilleure méthode d'échantillonnage en exécutant des simulations de transmission de maladies pour voir l'erreur de prédiction de chaque méthode d'échantillonnage.
EpiOS n'est pas encore disponible sur PyPI, mais le module peut être installé localement. Le répertoire doit d'abord être téléchargé sur votre ordinateur local, puis peut être installé à l'aide de la commande :
pip install -e .Nous vous recommandons également d'installer le modèle EpiABM pour générer les données de simulation d'infection. Vous pouvez d'abord télécharger le pyEpiabm à n'importe quel emplacement de votre machine, puis l'installer à l'aide de la commande :
pip install -e path/to/pyEpiabm Les documentations sont accessibles via le badge docs ci-dessus.
Voici un diagramme de classes UML pour notre projet : 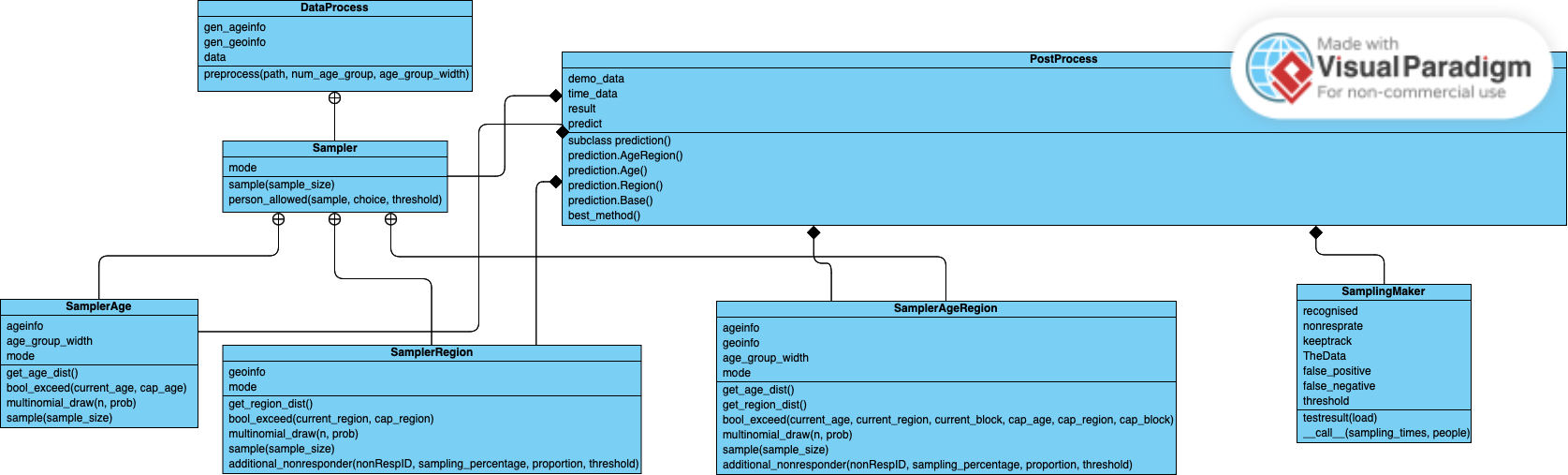
Le fichier params.py inclut tous les paramètres requis dans ce modèle. De plus, les fichiers du dossier input sont des exemples de fichiers temporaires générés lors du prétraitement des données. Il sera utilisé par les classes d'échantillonneurs. Le paramètre data_store_path dans chaque classe d'échantillonneur est le chemin d'accès pour stocker ces fichiers.
PostProcess pour générer des tracés Tout d'abord, vous devez définir un nouvel objet PostProcess et saisir les données démographiques demodata et timedata des données d'infection générées à partir de pyEpiabm. Deuxièmement, vous pouvez utiliser PostProcess.predict pour effectuer une prédiction basée sur différentes méthodes d'échantillonnage. Vous pouvez appeler directement la méthode d'échantillonnage que vous souhaitez utiliser comme méthode ; puis spécifiez les moments d'échantillonnage et la taille de l'échantillon. Ici, nous utiliserons AgeRegion comme méthode d'échantillonnage, [0, 1, 2, 3, 4, 5] comme points temporels à échantillonner et 3 pour être la taille de l'échantillon. Enfin, vous pouvez préciser si vous souhaitez prendre en compte les non-répondants et si vous souhaitez comparer vos résultats avec les vraies données en précisant les paramètres non_responder et comparison .
Pour un exemple de code, vous pouvez voir ce qui suit :
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)Maintenant, votre figurine sera enregistrée sur le chemin indiqué !
PostProcess pour sélectionner la meilleure méthode d'échantillonnage Tout d'abord, vous devez définir un nouvel objet PostProcess et saisir les données démographiques demodata et timedata des données d'infection générées à partir de pyEpiabm. Deuxièmement, vous pouvez utiliser PostProcess.best_method pour comparer les performances de différentes méthodes d'échantillonnage. Vous pouvez fournir les méthodes que vous souhaitez comparer ; puis spécifiez les intervalles d'échantillonnage pour l'échantillon et la taille de l'échantillon. Troisièmement, vous pouvez spécifier si vous souhaitez prendre en compte les non-répondants et si vous souhaitez comparer vos résultats avec les vraies données en spécifiant les paramètres non_responder et comparison . De plus, étant donné que les méthodes d'échantillonnage sont stochastiques, vous pouvez spécifier le nombre d'itérations exécutées pour obtenir les performances moyennes. De plus, parallel_computation peut être activé pour accélérer. Enfin, vous pouvez activer hyperparameter_autotune pour trouver automatiquement la meilleure combinaison d'hyperparamètres.
Pour un exemple de code, vous pouvez voir ce qui suit :
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed