
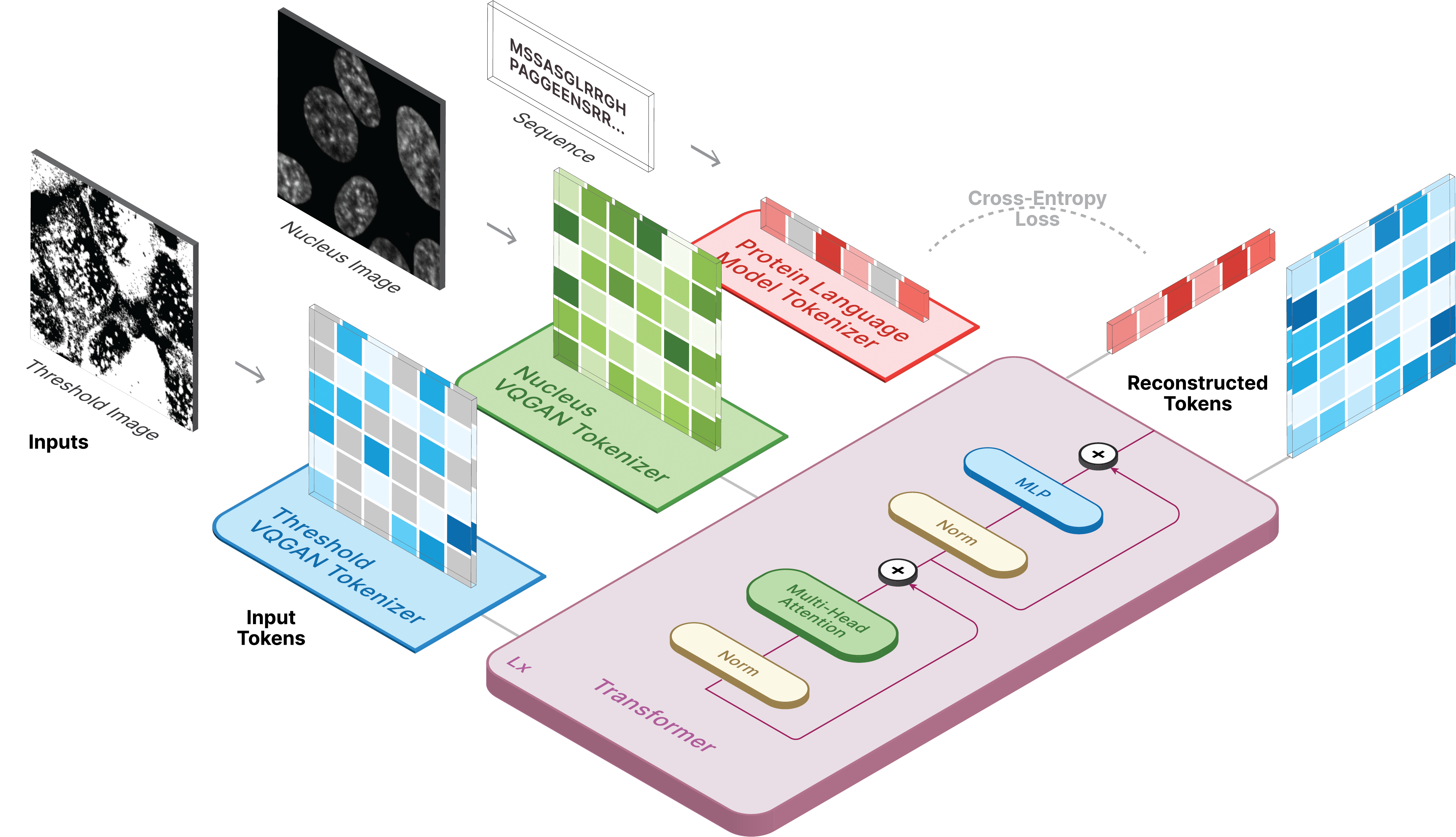
Ce référentiel est l'implémentation officielle de CELL-E 2 : Traduction de protéines en images et inversement avec un transformateur bidirectionnel de texte en image.
Créez un environnement virtuel et installez les packages requis via :
pip install -r requirements.txt
Ensuite, installez torch = 2.0.0 avec la version CUDA appropriée
Les modèles sont disponibles sur Hugging Face.
Nous disposons également de deux espaces où vous pouvez exécuter des prédictions sur vos propres données !
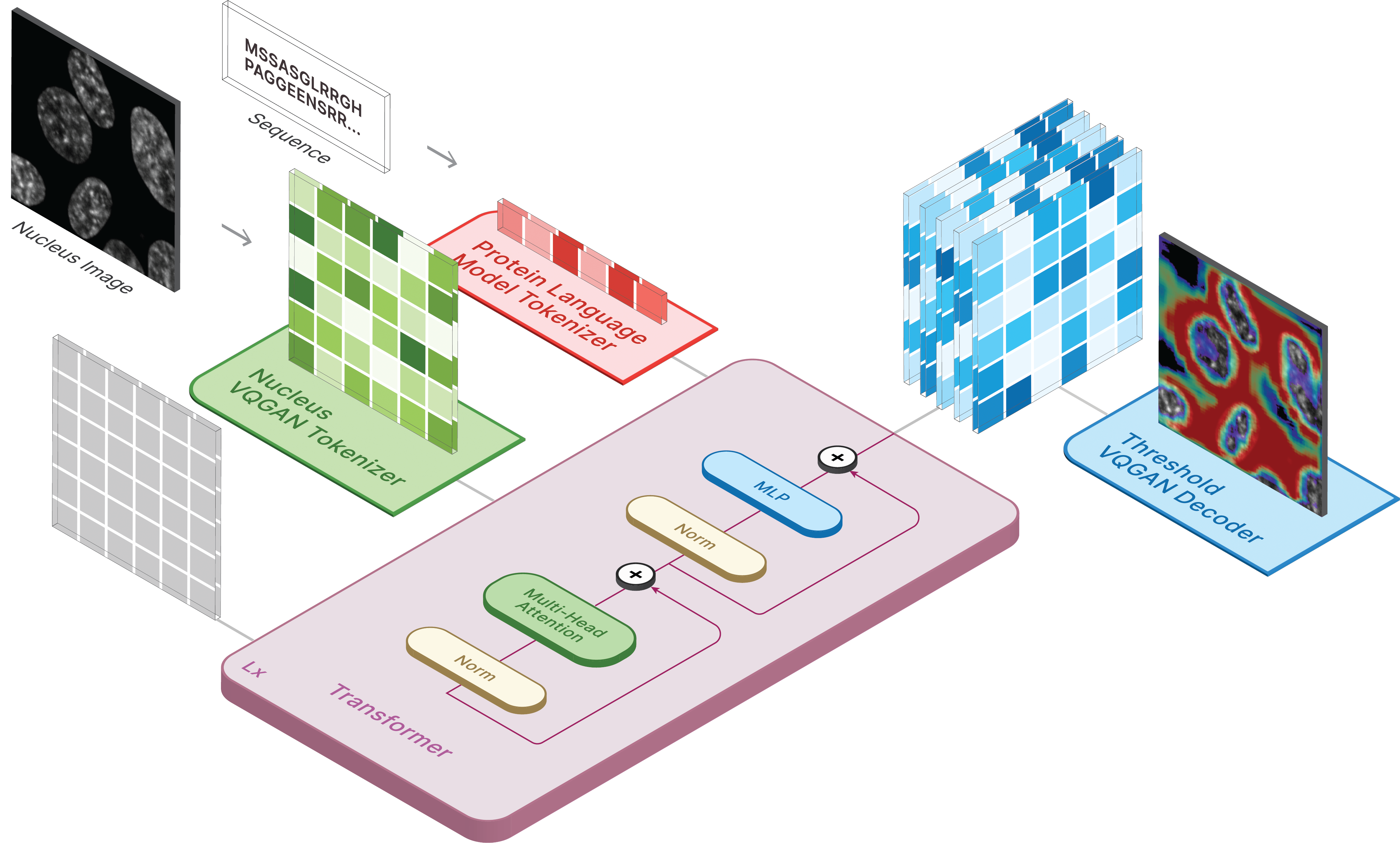
Pour générer des images, définissez le modèle enregistré comme ckpt_path. Cette méthode peut être instable, alors référez-vous à Demo.ipynb pour voir une autre façon de charger.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )La formation pour CELL-E se déroule en 3 étapes :
Si vous utilisez l’image du seuil protéique, définissez threshold: True pour l’ensemble de données.
Nous utilisons une version légèrement modifiée du code taming-transformers.
Pour vous entraîner, exécutez le script suivant :
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
Veuillez vous référer au dépôt d'origine pour les indicateurs supplémentaires, tels que --gpus .
Nous fournissons des scripts pour télécharger les images Human Protein Atlas et OpenCell dans le dossier des scripts. Un data_csv est nécessaire pour le chargeur de données. Vous devez générer un fichier csv contenant les colonnes nucleus_image_path , protein_image_path , metadata_path , split (train ou val) et sequence (facultatif). On suppose que ce fichier existe dans le même dossier data générales que les fichiers d'images et de métadonnées.
Les métadonnées sont un JSON qui doit accompagner chaque séquence protéique. Si une séquence n'apparaît pas dans le data_csv , elle doit apparaître dans metadata.json avec la clé nommée protein_sequence .
L'ajout de plus d'informations ici peut être utile pour interroger des protéines individuelles. Ils peuvent être récupérés via retrieve_metadata , qui crée une variable self.metadata dans l'objet dataset.
Pour vous entraîner, exécutez le script suivant :
python celle_main.py --base configs/celle.yaml -t True
Spécifiez --gpus dans le même format que VQGAN.
CELL-E contient les options suivantes :
ckpt_path : Reprendre la formation CELL-E 2 précédente. Modèle enregistré avec state_dictvqgan_model_path : modèle d'image de protéine enregistré (avec state_dict) pour l'encodeur d'image de protéinevqgan_config_path : Modèle d'image de protéine enregistré yamlcondition_model_path : modèle de condition (noyau) enregistré (avec state_dict) pour l'encodeur d'image protéiquecondition_config_path : modèle de condition (noyau) enregistré yamlnum_images : 1 si vous utilisez uniquement un encodeur d'image protéique, 2 si vous incluez un encodeur d'image de conditionimage_key : nucleus , target ou thresholddim : Dimension de l'intégration du modèle de langagenum_text_tokens : nombre total de jetons dans le modèle de langage (33 pour ESM-2)text_seq_len : Nombre total d'acides aminés considérésdepth : profondeur du modèle de transformateur, une profondeur plus profonde est généralement meilleure au détriment de la VRAMheads : nombre de têtes utilisées dans l'attention multi-têtesdim_head : taille des têtes d'attentionattn_dropout : Taux d'abandon de l'attention en formationff_dropout : Taux d'abandon par feed-forward en formationloss_img_weight : Pondération appliquée à la reconstruction d'image. poids du texte = 1loss_text_weight : Pondération appliquée à la reconstruction de l'image de condition.stable : normes de poids (lorsque des gradients explosifs se produisent)learning_rate : Taux d'apprentissage pour l'optimiseur Adammonitor : Param utilisé pour enregistrer les modèles Veuillez nous citer si vous décidez d'utiliser notre code pour une partie quelconque de votre recherche.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}