
Nous sommes fiers de présenter Asclepius , un modèle de langage clinique plus avancé. Ce modèle ayant été formé sur des notes cliniques synthétiques, il est accessible au public via Huggingface. Si vous envisagez d'utiliser CAMEL, nous vous recommandons fortement de passer à Asclepius. Pour plus d’informations, veuillez visiter ce lien.
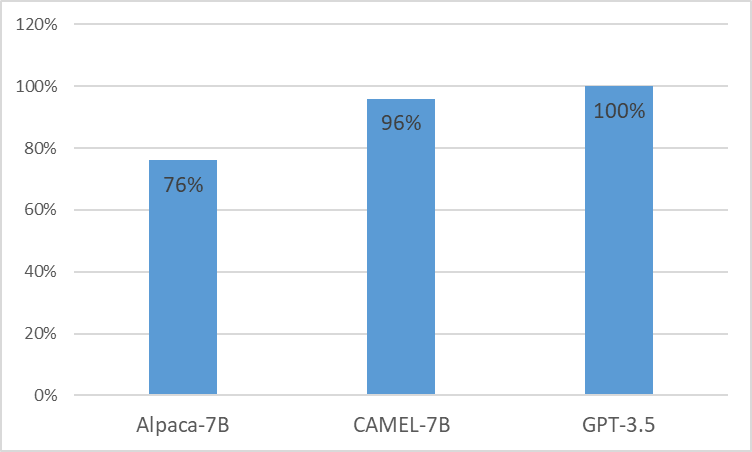
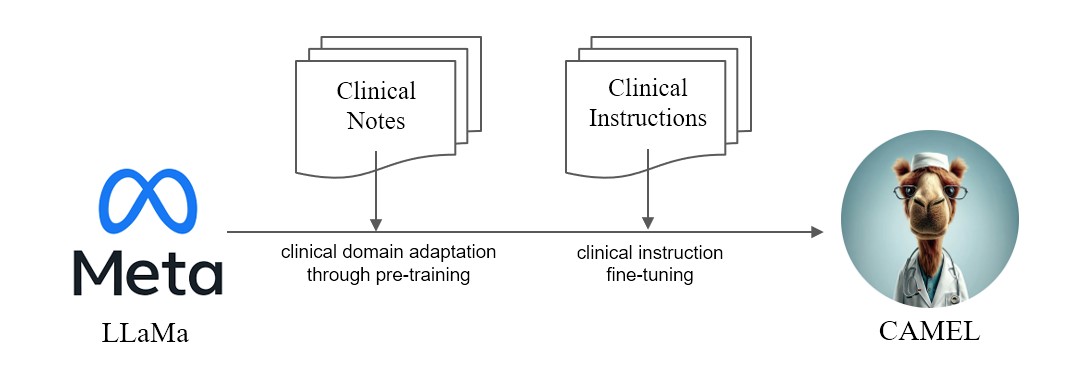
Nous présentons CAMEL , modèle cliniquement adapté amélioré de LLaMA. En tant que LLaMA pour sa fondation, CAMEL est en outre pré-formé sur les notes cliniques MIMIC-III et MIMIC-IV, et affiné sur les instructions cliniques (Figure 2). Notre évaluation préliminaire avec l'évaluation GPT-4 démontre que CAMEL atteint plus de 96 % de la qualité du GPT-3.5 d'OpenAI (Figure 1). Conformément aux politiques d'utilisation des données de nos données sources, notre ensemble de données d'instructions et notre modèle seront publiés sur PhysioNet avec un accès authentifié. Pour faciliter la réplication, nous publierons également tout le code, permettant ainsi à chaque établissement de santé de reproduire notre modèle en utilisant ses propres notes cliniques. Pour plus de détails, veuillez consulter notre article de blog .
En raison du problème de licence des ensembles de données MIMIC et i2b2, nous ne pouvons pas publier l'ensemble de données d'instructions et les points de contrôle. Nous publierions notre modèle et nos données via physionet dans quelques semaines.
conda create -n camel python=3.9 -y
conda activate camel
conda install pytorch torchvision torchaudio pytorch-cuda=11.8 -c pytorch -c nvidia -y
pip install pandarallel pandas jupyter numpy datasets sentencepiece openai fire
pip install git+https://github.com/huggingface/transformers.git@871598be552c38537bc047a409b4a6840ba1c1e4
<eos> .$ python pretraining_preprocess/mimiciii_preproc.py --mimiciii_note_path {MIMICIII_NOTE_PATH} --output_path {OUTPUT_PATH}$ python pretraining_preprocess/mimiciv_preproc.py --discharge_note_path {DISCHAGE_NOTE_PATH} --radiology_note_path {RADIOLOGY_NOTE_PATH} --output_path {OUTPUT_PATH}$ python pretraining_preprocess/tokenize_data.py --data_path {DATA_PATH} --save_path {SAVE_PATH} $ torchrun --nproc_per_node=8 --master_port={YOUR_PORT}
src/train.py
--model_name_or_path "decapoda-research/llama-7b-hf"
--data_path {DATA_FILE}
--bf16 True
--output_dir ./checkpoints
--num_train_epochs 1
--per_device_train_batch_size 2
--per_device_eval_batch_size 2
--gradient_accumulation_steps 8
--evaluation_strategy "no"
--save_strategy "steps"
--save_steps 1000
--learning_rate 2e-5
--weight_decay 0.
--warmup_ratio 0.03
--lr_scheduler_type "cosine"
--logging_steps 1
--fsdp "full_shard auto_wrap"
--fsdp_transformer_layer_cls_to_wrap 'LlamaDecoderLayer'
--tf32 True
--model_max_length 2048
--gradient_checkpointing True
REMARQUE : Pour générer des instructions, vous devez utiliser l’API Azure Openai certifiée.
Génération d'instructions
OPENAI_API_KEYOPENAI_API_BASEOPENAI_DEPLOYMENT_NAME$ python instructino/preprocess_note.py$ python instruction/de_id_gen.py --input {PREPROCESSED_NOTES} --output {OUTPUT_FILE_1} --mode inst$ python instruction/de_id_postprocess.py --input {OUTPUT_FILE_1} --output {OUTPUT_FILE_2}$ python instruction/de_id_gen.py --input {OUTPUT__FILE_2} --output {inst_output/OUTPUT_FILE_deid} --mode ans$ python instruction/instructtion_gen.py --input {PREPROCESSED_NOTES} --output {inst_output/OUTPUT_FILE} --source {mimiciii, mimiciv, i2b2}$ python instruction/merge_data.py --data_path {inst_output} --output {OUTPUT_FILE_FINAL}Exécuter le réglage fin des instructions
nproc_per_node et gradient accumulate step pour les adapter à votre matériel (taille globale du lot = 128). $ torchrun --nproc_per_node=8 --master_port={YOUR_PORT}
src/instruction_ft.py
--model_name_or_path "decapoda-research/llama-7b-hf"
--data_path {OUTPUT_FILE_FINAL}
--bf16 True
--output_dir ./checkpoints
--num_train_epochs 3
--per_device_train_batch_size 2
--per_device_eval_batch_size 2
--gradient_accumulation_steps 8
--evaluation_strategy "no"
--save_strategy "epoch"
--learning_rate 2e-5
--weight_decay 0.
--warmup_ratio 0.03
--lr_scheduler_type "cosine"
--logging_steps 1
--fsdp "full_shard auto_wrap"
--fsdp_transformer_layer_cls_to_wrap 'LlamaDecoderLayer'
--tf32 True
--model_max_length 2048
--gradient_checkpointing True
--ddp_timeout 18000
Exécuter le modèle sur MTSamples
CUDA_VISIBLE_DEVICES=0 python src/evaluate.py
--model_name {MODEL_PATH}
--data_path eval/mtsamples_instructions.json
--output_path {OUTPUT_PATH}
mtsamples_results.json dans le dossier eval .Exécutez GPT-4 pour évaluation
python eval/gpt4_evaluate.py --input {INPUT_PATH} --output {OUTPUT_PATH}
@misc{CAMEL,
title = {CAMEL : Clinically Adapted Model Enhanced from LLaMA},
author = {Sunjun Kweon and Junu Kim and Seongsu Bae and Eunbyeol Cho and Sujeong Im and Jiyoun Kim and Gyubok Lee and JongHak Moon and JeongWoo Oh and Edward Choi},
month = {May},
year = {2023}
publisher = {GitHub},
journal = {GitHub repository},
howpublished = {url{https://github.com/starmpcc/CAMEL}},
}