SHAP (SHapley Additive exPlanations) est une approche de la théorie des jeux pour expliquer le résultat de tout modèle d'apprentissage automatique. Il relie l'allocation optimale de crédits aux explications locales en utilisant les valeurs classiques de Shapley issues de la théorie des jeux et leurs extensions associées (voir les articles pour plus de détails et les citations).
SHAP peut être installé à partir de PyPI ou de conda-forge :
pip installer la forme ou conda install -c forme conda-forge
Bien que SHAP puisse expliquer le résultat de n'importe quel modèle d'apprentissage automatique, nous avons développé un algorithme exact à grande vitesse pour les méthodes d'ensemble d'arbres (voir notre article Nature MI). Les implémentations C++ rapides sont prises en charge pour les modèles d'arborescence XGBoost , LightGBM , CatBoost , scikit-learn et pyspark :
import xgboost
import shap
# train an XGBoost model
X , y = shap . datasets . california ()
model = xgboost . XGBRegressor (). fit ( X , y )
# explain the model's predictions using SHAP
# (same syntax works for LightGBM, CatBoost, scikit-learn, transformers, Spark, etc.)
explainer = shap . Explainer ( model )
shap_values = explainer ( X )
# visualize the first prediction's explanation
shap . plots . waterfall ( shap_values [ 0 ])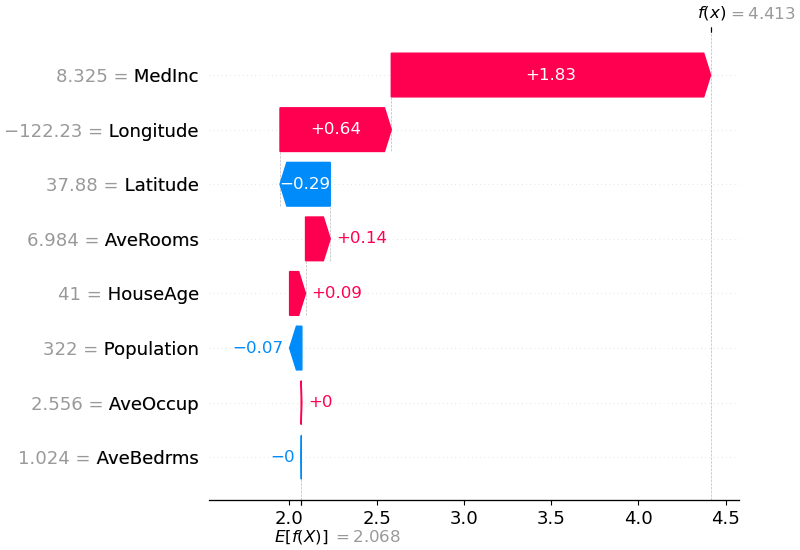
L'explication ci-dessus montre les fonctionnalités contribuant chacune à faire passer la sortie du modèle de la valeur de base (la sortie moyenne du modèle sur l'ensemble de données d'entraînement que nous avons transmis) à la sortie du modèle. Les caractéristiques qui poussent la prédiction vers le haut sont affichées en rouge, celles qui poussent la prédiction vers le bas sont en bleu. Une autre façon de visualiser la même explication consiste à utiliser un diagramme de force (ceux-ci sont présentés dans notre article Nature BME) :
# visualize the first prediction's explanation with a force plot
shap . plots . force ( shap_values [ 0 ])
Si nous prenons de nombreuses explications de tracé de force telles que celle présentée ci-dessus, les faisons pivoter de 90 degrés, puis les empilons horizontalement, nous pouvons voir des explications pour un ensemble de données entier (dans le cahier, ce tracé est interactif) :
# visualize all the training set predictions
shap . plots . force ( shap_values [: 500 ])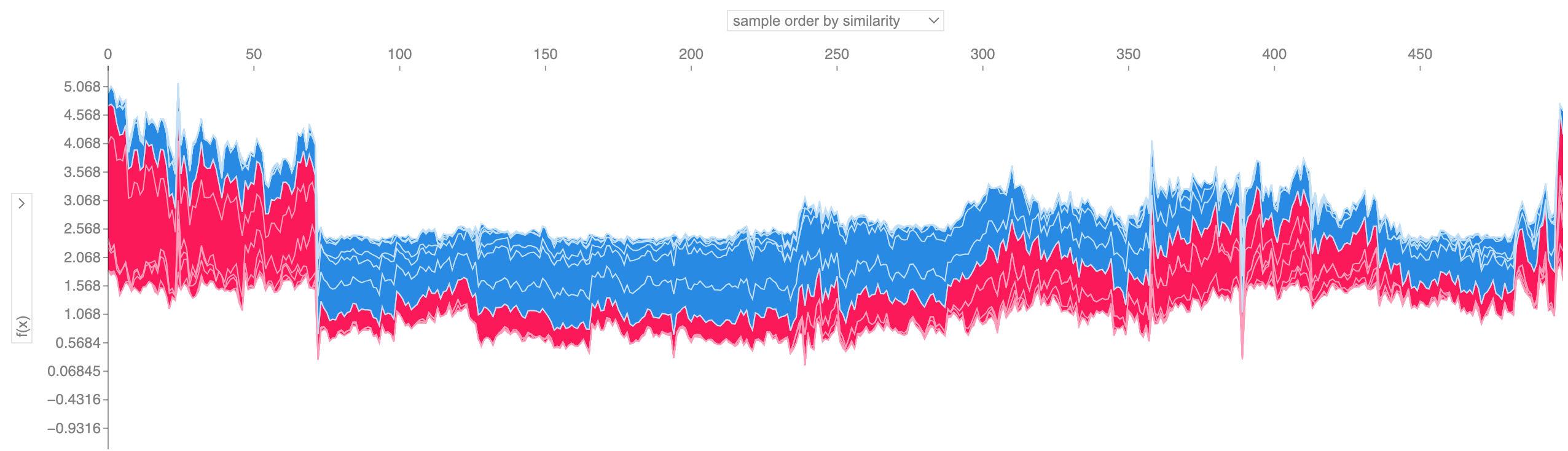
Pour comprendre comment une seule fonctionnalité affecte la sortie du modèle, nous pouvons tracer la valeur SHAP de cette fonctionnalité par rapport à la valeur de la fonctionnalité pour tous les exemples d'un ensemble de données. Étant donné que les valeurs SHAP représentent la responsabilité d'une entité dans une modification de la sortie du modèle, le graphique ci-dessous représente la modification du prix prévu de l'immobilier à mesure que la latitude change. La dispersion verticale à une valeur unique de latitude représente les effets d'interaction avec d'autres entités. Pour aider à révéler ces interactions, nous pouvons colorier par une autre fonctionnalité. Si nous transmettons l'intégralité du tenseur d'explication à l'argument color , le nuage de points sélectionnera la meilleure caractéristique à colorier. Dans ce cas, il choisit la longitude.
# create a dependence scatter plot to show the effect of a single feature across the whole dataset
shap . plots . scatter ( shap_values [:, "Latitude" ], color = shap_values )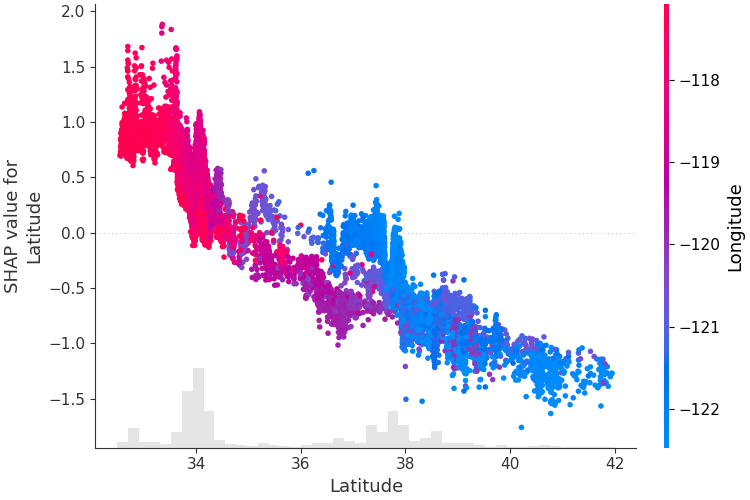
Pour avoir un aperçu des fonctionnalités les plus importantes pour un modèle, nous pouvons tracer les valeurs SHAP de chaque fonctionnalité pour chaque échantillon. Le graphique ci-dessous trie les entités en fonction de la somme des amplitudes des valeurs SHAP sur tous les échantillons et utilise les valeurs SHAP pour montrer la distribution des impacts de chaque entité sur la sortie du modèle. La couleur représente la valeur de la caractéristique (rouge haut, bleu bas). Cela révèle par exemple que des revenus médians plus élevés améliorent le prix prévu de l’immobilier.
# summarize the effects of all the features
shap . plots . beeswarm ( shap_values )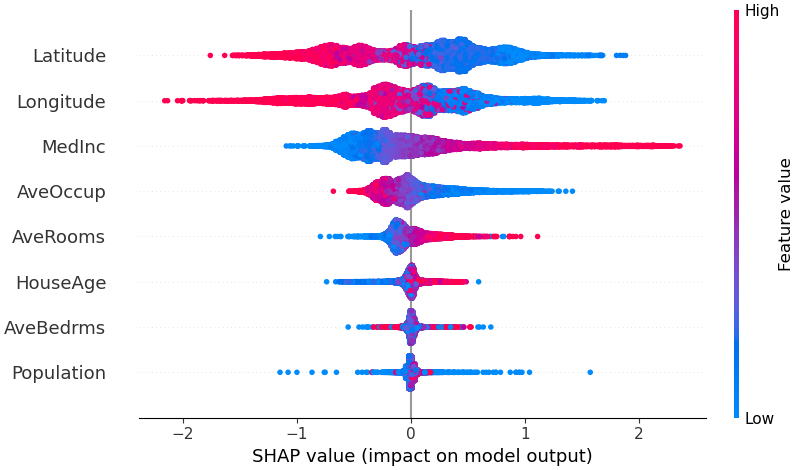
Nous pouvons également simplement prendre la valeur absolue moyenne des valeurs SHAP pour chaque fonctionnalité pour obtenir un tracé à barres standard (produit des barres empilées pour les sorties multi-classes) :
shap . plots . bar ( shap_values )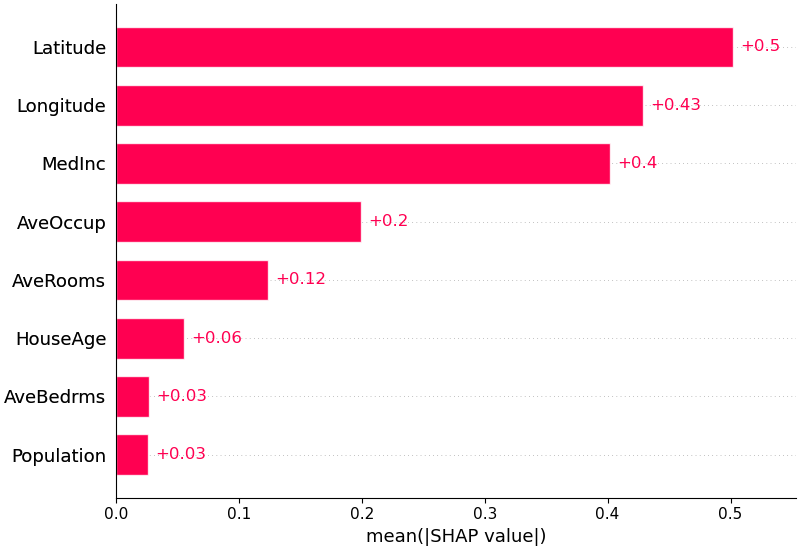
SHAP prend en charge spécifiquement les modèles de langage naturel comme ceux de la bibliothèque de transformateurs Hugging Face. En ajoutant des règles de coalition aux valeurs traditionnelles de Shapley, nous pouvons former des jeux qui expliquent le grand modèle PNL moderne en utilisant très peu d'évaluations de fonctions. Utiliser cette fonctionnalité est aussi simple que de transmettre un pipeline de transformateurs pris en charge à SHAP :
import transformers
import shap
# load a transformers pipeline model
model = transformers . pipeline ( 'sentiment-analysis' , return_all_scores = True )
# explain the model on two sample inputs
explainer = shap . Explainer ( model )
shap_values = explainer ([ "What a great movie! ...if you have no taste." ])
# visualize the first prediction's explanation for the POSITIVE output class
shap . plots . text ( shap_values [ 0 , :, "POSITIVE" ])Deep SHAP est un algorithme d'approximation à grande vitesse pour les valeurs SHAP dans les modèles d'apprentissage profond qui s'appuie sur une connexion avec DeepLIFT décrite dans l'article SHAP NIPS. L'implémentation ici diffère du DeepLIFT original en utilisant une distribution d'échantillons de fond au lieu d'une valeur de référence unique et en utilisant les équations de Shapley pour linéariser les composants tels que max, softmax, produits, divisions, etc. Notez que certaines de ces améliorations ont également été depuis intégré dans DeepLIFT. Les modèles TensorFlow et Keras utilisant le backend TensorFlow sont pris en charge (il existe également une prise en charge préliminaire pour PyTorch) :
# ...include code from https://github.com/keras-team/keras/blob/master/examples/demo_mnist_convnet.py
import shap
import numpy as np
# select a set of background examples to take an expectation over
background = x_train [ np . random . choice ( x_train . shape [ 0 ], 100 , replace = False )]
# explain predictions of the model on four images
e = shap . DeepExplainer ( model , background )
# ...or pass tensors directly
# e = shap.DeepExplainer((model.layers[0].input, model.layers[-1].output), background)
shap_values = e . shap_values ( x_test [ 1 : 5 ])
# plot the feature attributions
shap . image_plot ( shap_values , - x_test [ 1 : 5 ])Le graphique ci-dessus explique dix sorties (chiffres 0 à 9) pour quatre images différentes. Les pixels rouges augmentent la sortie du modèle tandis que les pixels bleus la diminuent. Les images d'entrée sont affichées à gauche et sous forme de supports en niveaux de gris presque transparents derrière chacune des explications. La somme des valeurs SHAP est égale à la différence entre la sortie attendue du modèle (moyenne sur l'ensemble de données d'arrière-plan) et la sortie actuelle du modèle. Notez que pour l'image « zéro », le milieu vide est important, tandis que pour l'image « quatre », l'absence de connexion en haut en fait un quatre au lieu d'un neuf.
Les dégradés attendus combinent les idées de Integrated Gradients, SHAP et SmoothGrad en une seule équation de valeur attendue. Cela permet d'utiliser un ensemble de données entier comme distribution d'arrière-plan (par opposition à une seule valeur de référence) et permet un lissage local. Si nous approchons le modèle avec une fonction linéaire entre chaque échantillon de données de fond et l'entrée actuelle à expliquer, et que nous supposons que les caractéristiques d'entrée sont indépendantes, les gradients attendus calculeront les valeurs SHAP approximatives. Dans l'exemple ci-dessous, nous avons expliqué comment la 7ème couche intermédiaire du modèle VGG16 ImageNet impacte les probabilités de sortie.
from keras . applications . vgg16 import VGG16
from keras . applications . vgg16 import preprocess_input
import keras . backend as K
import numpy as np
import json
import shap
# load pre-trained model and choose two images to explain
model = VGG16 ( weights = 'imagenet' , include_top = True )
X , y = shap . datasets . imagenet50 ()
to_explain = X [[ 39 , 41 ]]
# load the ImageNet class names
url = "https://s3.amazonaws.com/deep-learning-models/image-models/imagenet_class_index.json"
fname = shap . datasets . cache ( url )
with open ( fname ) as f :
class_names = json . load ( f )
# explain how the input to the 7th layer of the model explains the top two classes
def map2layer ( x , layer ):
feed_dict = dict ( zip ([ model . layers [ 0 ]. input ], [ preprocess_input ( x . copy ())]))
return K . get_session (). run ( model . layers [ layer ]. input , feed_dict )
e = shap . GradientExplainer (
( model . layers [ 7 ]. input , model . layers [ - 1 ]. output ),
map2layer ( X , 7 ),
local_smoothing = 0 # std dev of smoothing noise
)
shap_values , indexes = e . shap_values ( map2layer ( to_explain , 7 ), ranked_outputs = 2 )
# get the names for the classes
index_names = np . vectorize ( lambda x : class_names [ str ( x )][ 1 ])( indexes )
# plot the explanations
shap . image_plot ( shap_values , to_explain , index_names ) Les prédictions pour deux images d'entrée sont expliquées dans le graphique ci-dessus. Les pixels rouges représentent des valeurs SHAP positives qui augmentent la probabilité de la classe, tandis que les pixels bleus représentent des valeurs SHAP négatives qui réduisent la probabilité de la classe. En utilisant ranked_outputs=2 nous expliquons uniquement les deux classes les plus probables pour chaque entrée (cela nous évite d'expliquer les 1 000 classes).
Kernel SHAP utilise une régression linéaire locale spécialement pondérée pour estimer les valeurs SHAP pour n'importe quel modèle. Vous trouverez ci-dessous un exemple simple pour expliquer un SVM multiclasse sur l'ensemble de données d'iris classique.
import sklearn
import shap
from sklearn . model_selection import train_test_split
# print the JS visualization code to the notebook
shap . initjs ()
# train a SVM classifier
X_train , X_test , Y_train , Y_test = train_test_split ( * shap . datasets . iris (), test_size = 0.2 , random_state = 0 )
svm = sklearn . svm . SVC ( kernel = 'rbf' , probability = True )
svm . fit ( X_train , Y_train )
# use Kernel SHAP to explain test set predictions
explainer = shap . KernelExplainer ( svm . predict_proba , X_train , link = "logit" )
shap_values = explainer . shap_values ( X_test , nsamples = 100 )
# plot the SHAP values for the Setosa output of the first instance
shap . force_plot ( explainer . expected_value [ 0 ], shap_values [ 0 ][ 0 ,:], X_test . iloc [ 0 ,:], link = "logit" )L'explication ci-dessus montre quatre fonctionnalités, chacune contribuant à pousser la sortie du modèle de la valeur de base (la sortie moyenne du modèle sur l'ensemble de données d'entraînement que nous avons transmis) vers zéro. S'il y avait des fonctionnalités poussant l'étiquette de classe plus haut, elles seraient affichées en rouge.
Si nous prenons de nombreuses explications telles que celle présentée ci-dessus, les faisons pivoter de 90 degrés, puis les empilons horizontalement, nous pouvons voir des explications pour un ensemble de données entier. C'est exactement ce que nous faisons ci-dessous pour tous les exemples de l'ensemble de tests d'iris :
# plot the SHAP values for the Setosa output of all instances
shap . force_plot ( explainer . expected_value [ 0 ], shap_values [ 0 ], X_test , link = "logit" ) Les valeurs d'interaction SHAP sont une généralisation des valeurs SHAP aux interactions d'ordre supérieur. Un calcul rapide et exact des interactions par paires est implémenté pour les modèles d'arbres avec shap.TreeExplainer(model).shap_interaction_values(X) . Cela renvoie une matrice pour chaque prédiction, où les effets principaux se situent sur la diagonale et les effets d'interaction sont hors diagonale. Ces valeurs révèlent souvent des relations cachées intéressantes, comme par exemple le fait que le risque accru de décès culmine chez les hommes à 60 ans (voir le carnet NHANES pour plus de détails) :
Les blocs-notes ci-dessous présentent différents cas d'utilisation de SHAP. Regardez dans le répertoire notebooks du référentiel si vous souhaitez essayer de jouer vous-même avec les notebooks d'origine.
Une implémentation de Tree SHAP, un algorithme rapide et précis pour calculer les valeurs SHAP pour les arbres et les ensembles d'arbres.
Modèle de survie NHANES avec valeurs d'interaction XGBoost et SHAP - En utilisant les données de mortalité de 20 ans de suivi, ce cahier montre comment utiliser XGBoost et shap pour découvrir des relations complexes entre facteurs de risque.
Classification des revenus du recensement avec LightGBM - À l'aide de l'ensemble de données standard sur le revenu du recensement des adultes, ce bloc-notes entraîne un modèle d'arbre augmentant le gradient avec LightGBM, puis explique les prédictions à l'aide shap .
Prédiction des victoires de League of Legends avec XGBoost - À l'aide d'un ensemble de données Kaggle de 180 000 matchs classés de League of Legends, nous entraînons et expliquons un modèle d'arbre d'amélioration du gradient avec XGBoost pour prédire si un joueur gagnera son match.
Une implémentation de Deep SHAP, un algorithme plus rapide (mais seulement approximatif) pour calculer les valeurs SHAP pour les modèles d'apprentissage profond basé sur les connexions entre SHAP et l'algorithme DeepLIFT.
Classification des chiffres MNIST avec Keras - À l'aide de l'ensemble de données de reconnaissance d'écriture manuscrite MNIST, ce cahier entraîne un réseau neuronal avec Keras, puis explique les prédictions à l'aide shap .
Keras LSTM pour la classification des sentiments IMDB - Ce bloc-notes entraîne un LSTM avec Keras sur l'ensemble de données d'analyse des sentiments du texte IMDB, puis explique les prédictions à l'aide shap .
Une implémentation des gradients attendus pour approximer les valeurs SHAP pour les modèles d'apprentissage en profondeur. Il est basé sur des connexions entre SHAP et l’algorithme Integrated Gradients. GradientExplainer est plus lent que DeepExplainer et fait des hypothèses d'approximation différentes.
Pour un modèle linéaire avec des caractéristiques indépendantes, nous pouvons calculer analytiquement les valeurs SHAP exactes. Nous pouvons également prendre en compte la corrélation des caractéristiques si nous souhaitons estimer la matrice de covariance des caractéristiques. LinearExplainer prend en charge ces deux options.
Une implémentation de Kernel SHAP, une méthode indépendante du modèle pour estimer les valeurs SHAP pour n'importe quel modèle. Parce qu'il ne fait aucune hypothèse sur le type de modèle, KernelExplainer est plus lent que les autres algorithmes spécifiques au type de modèle.
Classification des revenus du recensement avec scikit-learn - À l'aide de l'ensemble de données standard sur le revenu du recensement des adultes, ce cahier forme un classificateur des k voisins les plus proches à l'aide de scikit-learn, puis explique les prédictions à l'aide shap .
Modèle ImageNet VGG16 avec Keras - Expliquez les prédictions du réseau neuronal convolutif classique VGG16 pour une image. Cela fonctionne en appliquant la méthode Kernel SHAP indépendante du modèle à une image segmentée en super-pixels.
Classification de l'iris - Une démonstration de base utilisant l'ensemble de données populaire sur les espèces d'iris. Il explique les prédictions de six modèles différents dans scikit-learn en utilisant shap .
Ces cahiers montrent de manière exhaustive comment utiliser des fonctions et des objets spécifiques.
shap.decision_plot et shap.multioutput_decision_plot
shap.dependence_plot
LIME : Ribeiro, Marco Tulio, Sameer Singh et Carlos Guestrin. "Pourquoi devrais-je vous faire confiance ? : Expliquer les prédictions de n'importe quel classificateur." Actes de la 22e Conférence internationale ACM SIGKDD sur la découverte des connaissances et l'exploration de données. ACM, 2016.
Valeurs d'échantillonnage de Shapley : Strumbelj, Erik et Igor Kononenko. "Expliquer les modèles de prédiction et les prédictions individuelles avec des contributions aux fonctionnalités." Systèmes de connaissances et d'information 41.3 (2014) : 647-665.
DeepLIFT : Shrikumar, Avanti, Peyton Greenside et Anshul Kundaje. "Apprendre des fonctionnalités importantes en propageant les différences d'activation." Préimpression arXiv arXiv:1704.02685 (2017).
QII : Datta, Anupam, Shayak Sen et Yair Zick. "Transparence algorithmique via l'influence quantitative des entrées : théorie et expériences avec des systèmes d'apprentissage." Sécurité et confidentialité (SP), Symposium IEEE 2016 sur. IEEE, 2016.
Propagation de la pertinence par couche : Bach, Sebastian et al. "Sur les explications par pixels des décisions de classificateur non linéaire par propagation de pertinence par couche." PloS un 10.7 (2015) : e0130140.
Valeurs de régression de Shapley : Lipovetsky, Stan et Michael Conklin. "Analyse de la régression dans l'approche de la théorie des jeux." Modèles stochastiques appliqués aux affaires et à l'industrie 17.4 (2001) : 319-330.
Interprète des arbres : Saabas, Ando. Interprétation des forêts aléatoires. http://blog.datadive.net/interpreting-random-forests/
Les algorithmes et les visualisations utilisés dans ce package sont principalement issus de recherches menées dans le laboratoire de Su-In Lee à l'Université de Washington et de Microsoft Research. Si vous utilisez SHAP dans votre recherche, nous apprécierions une citation du ou des articles appropriés :
force_plot et les applications médicales, vous pouvez lire/citer notre article Nature Biomedical Engineering (bibtex ; accès gratuit).