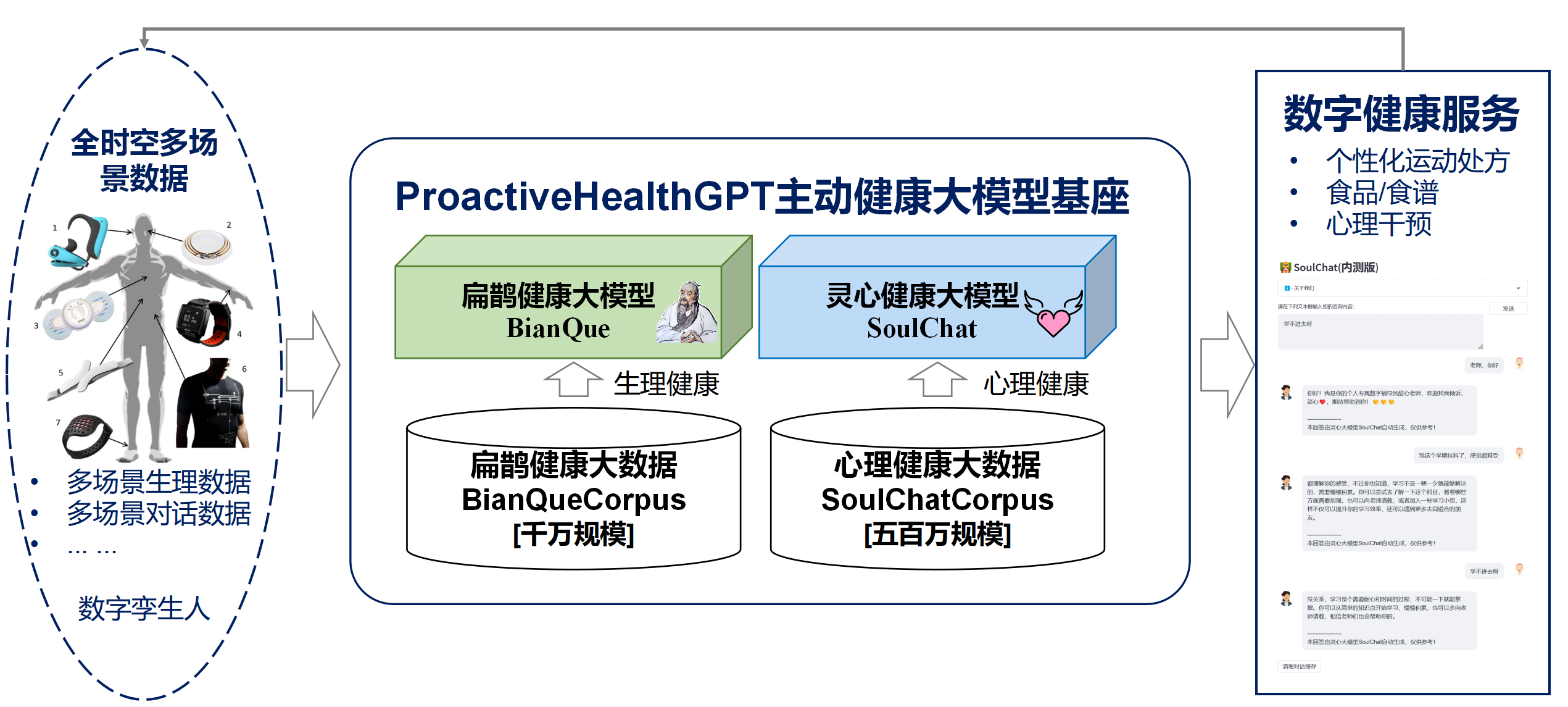
Basé sur les six caractéristiques de la santé active : initiative, prévention, précision, personnalisation, co-construction et partage, et autodiscipline, l'École des technologies du futur de l'Université de technologie de Chine du Sud-Laboratoire clé provincial des jumeaux numériques du Guangdong a lancé un projet open source. le système de santé actif de l'espace de vie dans le domaine chinois. Base modèle ProactiveHealthGPT, comprenant :
Nous espérons que la grande base de modèles de santé active de l'espace de vie ProactiveHealthGPT pourra aider la communauté universitaire à accélérer la recherche et l'application de grands modèles dans les domaines de la santé active tels que les maladies chroniques et le conseil psychologique. Ce projet est BianQue, un grand modèle de santé des espaces de vie .
Grâce à des recherches, nous avons constaté que dans le domaine de la santé, les utilisateurs ne décrivent généralement pas clairement leurs problèmes lors d'une série d'interactions et que les modèles de questions et réponses médicales open source actuellement courants (tels que : ChatDoctor, HuaTuo (anciennement connu sous le nom de HuaTuo), DoctorGLM , MedicalGPT -zh) se concentre sur la résolution du problème d'une seule série de descriptions d'utilisateurs, tout en ignorant la situation dans laquelle « les descriptions d'utilisateurs peuvent être insuffisantes ». Même le ChatGPT actuellement populaire rencontrera des problèmes similaires : si l'utilisateur ne force pas ChatGPT à adopter un format de questions et réponses via une description textuelle, ChatGPT préférera également la description de l'utilisateur et fournira rapidement des suggestions et des solutions qu'il juge appropriées. Cependant, les conversations réelles entre médecins et utilisateurs impliquent souvent « le médecin menant plusieurs séries de questions basées sur la description actuelle de l'utilisateur ». Et le médecin donne enfin des suggestions complètes basées sur les informations fournies par l'utilisateur, comme le montre la figure ci-dessous. Nous définissons le processus de questionnement continu du médecin comme une chaîne de questionnement (CoQ, Chain of Questioning) . Lorsque le modèle est au stade de la chaîne de questionnement, sa prochaine question est généralement déterminée par l'historique du contexte de la conversation.

Nous combinons les ensembles de données de questions et réponses médicales chinoises open source actuelles (MedDialog-CN, IMCS-V2, CHIP-MDCFNPC, MedDG, cMedQA2, Chinese-medical-dialogue-data) pour analyser les caractéristiques à un seul tour/multi-tours et caractéristiques de l'enquête du médecin, combinées aux mégadonnées de conversation sur la santé de l'espace de vie auto-construites à long terme du laboratoire, BianQueCorpus, un big data de santé Bianque à l'échelle de dizaines de millions, a été construit. Les données de conversation sont unifiées dans un format d'instruction sous la forme « Patient : xxxnMédecin : xxxnPatient : xxxnDocteur : », comme le montre la figure ci-dessous.
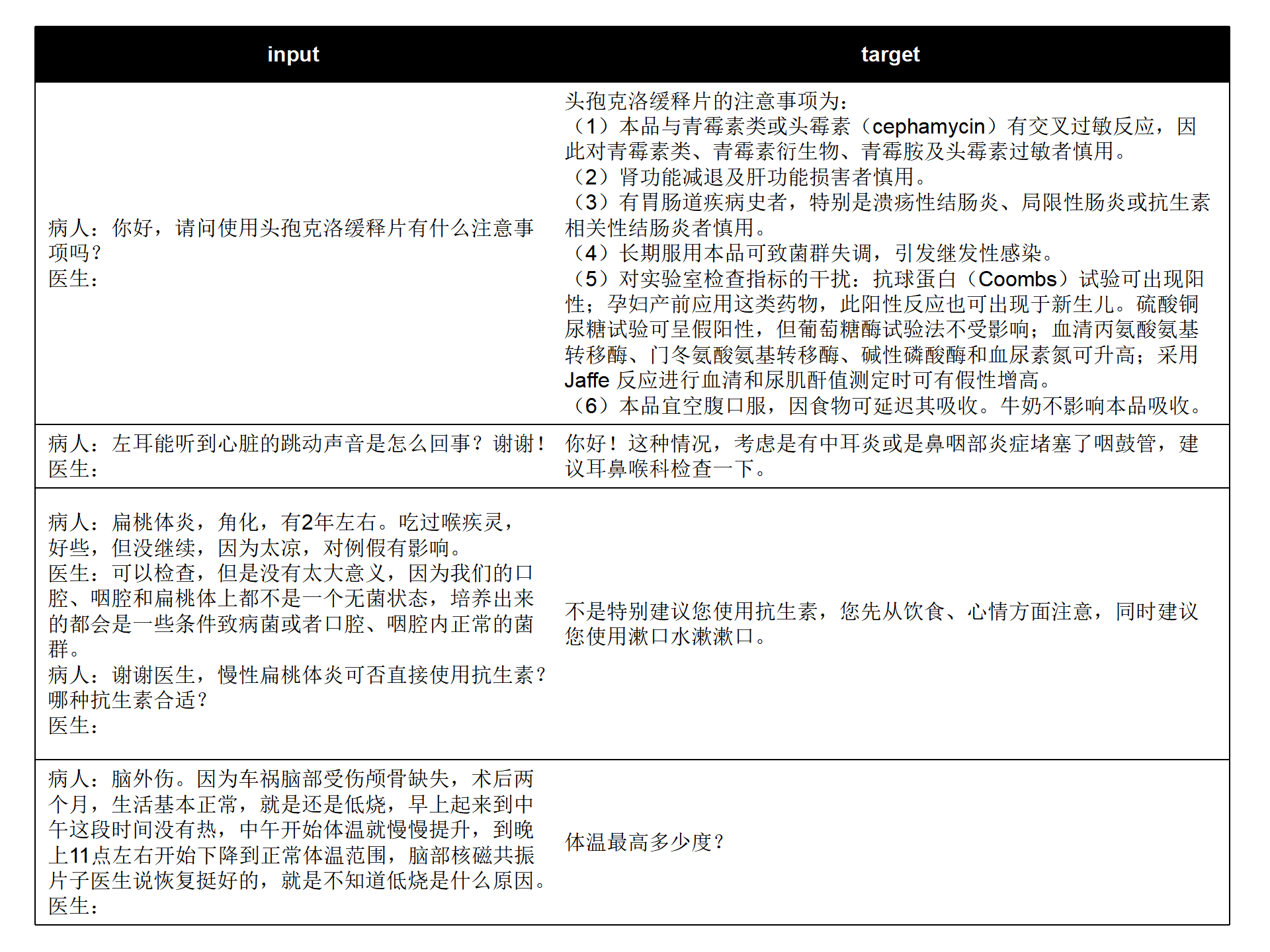
input: "病人:六岁宝宝拉大便都是一个礼拜或者10天才一次正常吗,要去医院检查什么项目n医生:您好n病人:六岁宝宝拉大便都是一个礼拜或者10天才一次正常吗,要去医院检查什么项目n医生:宝宝之前大便什么样呢?多久一次呢n病人:一般都是一个礼拜,最近这几个月都是10多天n医生:大便干吗?n病人:每次10多天拉的很多n医生:"
target: "成形还是不成形呢?孩子吃饭怎么样呢?"
Les données de formation contiennent une grande quantité de texte cible qui constitue le contenu de la demande du médecin plutôt que des conseils directs, ce qui contribuera à améliorer la capacité de recherche du modèle d'IA.
cd ~
git clone https://github.com/scutcyr/BianQue.git cd BianQue
conda env create -n proactivehealthgpt_py38 --file proactivehealthgpt_py38.yml
conda activate proactivehealthgpt_py38
pip install cpm_kernels
pip install torch==1.13.1+cu116 torchvision==0.14.1+cu116 torchaudio==0.13.1 --extra-index-url https://download.pytorch.org/whl/cu116 cd BianQue
conda create -n proactivehealthgpt_py38 python=3.8
conda activate proactivehealthgpt_py38
pip install torch==1.13.1+cu116 torchvision==0.14.1+cu116 torchaudio==0.13.1 --extra-index-url https://download.pytorch.org/whl/cu116
pip install -r requirements.txt
pip install rouge_chinese nltk jieba datasets
# 以下安装为了运行demo
pip install streamlit
pip install streamlit_chat[Supplément] Configuration de CUDA-11.6 sous Windows : Téléchargez et installez CUDA-11.6, téléchargez cudnn-8.4.0, décompressez et copiez les fichiers dans le chemin correspondant à CUDA-11.6, reportez-vous à : Utiliser conda pour installer pytorch sous win11-cuda11 . 6-Idées générales d'installation
Appelez le modèle BianQue-2.0 en Python :
import torch
from transformers import AutoModel , AutoTokenizer
# GPU设置
device = torch . device ( "cuda" if torch . cuda . is_available () else "cpu" )
# 加载模型与tokenizer
model_name_or_path = 'scutcyr/BianQue-2'
model = AutoModel . from_pretrained ( model_name_or_path , trust_remote_code = True ). half ()
model . to ( device )
tokenizer = AutoTokenizer . from_pretrained ( model_name_or_path , trust_remote_code = True )
# 单轮对话调用模型的chat函数
user_input = "我的宝宝发烧了,怎么办?"
input_text = "病人:" + user_input + " n医生:"
response , history = model . chat ( tokenizer , query = input_text , history = None , max_length = 2048 , num_beams = 1 , do_sample = True , top_p = 0.75 , temperature = 0.95 , logits_processor = None )
# 多轮对话调用模型的chat函数
# 注意:本项目使用"n病人:"和"n医生:"划分不同轮次的对话历史
# 注意:user_history比bot_history的长度多1
user_history = [ '你好' , '我最近失眠了' ]
bot_history = [ '我是利用人工智能技术,结合大数据训练得到的智能医疗问答模型扁鹊,你可以向我提问。' ]
# 拼接对话历史
context = " n " . join ([ f"病人: { user_history [ i ] } n医生: { bot_history [ i ] } " for i in range ( len ( bot_history ))])
input_text = context + " n病人:" + user_history [ - 1 ] + " n医生:"
response , history = model . chat ( tokenizer , query = input_text , history = None , max_length = 2048 , num_beams = 1 , do_sample = True , top_p = 0.75 , temperature = 0.95 , logits_processor = None )Ce projet fournit bianque_v2_app.py comme exemple d'utilisation du modèle BianQue-2.0. Vous pouvez démarrer le service via la commande suivante, puis y accéder via http://<your_ip>:9005.
streamlit run bianque_v2_app.py --server.port 9005En particulier, dans bianque_v2_app.py, vous pouvez modifier le code suivant pour remplacer la carte graphique spécifiée :
os . environ [ 'CUDA_VISIBLE_DEVICES' ] = '1' Pour les utilisateurs d'une seule carte graphique Windows, vous devez la modifier comme suit : os.environ['CUDA_VISIBLE_DEVICES'] = '0' , sinon une erreur sera signalée !
Vous pouvez spécifier le chemin du modèle comme étant un chemin local en modifiant le code suivant :
model_name_or_path = "scutcyr/BianQue-2"Nous fournissons également bianque_v1_app.py comme exemple d'utilisation du modèle BianQue-1.0, et bianque_v1_v2_app.py comme exemple d'utilisation conjointe du modèle BianQue-1.0 et du modèle BianQue-2.0.
Sur la base du big data BianQue Health BianQueCorpus, nous avons choisi ChatGLM-6B comme modèle d'initialisation et avons obtenu la nouvelle génération de BianQue [BianQue-2.0] après un entraînement de réglage fin de tous les paramètres. Différent du modèle Bianque-1.0, Bianque-2.0 a étendu les données telles que les instructions sur les médicaments, les instructions de connaissances de l'encyclopédie médicale et les instructions de distillation ChatGPT, renforçant ainsi les suggestions et les capacités de requête de connaissances du modèle. Vous trouverez ci-dessous deux échantillons de test.
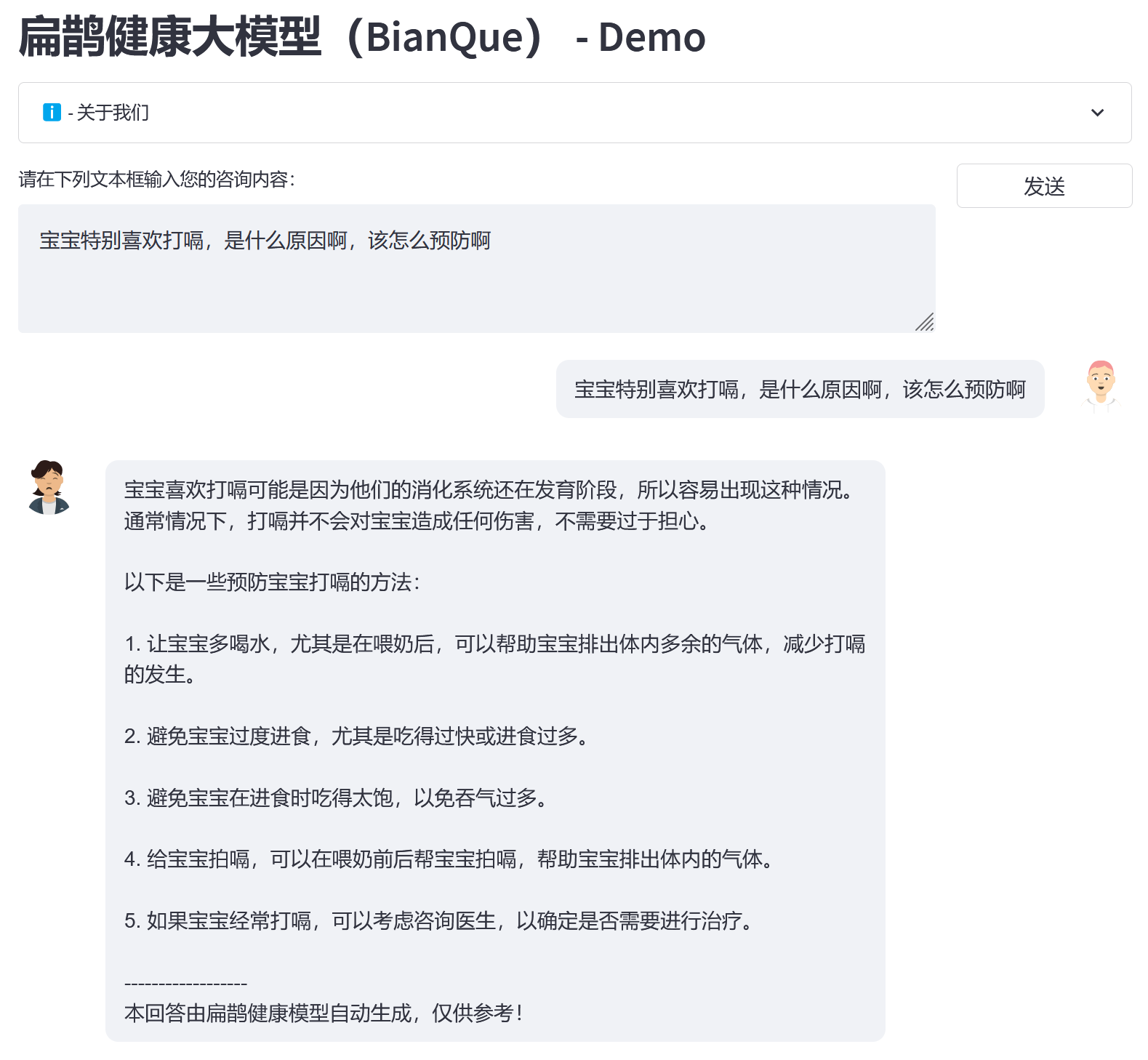
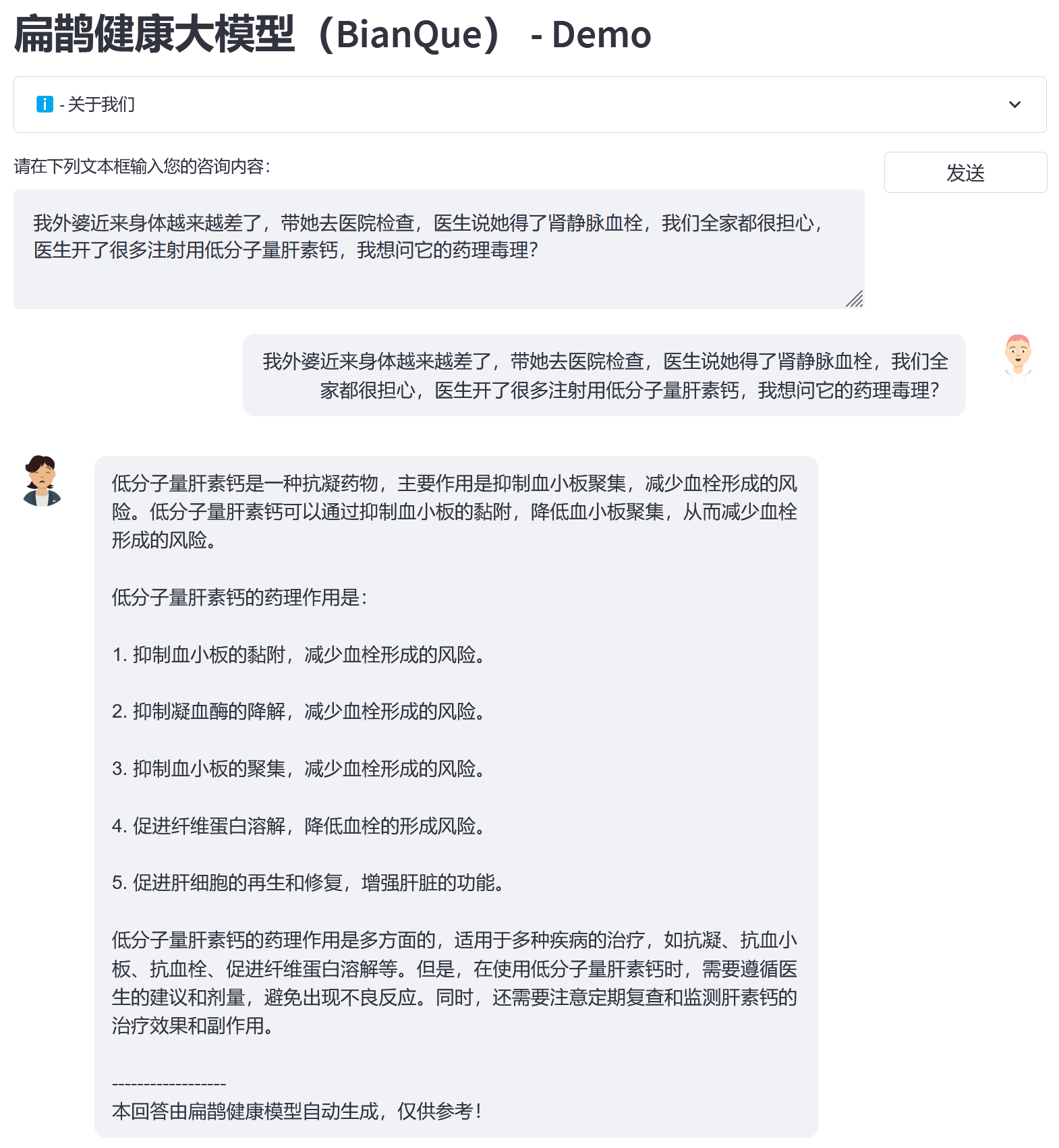
Utilisez la commande suivante pour utiliser conjointement Bianque-2.0 et Bianque-1.0 pour créer des services de santé actifs :
streamlit run bianque_v1_v2_app.py --server.port 9005Voici un exemple d'application : les premiers tours sont le processus d'enquête via le modèle Bianque-1.0, et le dernier cycle de réponses est la réponse via le modèle Bianque-2.0.
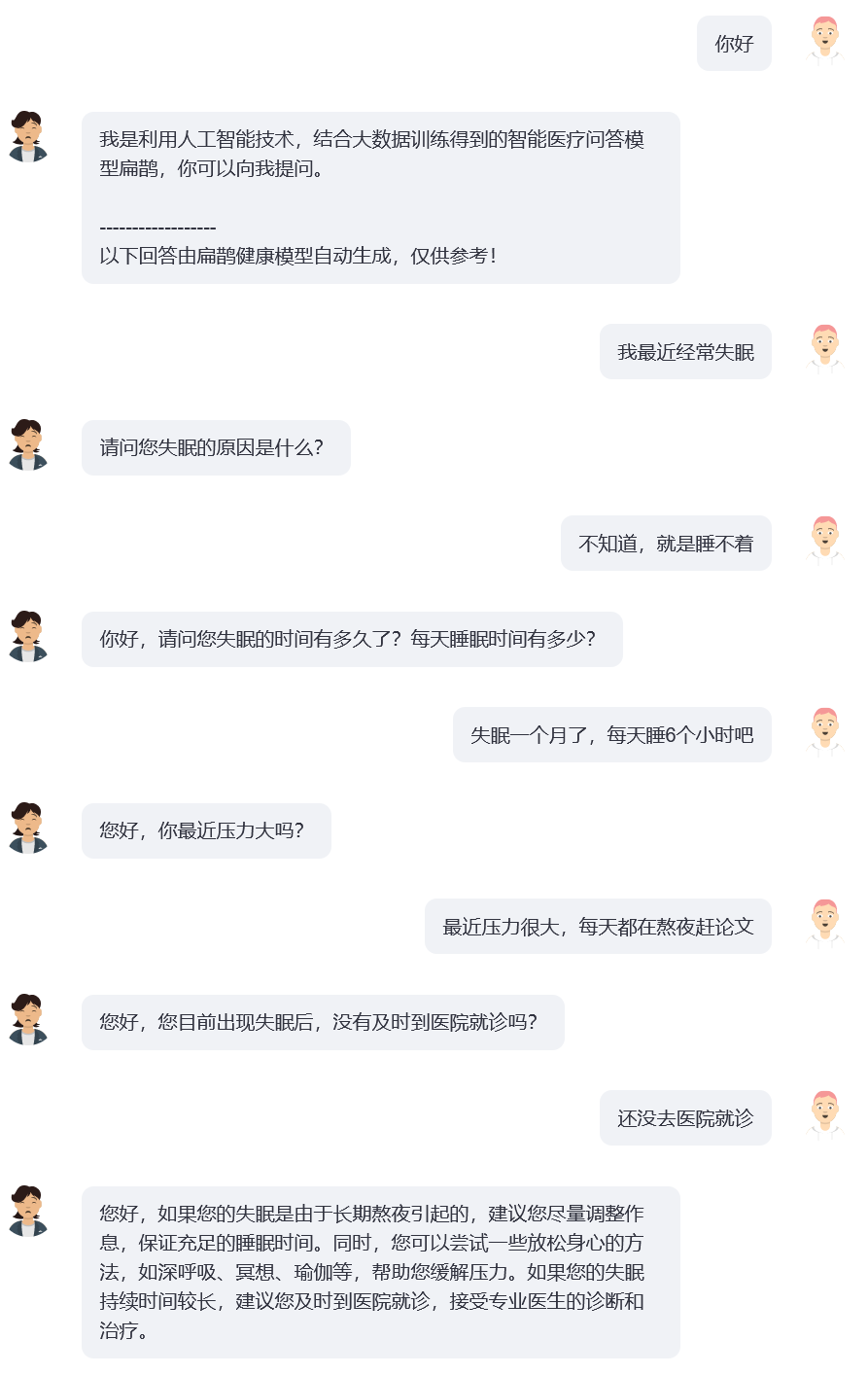
BianQue-1.0 (BianQue-1.0) est un vaste modèle de dialogue médical affiné grâce à des instructions et à plusieurs séries de dialogues d'enquête. Après des recherches, nous avons constaté que dans le domaine médical, les médecins doivent souvent passer par plusieurs séries d'enquêtes avant de prendre une décision. Il ne s'agit pas d'un simple modèle « instruction-réponse ». Lorsque les utilisateurs consultent un médecin, ils ne l'informent souvent pas de la situation complète au début, le médecin doit donc continuer à poser des questions avant de pouvoir finalement poser un diagnostic et donner des suggestions raisonnables. Sur cette base, nous avons construit BianQue-1.0 (BianQue-1.0) , dans le but de renforcer les capacités d'interrogation du système d'IA pour simuler le processus d'interrogation du médecin. Nous définissons cette capacité comme la partie « demander » de « regarder, entendre, demander et comprendre ». En tenant compte de l'architecture actuelle du modèle chinois, du nombre de paramètres et de la puissance de calcul requise, nous avons utilisé ClueAI/ChatYuan-large-v2 comme modèle de base et affiné 1 époque sur 8 cartes graphiques NVIDIA RTX 4090 pour obtenir Bianque- 1.0 (BianQue-1.0) , l'ensemble de données mixtes d'instructions de questions et réponses médicales chinoises et de dialogues d'enquête à plusieurs tours utilisé pour la formation contient plus de 9 millions d'échantillons, ce qui a pris environ 16 jours pour terminer une période de formation. Nous prévoyons de mener plusieurs versions de recherche itérative sur le modèle basées sur les capacités « voir, entendre, demander et ressentir » du modèle Bian Que, combinées aux connaissances d'experts médicaux, à la technologie multimodale et aux calculs de signaux multiphysiologiques. Le modèle BianQue accueille vos contributions ! Nous vous encourageons à signaler les problèmes, à contribuer aux relations publiques et à participer aux discussions sur la page BianQue GitHub. Nous sommes impatients de coopérer avec davantage d'universités, d'hôpitaux, de laboratoires de recherche, d'entreprises, etc. pour mener conjointement des recherches sur le modèle Bianque de nouvelle génération. Pour de telles demandes (et autres demandes ne pouvant pas être effectuées sur GitHub), veuillez envoyer un e-mail directement à [email protected].
Les quatre méthodes de diagnostic « regarder, sentir, demander et ressentir » ont été inventées par Bian Que. Les « Quatre méthodes de diagnostic » (inspection, odorat, interrogatoire et palpation) sont les méthodes de diagnostic de base en médecine traditionnelle chinoise. Parmi eux, la capacité de « demander » peut être modélisée dans le modèle linguistique. Nous définissons la capacité de « demander » du modèle d'IA comme :
Capacité « Demander » : Le modèle d'IA peut mener des enquêtes appropriées sur la base des informations saisies par le patient et combinées avec des connaissances médicales, et donner un "diagnostic ou des conseils appropriés" sur la base des informations du patient obtenues lors d'enquêtes ultérieures. Le processus de « demande » est généralement un processus de dialogue à plusieurs tours.
Voici 2 exemples simples :
Exemple 1
Utilisateur : Je me sens complètement épuisé ces derniers temps. Bian Que-1.0 : Est-ce à cause de la fatigue ou du fait de veiller tard ?
Utilisateur : Je me suis couché tard cette semaine pour rattraper mon retard. Bian Que-1.0 : Eh bien, rester éveillé tard peut facilement provoquer divers inconforts, tels que des étourdissements, de la fatigue, de l'insomnie, de la rêverie, etc. Vous pouvez prendre de l'oryzanol, de la vitamine B1 et du liquide Anshen Bu Nao pour éviter de rester éveillé.
Exemple 2
Utilisateur : Je souffre beaucoup d'insomnie ces derniers temps. Bianque-1.0 : Depuis combien de temps souffrez-vous d'insomnie ?
Utilisateur : Cela fait deux semaines Bianque-1.0 : Comment se passe ton sommeil ?
Utilisateur : Combien d'heures devez-vous vous coucher avant de pouvoir dormir ? Bian Que-1.0 : Vous pouvez prendre certains médicaments par voie orale. Utilisateur : Avez-vous des recommandations spécifiques ? Bian Que-1.0 : Je dois d'abord vérifier. : De quels tests ai-je besoin ?
Bian Que-1.0 : Électrocardiogramme
import os
from transformers import T5Tokenizer , T5ForConditionalGeneration , AutoTokenizer
import torch
device = torch . device ( "cuda" if torch . cuda . is_available () else "cpu" )
tokenizer = T5Tokenizer . from_pretrained ( "scutcyr/BianQue-1.0" )
model = T5ForConditionalGeneration . from_pretrained ( "scutcyr/BianQue-1.0" )
model = model . to ( device )
def preprocess ( text ):
text = text . replace ( " n " , " \ n" ). replace ( " t " , " \ t" )
return text
def postprocess ( text ):
return text . replace ( " \ n" , " n " ). replace ( " \ t" , " t " )
def answer ( user_history , bot_history , sample = True , top_p = 1 , temperature = 0.7 ):
'''sample:是否抽样。生成任务,可以设置为True;
top_p:0-1之间,生成的内容越多样
max_new_tokens=512 lost...'''
if len ( bot_history ) > 0 :
context = " n " . join ([ f"病人: { user_history [ i ] } n医生: { bot_history [ i ] } " for i in range ( len ( bot_history ))])
input_text = context + " n病人:" + user_history [ - 1 ] + " n医生:"
else :
input_text = "病人:" + user_history [ - 1 ] + " n医生:"
return "我是利用人工智能技术,结合大数据训练得到的智能医疗问答模型扁鹊,你可以向我提问。"
input_text = preprocess ( input_text )
print ( input_text )
encoding = tokenizer ( text = input_text , truncation = True , padding = True , max_length = 768 , return_tensors = "pt" ). to ( device )
if not sample :
out = model . generate ( ** encoding , return_dict_in_generate = True , output_scores = False , max_new_tokens = 512 , num_beams = 1 , length_penalty = 0.6 )
else :
out = model . generate ( ** encoding , return_dict_in_generate = True , output_scores = False , max_new_tokens = 512 , do_sample = True , top_p = top_p , temperature = temperature , no_repeat_ngram_size = 3 )
out_text = tokenizer . batch_decode ( out [ "sequences" ], skip_special_tokens = True )
print ( '医生: ' + postprocess ( out_text [ 0 ]))
return postprocess ( out_text [ 0 ])
answer_text = answer ( user_history = [ "你好!" ,
"我最近经常失眠" ,
"两周了" ,
"上床几小时才睡得着" ],
bot_history = [ "我是利用人工智能技术,结合大数据训练得到的智能医疗问答模型扁鹊,你可以向我提问。" ,
"失眠多久了?" ,
"睡眠怎么样?" ])conda env create -n bianque_py38 --file py38_conda_env.yml
conda activate bianque_py38
pip install torch==1.13.1+cu116 torchvision==0.14.1+cu116 torchaudio==0.13.1 --extra-index-url https://download.pytorch.org/whl/cu116 cd scripts
bash run_train_model_bianque.shBianQue-1.0 (BianQue-1.0) n'a été formé que pendant 1 époque. Bien que le modèle ait certaines capacités d'enquête médicale, il présente toujours les limitations suivantes :
**BianQue-2.0(BianQue-2.0)** utilise les poids du modèle ChatGLM-6B et doit suivre son MODEL_LICENSE Par conséquent, ce projet ne peut être utilisé qu'à des fins de recherche non commerciales .
Ce projet a été lancé par le Laboratoire clé des jumeaux numériques de la province du Guangdong, École des technologies du futur, Université de technologie de Chine du Sud. Il est soutenu par le Centre de recherche en ingénierie des réseaux d'information, École d'électronique et d'information et d'autres départements de l'Université de technologie de Chine du Sud. Il remercie également l'hôpital de santé maternelle et infantile du Guangdong, le centre médical pour femmes et enfants du gouvernement municipal de Guangzhou, le troisième hôpital affilié à l'université Sun Yat-sen, l'institut de recherche sur l'intelligence artificielle du centre national des sciences de Hefei et d'autres unités coopératives.
Nous tenons également à remercier les médias ou comptes publics suivants pour avoir rendu compte de ce projet (sans ordre particulier) :
Couverture médiatique Quotidien du Peuple, China.com, Guangming.com, TOM Technology, Future.com, Dazhong.com, China Development Report Network, China Daily Network, Xinhua News Network, China.com, Toutiao, Sohu, Tencent News, NetEase News , Réseau d'information chinois, Réseau de communication chinois, Réseau de rapports sur les villes de Chine, Réseau des villes de Chine
Construction d'un laboratoire de compte public dans le Guangdong, voix intelligente pour la nouvelle jeunesse, apprentissage profond et PNL, AINLP
@misc { chen2023bianque ,
title = { BianQue: Balancing the Questioning and Suggestion Ability of Health LLMs with Multi-turn Health Conversations Polished by ChatGPT } ,
author = { Yirong Chen and Zhenyu Wang and Xiaofen Xing and huimin zheng and Zhipei Xu and Kai Fang and Junhong Wang and Sihang Li and Jieling Wu and Qi Liu and Xiangmin Xu } ,
year = { 2023 } ,
eprint = { 2310.15896 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CL }
}