spfy : plate-forme de prédiction des sous-types à partir des séquences du génome entier d'E.coli et qui crée des données graphiques pour des analyses comparatives à l'échelle de la population.
Publié sous le nom : Le,KK, Whiteside,MD, Hopkins,JE, Gannon,VPJ, Laing,CR spfy : une base de données graphique intégrée pour la prédiction en temps réel des phénotypes bactériens et des analyses comparatives en aval. Base de données (2018) Vol. 2018 : numéro d'article bay086 ; est ce que je:10.1093/base de données/bay086
En direct : https://lfz.corefacility.ca/superphy/spfy/
git clone --recursive https://github.com/superphy/spfy.gitcd spfy /docker-compose upType EC :
PanPredic :
Image Docker pour Conda :
Comparaison de différents groupes de population :
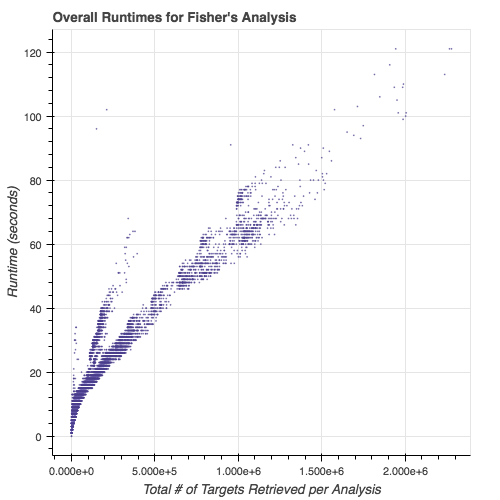
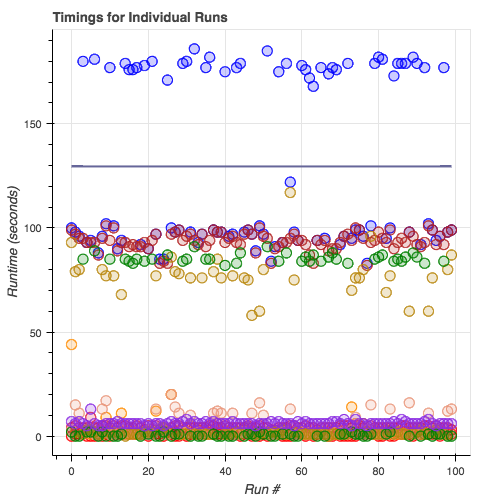
Runtimes des modules de sous-typage :
cd app/python -m modules/savvy -i tests/ecoli/GCA_001894495.1_ASM189449v1_genomic.fna où l'argument après le -i est votre fichier génome (FASTA). 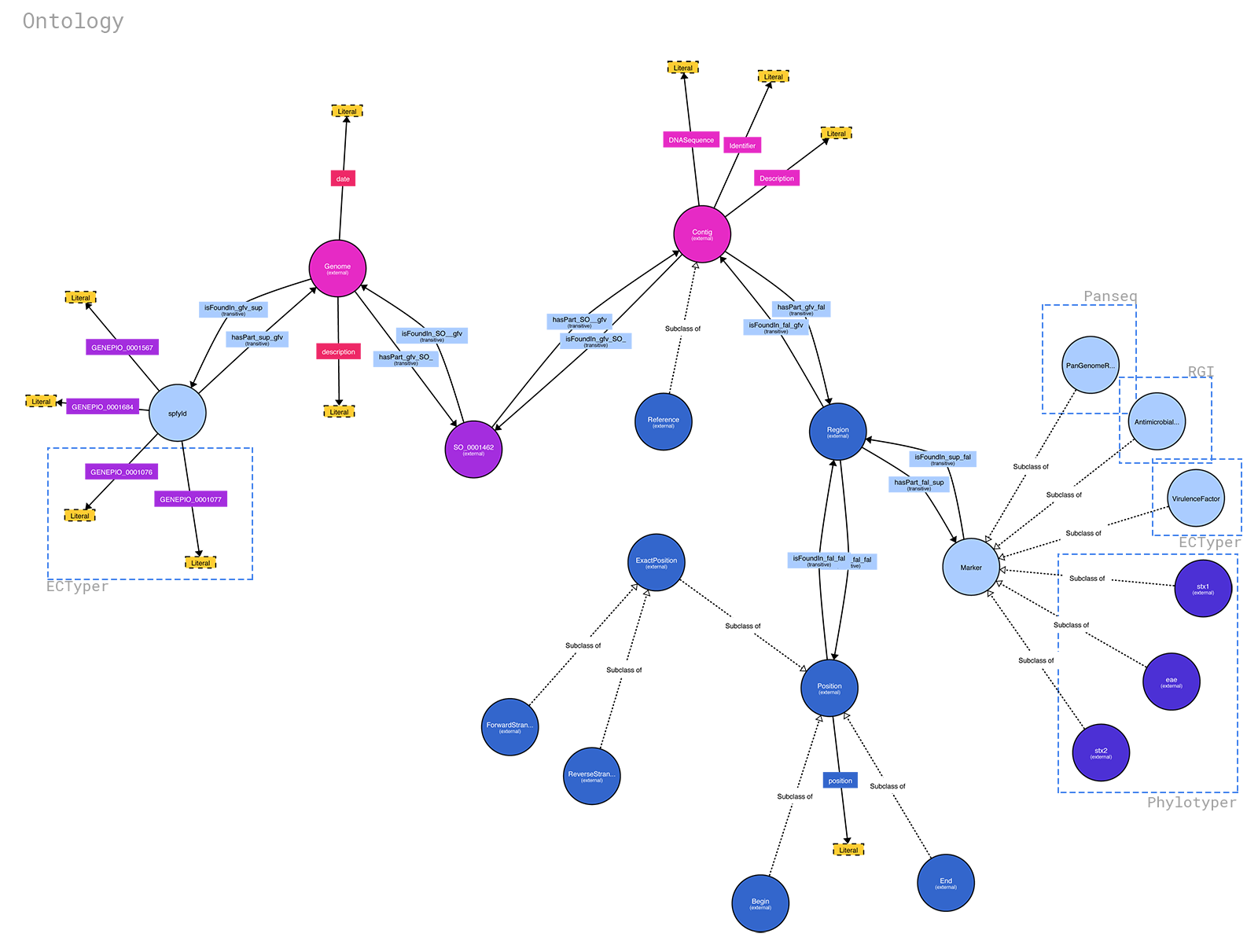
L'ontologie pour spfy est disponible sur : https://raw.githubusercontent.com/superphy/backend/master/app/scripts/spfy_ontology.ttl. Elle a été générée à l'aide de https://raw.githubusercontent.com/superphy/backend/master /app/scripts/generate_ontology.py avec les fonctions partagées du code backend de spfy . Si vous souhaitez l'exécuter, faites : 1. cd app/ 2. python -m scripts/generate_ontology qui mettra l'ontologie dans app/
Vous pouvez générer un joli diagramme à partir du fichier .ttl en utilisant http://www.visualdataweb.de/webvowl/
Note
actuellement configuré uniquement pour les fichiers .fna
Vous pouvez contourner le site Web frontal tout en mettant les tâches de sous-typage en file d'attente en :
/datastore dans les conteneurs.Par exemple, si vous conservez vos fichiers dans
/home/bob/ecoli-genomes/, vous modifierez le fichierdocker-compose.ymlet remplacerez :volumes : - /datastoreavec:
volumes : - /home/bob/ecoli-genomes:/datastore
docker-compose down docker-compose up -d
docker exec -it backend_webserver_1 sh python -m scripts/sideload exit
Notez que des résidus peuvent être créés dans votre dossier génome.

| Docker Image | Ports | Noms | Description |
|---|---|---|---|
| back-end-rq | 80/tcp, 443/tcp | back end_wor ker_1 | les principaux travailleurs de la file d'attente Redi |
| back-end - rq-b laze grap h | 80/tcp, 443/tcp | back end_wor ker-blaz egra ph-i ds_ 1 | cela donne la ratio des gènes spfy ID pour la base de données blaz egra ph |
| back-end | 0,0. 0.0 : 8000 ->80 /tcp, 443/tcp | back end_web -ngi nx-u wsgi _1 | le back-end du flacon qui gère les tâches d'enquête |
| super rphy /bla zegr aph : 2.1. Référence 4 pouces | 0,0. 0,0 : 8080 ->80 80/t cp | retour end_bla zegr aph_1 | Blaz egra ph Base de données |
| redi s:3. 2 | 6379 /tcp | back end_red is_ 1 | Base de données Redi |
| réagir | 0,0. 0,0 : 8090 ->50 00/t cp | retour end_rea ctap p_1 | front t-en d pour spfy |
L'image superphy/backend-rq:2.0.0 est évolutive : vous pouvez créer autant d'instances que vous en avez besoin/disposez de puissance de traitement. L'image est chargée d'écouter la file d'attente multiples (12 travailleurs) qui gère la plupart des tâches, y compris les appels RGI . Il écoute également la file d'attente singles (1 travailleur) qui exécute ECTyper . Ceci est fait car RGI est la partie la plus lente de l’équation. La gestion des travailleurs est gérée par supervisor .
L'image superphy/backend-rq-blazegraph:2.0.0 n'est pas évolutive : elle est chargée d'interroger la base de données Blazegraph pour les entrées en double et d'attribuer des ID spfy dans un ordre séquentiel . Ses fonctions sont aussi minimes que possible pour améliorer les performances (car la génération d'ID est le seul goulot d'étranglement dans les pipelines autrement parallèles) ; les comparaisons sont effectuées par les hachages sha1 des fichiers soumis et les non-doublons ont leurs identifiants réservés en liant l'ID spfy généré au hachage du fichier. La gestion des travailleurs est gérée par supervisor .
Le superphy/backend:2.0.0 qui exécute les points de terminaison Flask utilise supervisor pour gérer les processus internes : nginx , uWsgi .
database['blazegraph_url'] dans /app/config.py Les étapes requises pour ajouter de nouveaux modules sont documentées dans le Guide du développeur.