Référentiel contenant des données et du code pour le génotypage du guar ( Cyamopsis tetragonoloba (L.) Taub.) et le projet GWAS. Une brève description du contenu du référentiel :
Le script make_consensus_vcf.py a été utilisé pour générer l'ensemble de données de variantes final à l'aide d'appels de variantes bruts GATK-HC, NGSEP et TASSEL 5, le script make_snp_stats.py a été utilisé avec les mêmes entrées pour explorer les statistiques de qualité des appels de variantes.
Le notebook callset_refinement_filtering.ipynb a été utilisé pour exécuter le contrôle qualité des variantes et construire l'ensemble de données filtré final des variantes communes.
Le carnet genetic_analysis.ipynb a été utilisé pour effectuer la plupart des analyses génétiques, y compris l'ACP, l'analyse de déséquilibre de liaison et l'analyse d'association basée sur un modèle linéaire généralisé.
Le script prepare_farmcpu.py a été utilisé pour formater les données d'entrée pour l'outil FarmCPU (le tracé QQ des valeurs p résultantes est présenté dans l'article et ci-dessous).
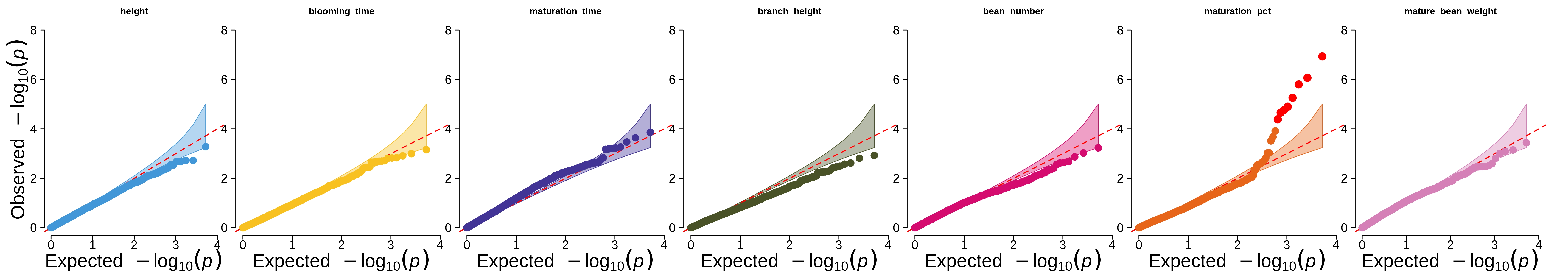
Le fichier admix_file_create.py a été utilisé pour prétraiter les génotypes pour l'analyse ADMIXTURE (une partie de cela a été réalisée en utilisant Hail dans genetic_analysis.ipynb ).
Le script guar_stat.R a été utilisé pour exécuter une analyse statistique des données finales, effectuer une analyse d'association à l'échelle du génome à l'aide de l'outil FarmCPU et tracer les chiffres principaux.
Le script parse_vcf_fcpu.py a été utilisé pour formater la table finale des génotypes SNP disponible sous forme de fichier genotypes.xlsx dans ce référentiel.